Геномное окружение. База данных GO
Получение информации о КОГе белка
В первом семестре мне был выдан белок YP_694641.1. К настоящему моменту он переименован
и имеет идентификатор
WP_003452659.1. Белок является N-ацетилманозамин-6-фосфат 2-эпимеразой из бактерии
Clostridium perfringens. Всего в нем 221 аминокислотный остаток.
С помощью сервиса CDD
(Conserved Domain Database) для данного белка был получен список хитов (Рис. 1),
из которого были выбраны те, которые относят белок к тому или иному КОГу.
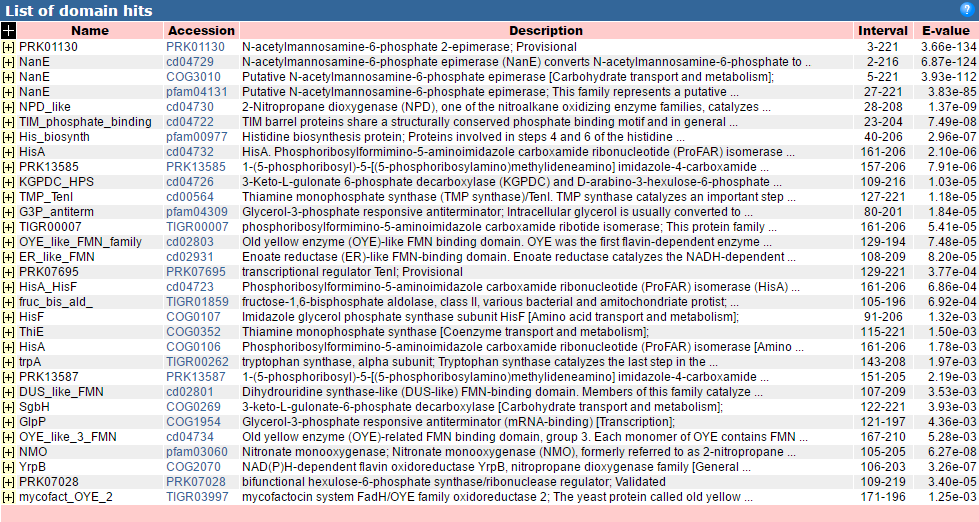 |
| Рис. 1. Список хитов для белка WP_003452659.1 |
Таких хитов, соответственно и КОГов, оказалось несколько (7). Информация по каждому из КОГов была получена из последнего релиза базы данных и представлена в Таблице 1.
| ID | E-value | Координаты | Название | Функциональная категория |
| COG3010 | 3.93e-112 | 5-221 | Putative N-acetylmannosamine-6-phosphate epimerase (Предполагаемая N-ацетилманозамин-6-фосфат 2-эпимераза) |
G - Carbohydrate transport and metabolism (Транспорт и метаболизм углеводов) |
| COG0107 | 1.32e-03 | 91-206 | Imidazole glycerol phosphate synthase subunit HisF (Субъединица имидазол-глицерол-фосфат-синтазы HisF) |
E - Amino acid transport and metabolism (Транспорт и метаболизм аминокислот) |
| COG0352 | 1.50e-03 | 115-221 | Thiamine monophosphate synthase (Тиамин-монофосфат-синтаза) |
H - Coenzyme transport and metabolism (Транспорт и метаболизм коферментов) |
| COG0106 | 1.78e-03 | 161-206 | Phosphoribosylformimino-5-aminoimidazole carboxamide ribonucleotide (ProFAR) isomerase (Фосфорибозилформино-5-аминоимидазол-карбоксамид-рибонуклеотид-изомераза) |
E - Amino acid transport and metabolism (Транспорт и метаболизм аминокислот) |
| COG0269 | 3.93e-03 | 122-221 | 3-keto-L-gulonate-6-phosphate decarboxylase (3-кето-L-гулонат-6-фосфат-декарбоксилаза) |
G - Carbohydrate transport and metabolism (Транспорт и метаболизм углеводов) |
| COG1954 | 4.36e-03 | 121-197 | Glycerol-3-phosphate responsive antiterminator (mRNA-binding) (Глицерол-3-фосфат-чувствительный антитерминатор (мРНК-связывающий)) |
K - Transcription (Транскрипция) |
| COG2070 | 3.26e-07 | 106-203 | NAD(P)H-dependent flavin oxidoreductase YrpB, nitropropane dioxygenase family (НАД(Ф)H-зависимая флавин-оксидоредуктаза YrpB, семейство нитропропановых диоксигеназ) |
R - General function prediction only (Предсказание только общей функции) |
| Таблица 1. КОГи для белка WP_003452659.1 | ||||
Визуализация геномного окружения
Далее было необходимо получить изображение геномного окружения КОГа c помощью сервисов
STRING и COGNAT.
Из множества обнаруженных КОГов я выбрала один с наилучшим E-value, а именно
COG3010.
Для начала было получено изображение с помощью STRING (Рис. 2.). Поиск проводился по последовательности
нашего белка в пределах организма Clostridium perfringens.
Параметры программы были взяты по умолчанию:
максимальное количество взаимодействующих по первой линии белков (first shell interactors) - 10,
минимальный требуемый score взаимодействия - 0.400.
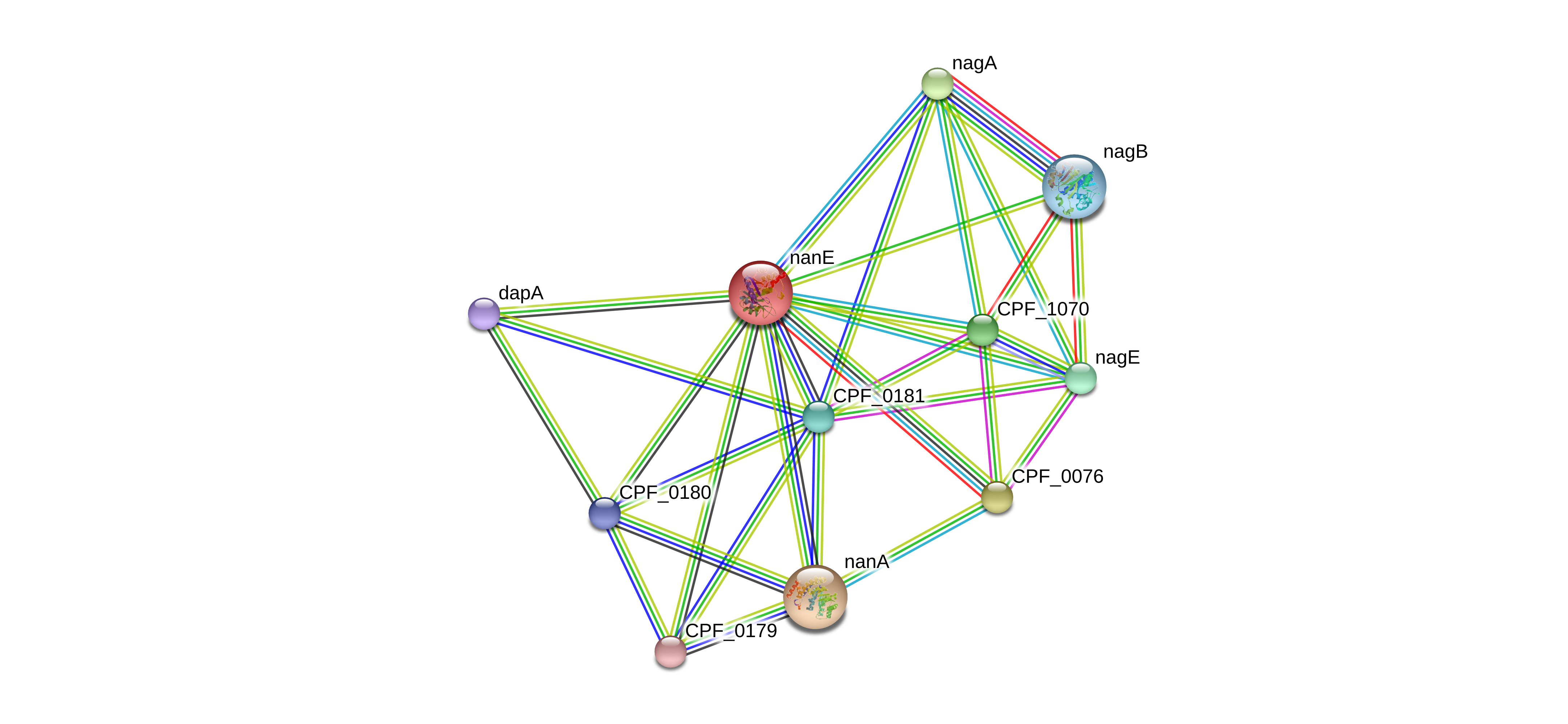 |
| Рис. 2. Геномное окружение COG3010, программа STRING |
На данном рисунке каждый узел графа символизирует белок (а точнее - совокупность всех его изоформ, транскрибирующихся с одного и того же локуса гена). Ребра графа символизируют белок-белковые взаимосвязи (это не обязательно означает, что белки физически связывают друг друга; они могут быть связаны и функционально, например совместно выполнять какую-либо функцию). Белки, для которых известна 3D-структура отображены большимим узлами с соответсвующим изображением внутри, остальные белки отображены узлами меньшего размера.
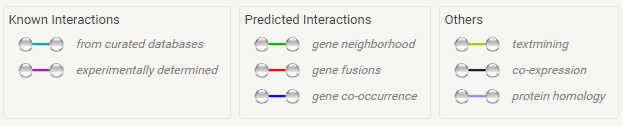 |
| Рис. 3. Значения цветов ребер |
На нашем графе наибольшее число ребер окрашены в зеленый, салатовый или синий (взаимосвязи, которым нельзя верить на 100%). Однако есть и достаточное количество (5) достоверных взаимосвязей, установленных экспериментально.
На рисунке 4 представлена таблица, отражающая все взаимосвязи нашего белка с остальными белками на графе, а также score данных взаимосвязей.
 |
| Рис. 4. Взаимосвязи белка WP_003452659.1 |
В выданной группе белков наблюдается консервативное геномное окружение. Каждый из белков связан с любым из остальных зеленым ребром, отражающим генетическое соседство (gene neighborhood). Наблюдаются очевидные повторяющиеся паттерны расположения генов в разных группах организмов (Рис. 5.).
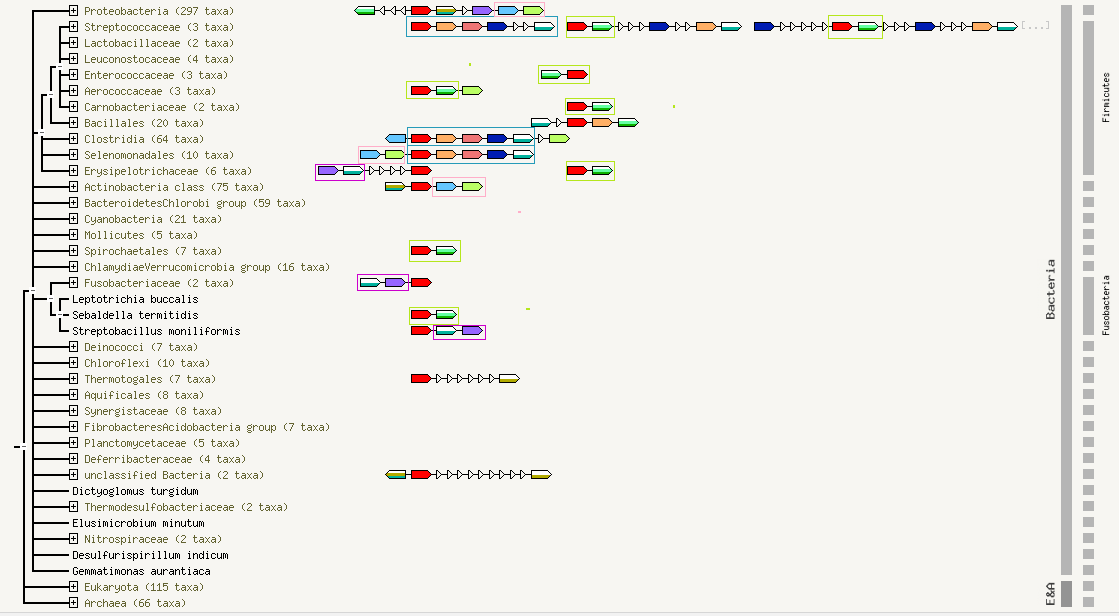 |
| Рис. 4. Взаимосвязи белка WP_003452659.1 |
| ID | Описание |
| COG3010 | Переводит N-ацетилманозамин-6-фосфат в N-ацетилглюкозамин-6-фосфат |
| COG0329 | Катализирует конденсацию (S)-аспартат-бета- семиальдегид (S)-ASA и пирувата в 4-гидроксил- тетрагидродипиколинат (N-ацетилнейрамиат лиаза) |
| COG0591 | симпортер |
| COG2731 | белок семейства YhcH YjgK YiaL |
| COG1940 | белок семейства ROK |
| Таблица 2. Описания COGов, входящих в консервативный паттерн | |
Описания COGов паттерна представлены в таблице 2.
Как видно из описаний, как минимум первые два COGа связаны функционально.
Кроме описанного паттерна встречаются и более короткие (выделены одинаковыми цветами на рисунке 4), например, паттерн включающий уже описанный COG3010(красный) и COG1263 (бело-зеленый), выполняющий функцию фосфотрансферазной системы (салатовая рамка), и другие.
В пределах паттернов наблюдается некоторая вариабельность. Так, для уже описанного самого длинного паттерна в кладе Streptococcaceae наблюдается дополнительная вставка двух участков, включающих COG4409 (функция - нейраминидаза) и COG0673 (функция - оксидоредуктаза).
В паттернах, выделенных фиолетовыми и салатовыми рамками, наблюдаются перемены COGов местами.
Отнесение белка N-ацетилманозамин-6-фосфат 2-эпимеразs из бактерии Clostridium perfringens к терминам GO
С помощью инструмента AmiGO поиком BLAST в базе данных GO был обнаружен белок, наиболее похожий на WP_003452659.1.
(UniProtKB - Q8XNZ3 (NANE_CLOPE)).
Им оказался белок Q71VW2 (NANE_LISMF) - предполагаемая N-ацетилманозамин-6-фосфат 2-эпимераза
из организма Listeria monocytogenes serotype 4b. Изначально выданный мне белок выполняет ту
же функцию, но принадлежит другому организму - Clostridium perfringens. Тем не менее эти бактерии достаточно
близки и обе относятся к кладе Firmicutes.
 |
| Рис. 5. Выравнивание белков NANE_CLOPE и NANE_LISMF |
Очевидно, что найденный в БД GO белок не является тем же самым, что и исходный.
Тем не менее степень сходства у этих белков хоть и не высокая, но достаточная, о чем можно судить по неплохому E-value и удовлетворительному проценту совпадений.
На странице белка Q71VW2 (NANE_LISMF) по ссылке N term associations была получена информация о терминах GO, отнесенных к данному белку. Их оказалось всего 2. Найденные термины представлены в таблице 3.
| Аспект | Идентификатор GO | Название термина | Перевод названия | Код типа достоверности |
| Биологический процесс (Biological process) | GO:0006040 | Amino sugar metabolic process | Метаболизм аминосахаров | ISS |
| Молекулярная функция (Molecular function) | GO:0016857 | Racemase and epimerase activity, acting on carbohydrates and derivatives | Рацемазная активность по отношению к углеводам и их производным | ISS |
| Таблица 3. Термины GO, отнесенные к белку с идентификатором Uniprot Q71VW2 (NANE_LISMF) | ||||
Объяснения встречающихся в таблице 3 кодов достоверности представленs в таблице 4.
| Код типа достоверности | Расшифровка кода | Объяснение |
| ISS | Inferred from Sequence or Structural Similarity (основан на сходстве последовательностей или структурном сходстве) | Используется, когда аннотация проводилась на основании анализа последовательностей, причем
данный анализ был проверен вручную. В случае, когда имел место только автоматический анализ,
корректным является использование кода IEA. Общий код ISS используется при применение комбинации анализирующих последовательности инструментов и методов. Если был применен только один метод/инструмент используют одну из подкатегорий ISS: ISA (Inferred from Sequence Alignment) - заключение на основании парного или множественного выравнивания, ISO (Inferred from Sequence Orthology) - заключение на основании оценки ортологичности продуктов генов из разных организмов, ISM (Inferred from Sequence Model) - заключение на основании какого-либо метода моделирования (например Hidden Markov Models). ISS также можно использовать при наличии структурного сходства с экспериментально описанными продуктами генов, установленного с помощью кристаллографии, ЯМР или вычислительных предсказаний. Однако на практике код ISS крайне редко применяют для аннотации, основанной только на информации о структуре. |
| Таблица 4. Описание кодов достоверности | ||