Поиск по PDB и содержание PDB-файлов
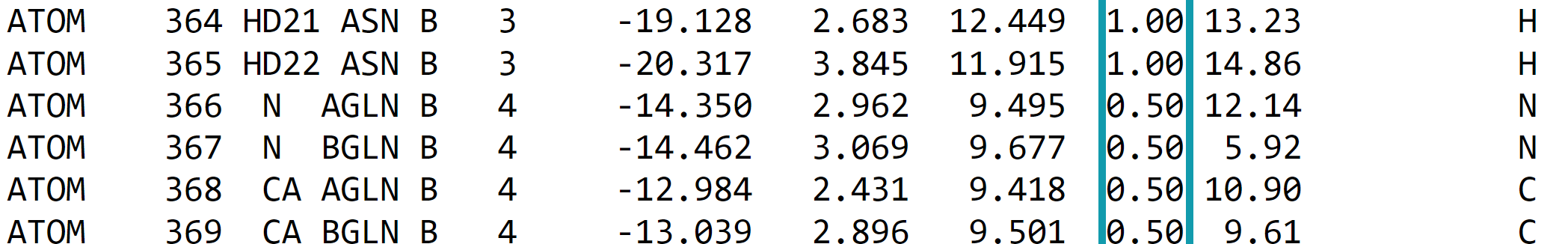 |
| Рисунок 1. Строки PDB-файла структуры 1mso, в которых occupancy не равен 1 |
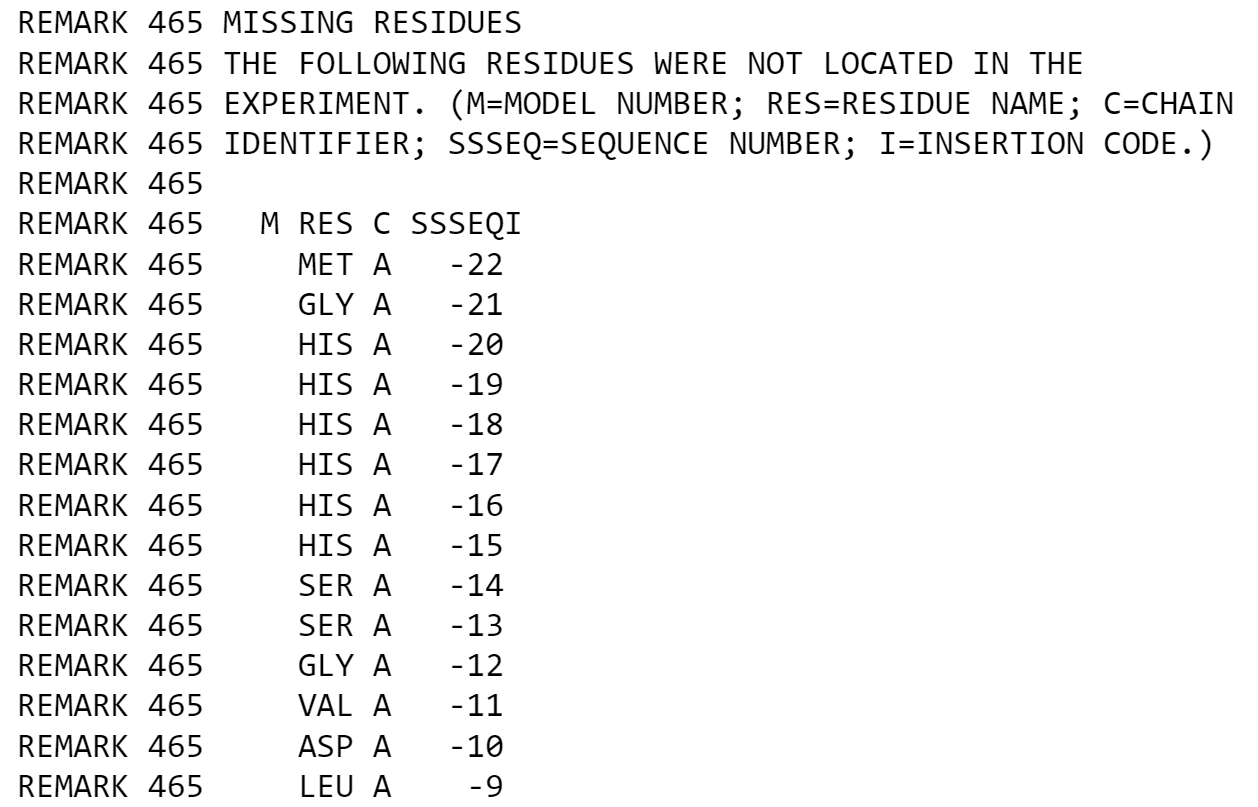 |
| Рисунок 3. Поле Missing residues в PDB-файле структуры 1csk |
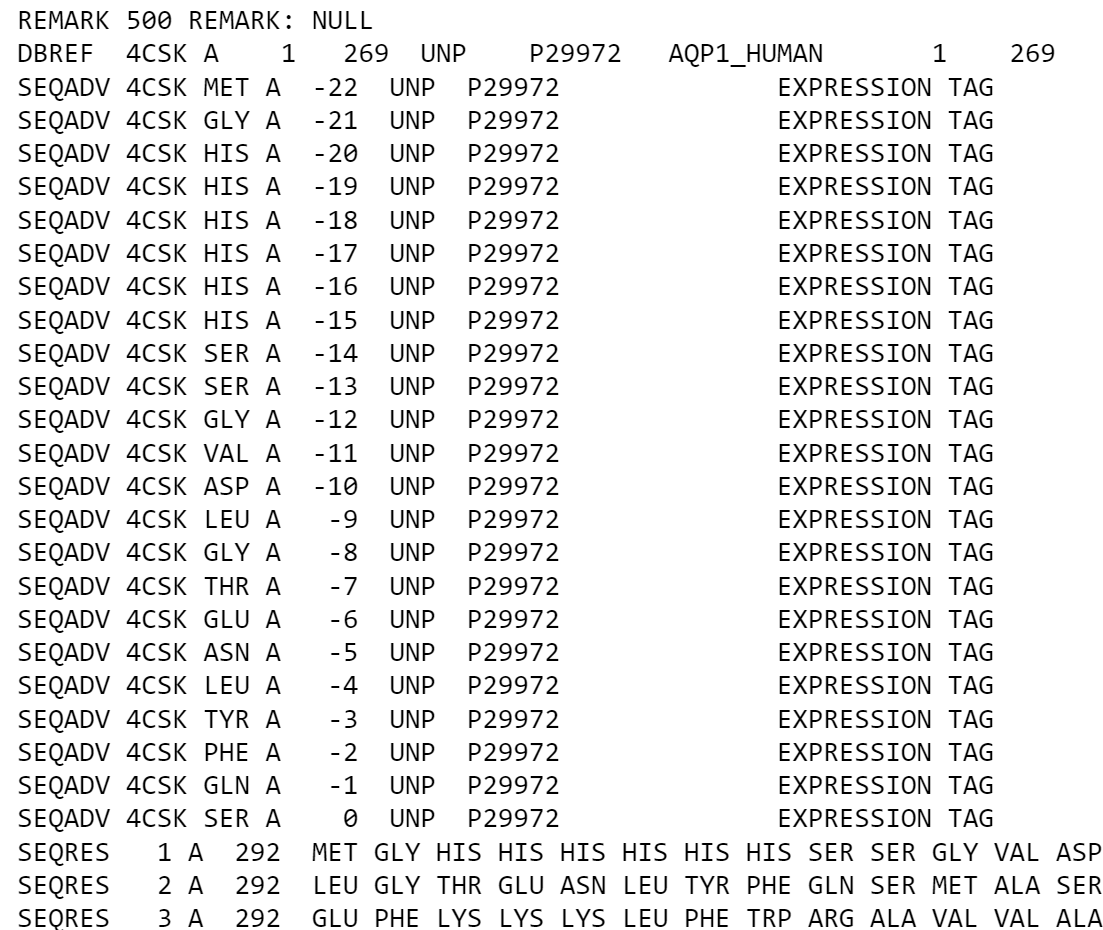 |
| Рисунок 4. Поля SEQRES м SEQADV в PDB-файле структуры 1csk |
 |
| Рисунок 5. Лучший и худший B-факторы в структуре 1k88 |
Коэффициент заполнения атома - доля элементарных ячеек, в которых присутствует данный атом в указанной позиции. В норме он равен 1, но может быть < 1 при наличии альтернативных позиций.
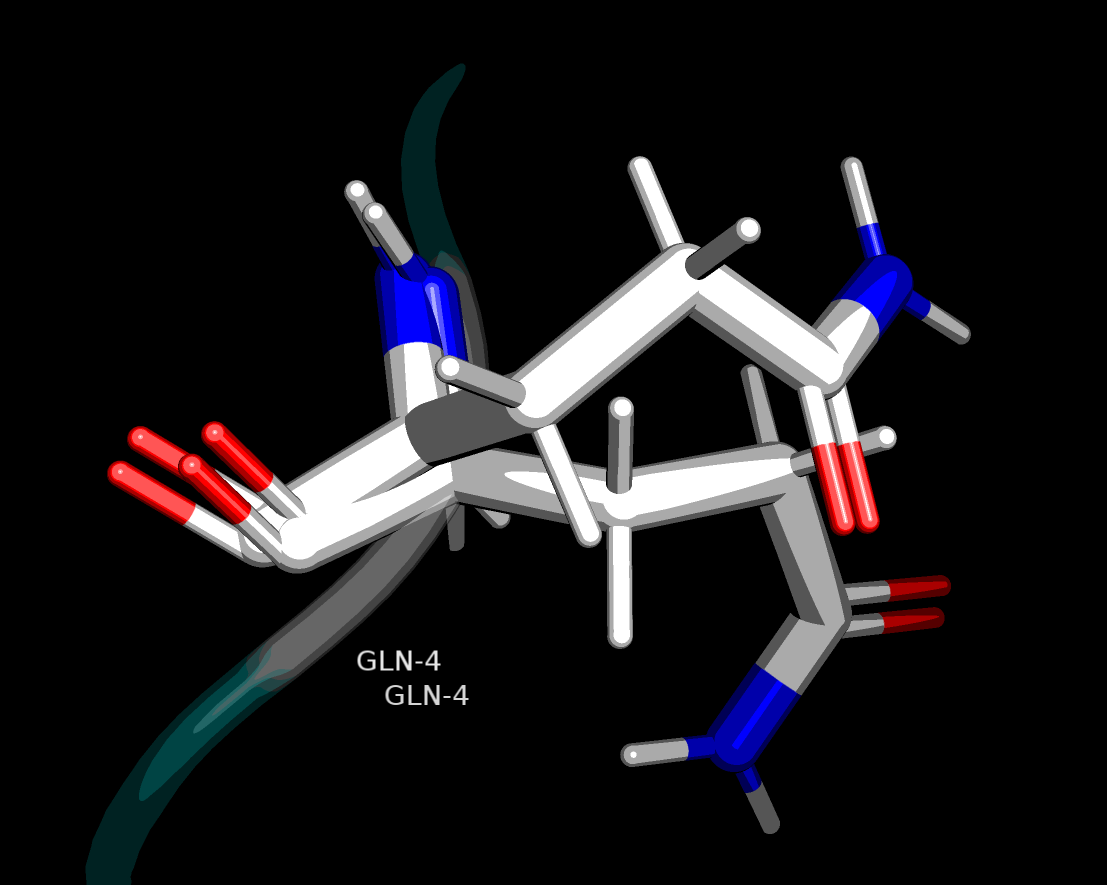 |
| Рисунок 2. Gln4 в структуре 1mso, принимающий альтернативные конформации |
Чтобы найти подходящую структуру, в PDBe был проведен расширенный поиск с параметрами "Experimental method: x-ray diffraction" и "Resolution ≤ 1". Подходящей оказалась структура человеческого инсулина (PDB ID: 1mso) c разрешением 1 Å.
На рисунке 1 показаны строчки PDB-файла с выделенной колонкой Occupancy. Видно, что в остатке глутамина атом остовный атом азота (366-367) и атом CA (368-369) принимают два альтернативных положения, каждое из которых равновероятно представлено среди всех элементарных ячеек (occupancy = 0.5).
На рисунке 2 представлено изображение данного остатка глутамина с альтернативными конформациями. Вполне ожидаемо он находится в неструктурированной петле, экспонированной на поверхность белковой глобулы.
Далее было необходимо найти Missing residues - остатки, которые не удвалось расшифровать в РСА- эксперименте, например ввиду подвижности соответствующих регионов белка. Для этого в PDBe проводился поиск по структурам плохого разрешения ("Resolution > 3"). В выбранной структуре человеческого аквапорина (PDB ID: 4csk) c разрешением 3.28 Å таких остатков оказалось 59 (Рис. 3).
В этой же структуре нашелся пример и для следующей задачи, где было необходимо найти несовпадения последовательностей природного белка из Uniprot и белка, который кристаллизовали для РСА-эксперимента. В поле SEQRES прописана последовательность белка, по которому была получена структура, в поле DBREF - кросс-референсы между ней и последовательностями из других баз данных, в поле SEQADV - все найденные между ними различия. Рассматриваемый белок имеет только один вариант последовательности в Uniprot. Последовательность, по которой нарабатывался белок для кристаллизации, отличается наличием экспрессионного тега (Рис. 4).
В последнем задании требовалось найти в PDB-файле худший и лучший B-фактор в структуре. B-фактор (температурный фактор) - число, характеризующее надежность определения координат атома. Хорошими являются значения 10-20. Если же фактор для атома больше 60, то биологических выводов по его положению делать не стоит.
На рисунке 5 показаны строчки PDB-файла структуры человеческой прокаспазы-7 (PDB ID: 1k88), в которых прописаны атомы с лучшим (зеленый) и худшим (красный) B-фактором. Значения B-фактора для всех атомов этой структуры довольно большие, что свидетельствует о высокой степени "размазывания" электронной плотности.