Совмещение
С помощью сервиса PDBeFold, производящего структурные выравнивания белков с моделями из PDB, был произведен поиск структурных гомологов белка Fis1 (PDB ID: 3o48), использованного в предыдущих практикумах. Сервис выдал для Fis1 127 находок, из которых 12 имели RMSD 0,8-3 Å и N_align 50-90%. Из них для дальнейшей работы были выбраны 4 гомолога (Таблица 1).
| Таблица 1. Структурные гомологи 3o48, найденные PDBeFold | ||||||
| PDB | Название | Организм | N_match | N_align | N_align/N_query | RMSD |
|---|---|---|---|---|---|---|
| 1pc2:A | Human mitochondria fission protein | Homo sapiens | 152 | 113 | 0,876 | 1.64 |
| 1iyg:A | Fis1p-like and CGI-135 homologous domain | Mus musculus | 133 | 116 | 0,899 | 1.82 |
| 3sz7:A | Sgt2 TPR domain | Aspergillus fumigatus | 151 | 106 | 0,822 | 2.40 |
| 2vgx:B | Type III Secretion Translocator Chaperone SycD | Yersinia enterocolitica | 145 | 100 | 0,775 | 2.36 |
Файл с совмещёнными структурами: send.pdb
На рисунке 1 показано совмещение выравненных структур. В целом, они достаточно хорошо накладываются друг на друга за исключением петель и более длинных, чем у 3о48, концевых неструктурированных участков отдельных гомологов.
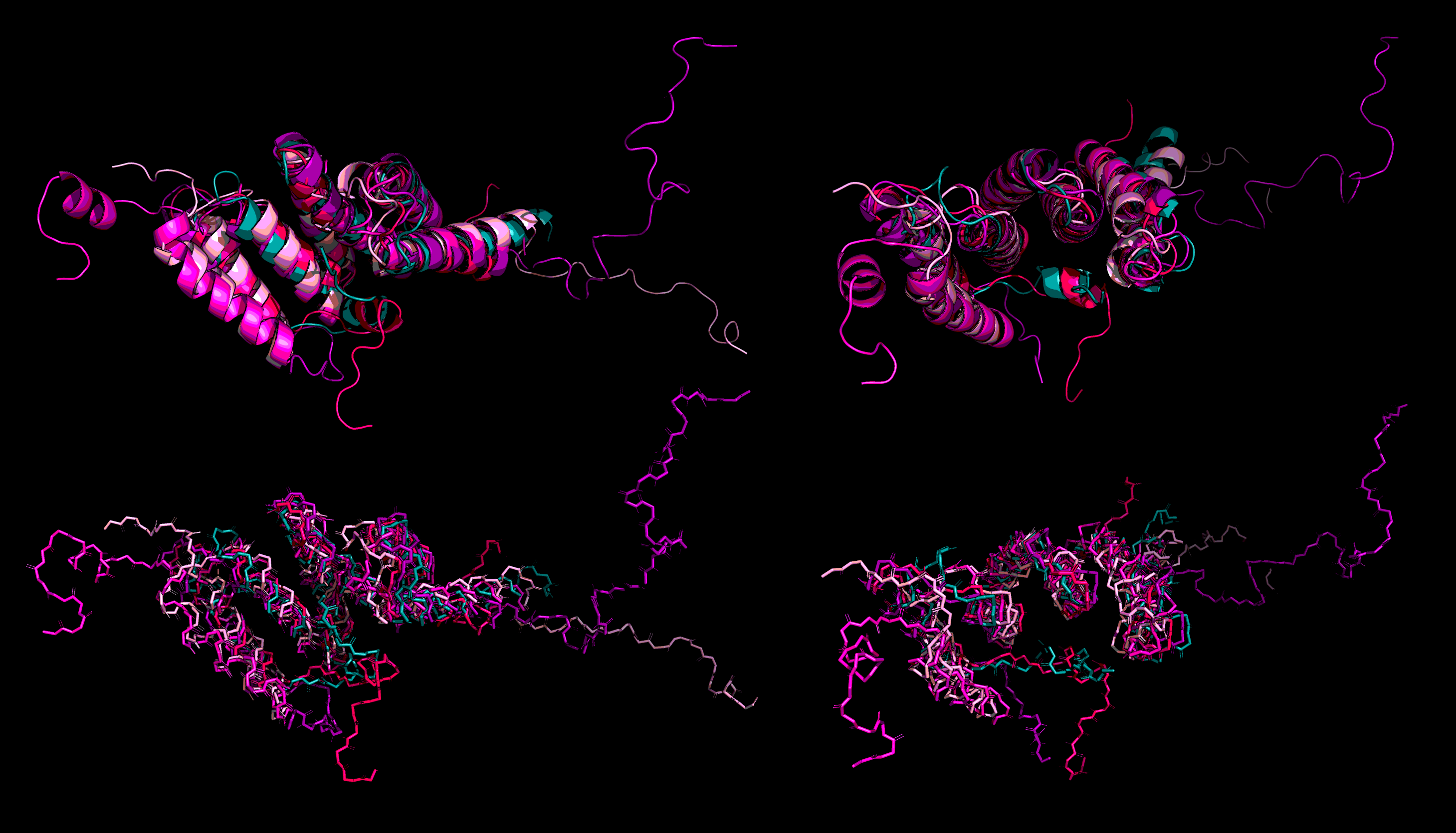 |
| Рисунок 1. Пространственное cовмещение структурных гомологов 3о48 в двух проекциях в изображении cartoons и backbone. Deepteal - 3о48, hotpink - 1iyg:A, purple - 1pc2:А, magenta - 2vgx, lightpink - 3sz7 |
Выравнивания отличаются достаточно сильно. Визуально, в выравнивании по последовательностям несколько меньше гэпов и невыровненных остатков.
 |
| Рисунок 2. Структурное выравнивание гомологов Fis1 |
 |
| Рисунок 3. Выравнивание гомологов Fis1 по последовательностям, построенное Muscle |
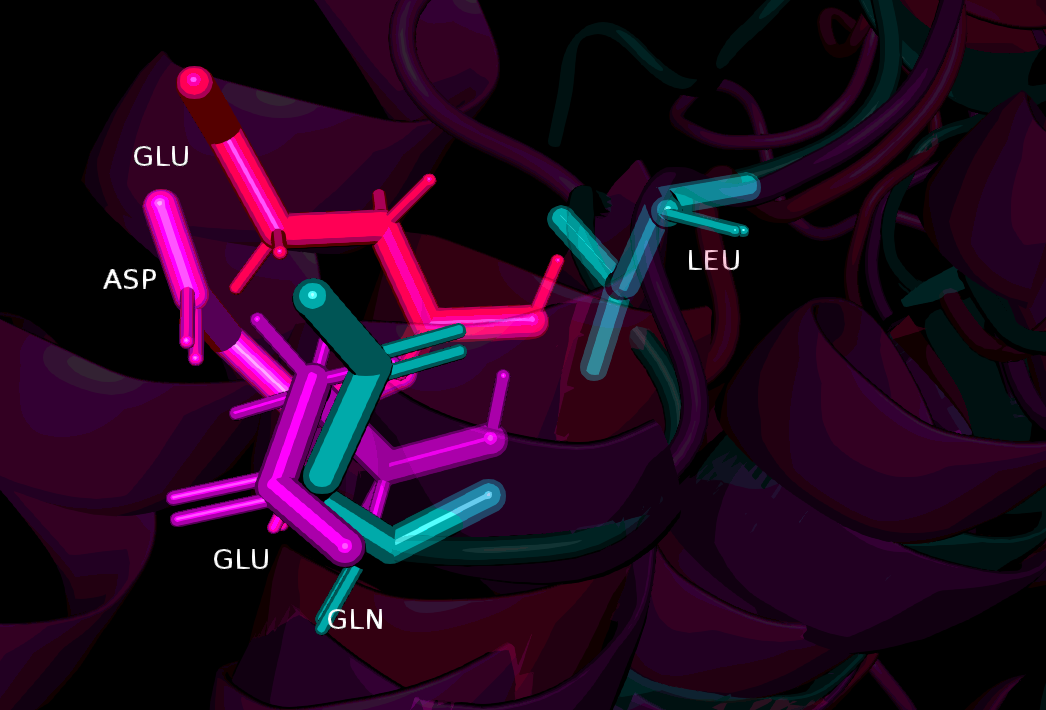 |
| Рисунок 4. Остаток Gln в 3о48 (бирюзовый), выравненный с остатками Glu и Asp из 1iyg, 1pc2 и 2vgx (оттенки розового) по структуре, но не по последовательности. Справа - Leu из 3о48, стоящий в аналогичном положении в выравнивании последовательностей |
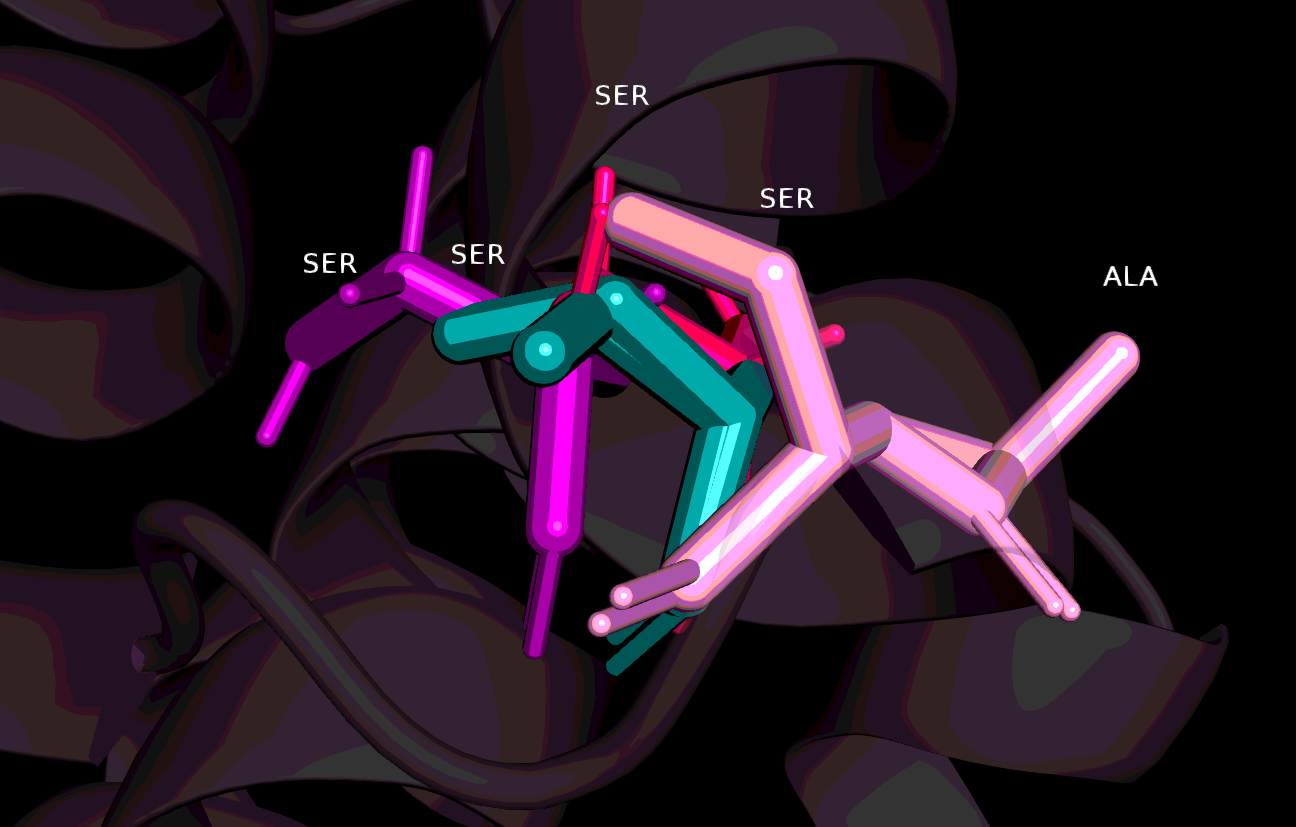 |
| Рисунок 5. Остаток Ser 3sz7 (светло-розовый), выравненный с остатками Ser из 1iyg, 1pc2 и 3о48 по структуре, но не по последовательности, и остаток Ala 3sz7, выравненный с остатками Ser из 1iyg, 1pc2 и 3о48 по последовательности, но не по структуре |
Рассмотрим несколько различающихся позиций подробно.
Так, в позиции 40 структурного выравнивания в 3о48 стоит Gln, выравненный с тройкой отрицательно заряженных полярных аминокислот (EED) из других гомологов. В выравнивании последовательностей с этой же тройкой выравнен Leu (позиция 19). Посмотрим, как данный участок выглядит на совмещении структур (Рис. 4). Полярные остатки EED из структур 1iyg, 1pc2 и 2vgx соответственно показаны в оттенках розового, два варианта выровненного с ними остатка (Gln "по версии структурного выравнивания" и Leu "по версии выравнивания последовательностей") показаны бирюзовым. Видно, что Gln, предсказанный структурным выравниванием, довольно хорошо совмещается c остатками из других гомологов и вместе с ними лежит в участке α-спирали, в то время как предсказанный Muscle Leu находится далеко от остальных остатков и лежит в петле. Скорее всего, более правильным в данном случае является выравнивание по структуре.
В позиции 80 структурного выравнивания наблюдается 4 серина в структурах 3о48, 1iyg, 1pc2 и 3sz7. В соответствующей колонке выравнивания по последовательностям на месте Ser 3sz7 стоит аланин. На рисунке 5 представлено изображение данных остатков в совмещенных структурах. В данном случае "альтернативные" остатки являются соседями по цепи. Разумеется, Ser из 3sz7 (светло-розовый), накладывается на все другие Ser лучше, чем Ala из той же структуры, однако по расположению в пространстве Ala отстоит не особенно далеко, к тому же в выравнивании по последовательностям он совпадает с Ala из гомолога 2vgx (на рисунке не показан). Поэтому в этой ситуации сложно однозначно сказать, какое из выравниваний более правильное.
.