Поиск последовательностей
Для выполнения задания, за основу, мною была выбрана бактерия Nonlabens marinus S1-08. Запрос "Nonlabens" с фильтрами Reference и chromosome+.Выпало 4 результата. Наиболее интересным результатом является выравнивание геномов Nonlabens marinus S1-08 и Nonlabens ponticola. Были взяты хромосома Nonlabens marinus S1-08 с AC в GenBank:AP014548.1 и хромосома Nonlabens ponticola с AC в GenBank:CP034549.1.
Локальное сходство
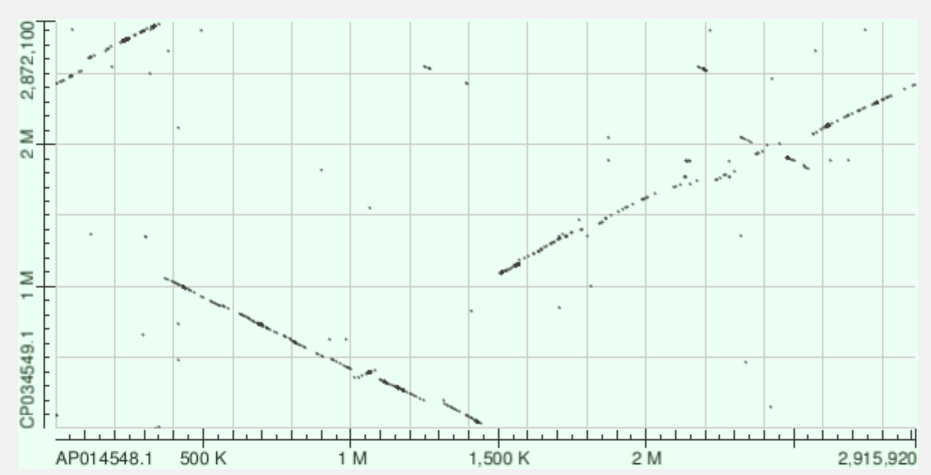
Рисунок 1: Карта локального сходства хромосом Nonlabens marinus S1-08 (ось X) и Nonlabens ponticola (ось Y), построенная алгоритмом megablast
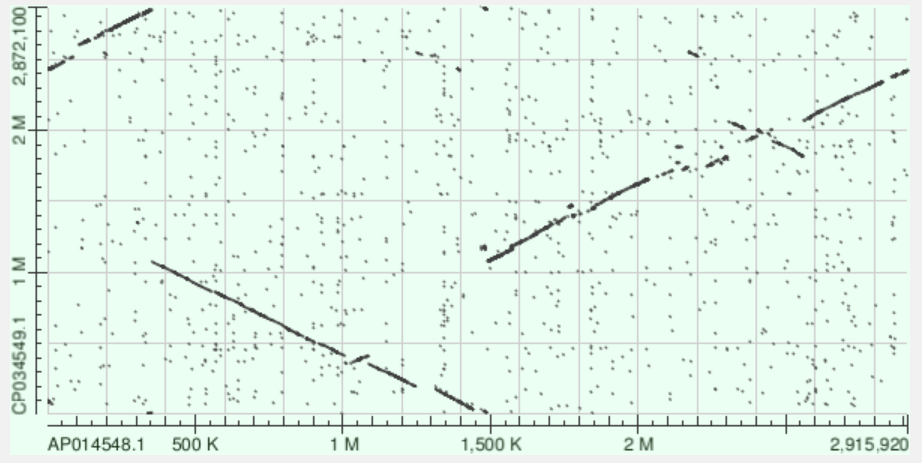
Рисунок 2: Карта локального сходства Nonlabens marinus S1-08 (ось X) и Nonlabens ponticola (ось Y), построенная алгоритмом blastn
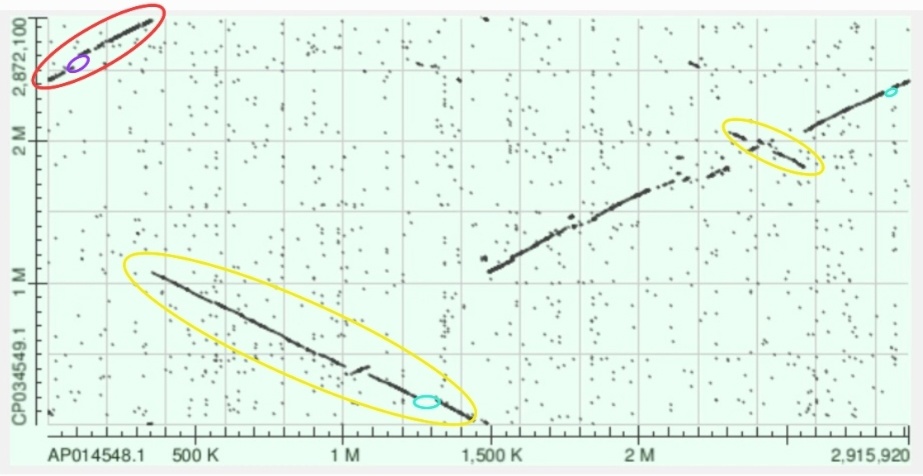
Рисунок 3: Карта локального сходства Nonlabens marinus S1-08 (ось X) и Nonlabens ponticola (ось Y), построенная алгоритмом blastn
Из карты локального сходства можно сделать вывод,
что последовательности выровнены хорошо практически по всей длине.
Красным отмечен участок, показывающий, что ориджины двух хромосом не совпадают.Но если мысленно перенести этот вправо, то он совпарает с верхним концом линии выравнивания.
Желтым отмечены инверсии.Бирюзовым отмечены делеции, а фиолетовым вставка.
В выдаче blastn появляется множество отдельных точек, что говорит о многочисленных локальных сходствах.