1.Загрузка и создание.Подготовка чтений
по секвенированию бактерии Buchnera aphidicola, который был предоставлен мне, - SRR4240360.
Скачен файл был с помощью команды:
wget ''ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/000/SRR4240360/SRR4240360.fastq.gz''
Затем был создан файл с адаптерам Illumina для одноконцевых чтений:
cat /mnt/scratch/NGS/adapters/* > adapters.fasta
Потом выполняем чистку чтений от возможных остатков адаптеров:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240360.fastq.gz SRR4240360_noadapt.fastq ILLUMINACLIP:adapters.fasta:2:7:7
Удаляем с концов чтений нуклеотиды качества ниже 20 и оставляем чтения длины не менее 32 нуклеотидов:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240360_noadapt.fastq SRR4240360_trimmed.fastq TRAILING:20 MINLEN:32
2.Запуск программы velveth
Подготавливаем к-меры для построения графов:
velveth ./velv 31 -fastq -short SRR4240360_trimmed.fastq
3.Запуск программы velveth
Построение графов де Брёйна и сборка контигов:
velvetg ./velv
N50 = 43070. Самыми длинными контигами оказались NODE_1 (113474) с покрытием 33.5,
NODE_5 (91818) с покрытием 36.3 и NODE_4 (64155) с покрытием 31.6.
Также среди контигов выделяется NODE_10 (50333) с покрытием 35.8.
4.Анализ контигов
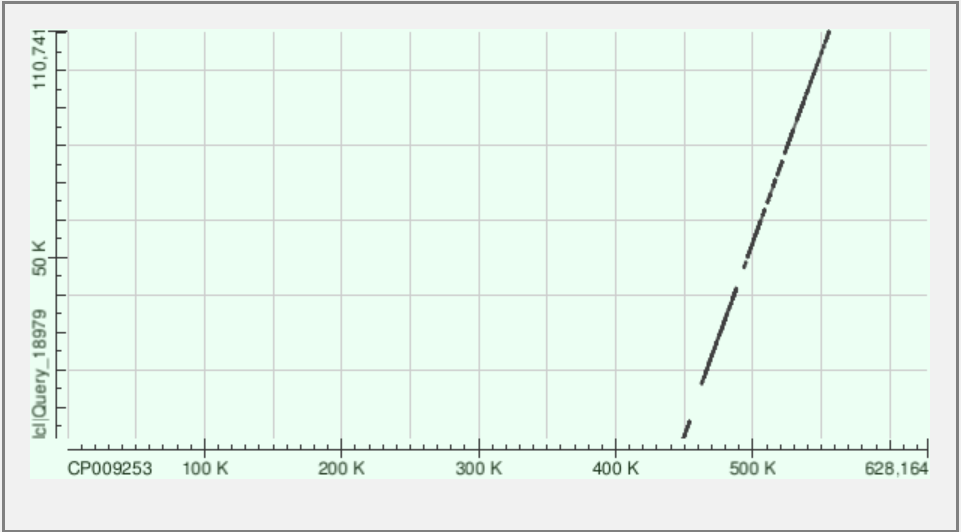
Рисунок 1.DotPlot NODE_1
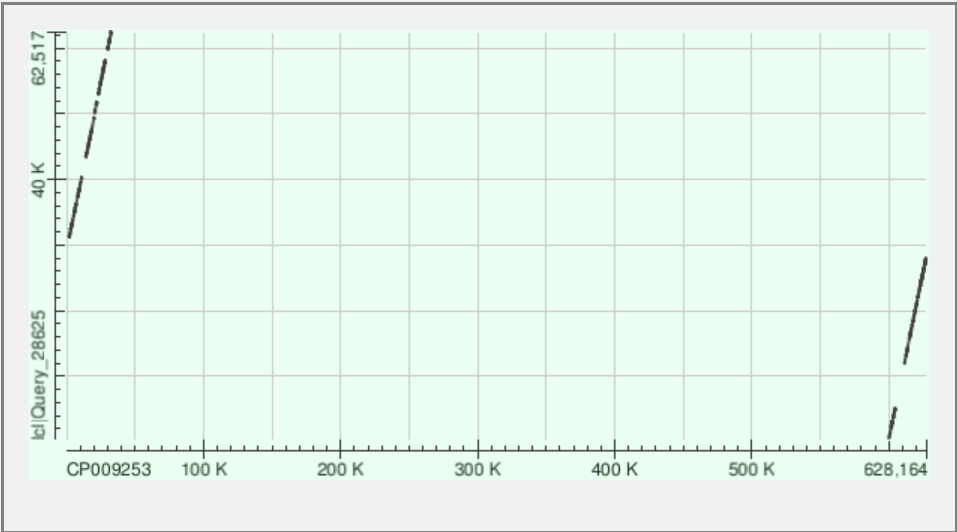
Рисунок 2. DotPlot NODE_4
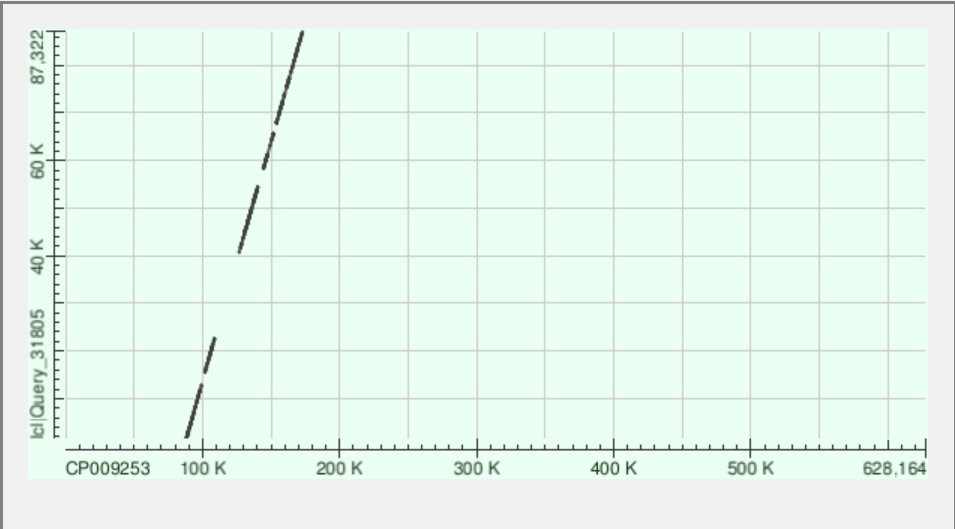
Рисунок 3. DotPlot NODE_5
Самые длинные контиги были проанализированны с помощью megablast. Контиг NODE_1 располагается ближе к концу записи из банка. Контиг NODE_4 ложится на начало записи из банка, но претерпевает разрыв, так как за начало кольцевой хромосомы бактерии авторы выбрали другой регион. Контиг NODE_1 располагается в первой трети записи из банка. Контиги выравнились фрагмантами. Это может говорить о том, что в геноме есть неконсервативные регионы.