Выбор короткого контига и различные варианты BLAST
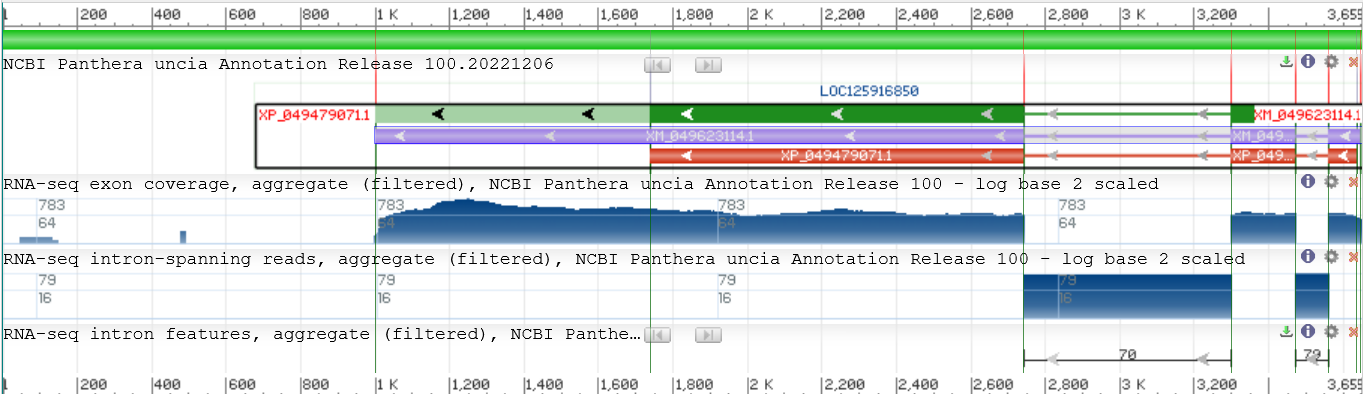
Для работы была выбран контиг хромосомы 1 Panthera uncia с индентификатором NW_026057871 с длиной 3655 bp. На этом участке расположен ген " Voltage-dependent T-type calcium channel subunit alpha-1H-like" (XP_049479071.1), есть CDS, состоящий из 3 участков.
Контиг сохранен в файл
Рисунок 1: Выбранный участок хромосомы.
1.Blastn - алгоритм, который принимает на вход нуклеотидную последовательность и ищет похожие последовательности из нуклеотидной базы данных, обычно применяется для поиска гомологичных последовательностей ДНК или РНК, например, не кодирующих белки.
blastn: исключен таксон Млекопитающие - Mammalia(taxid 40674). Использовались значения по умолчанию (длина слова 11,E-value 0,05) и максимальное число находок 5000. Всего получено находок 932. В начале очень много представителей класса птиц(Aves).
2. Megablast - алгоритм, который принимает на вход нуклеотидную последовательность и ищет очень похожие последовательности из нуклеотидной базы данных, обычно применяется для поиска близких видов или,например, для картирования чтений на геном.
megablast: исключен таксон Млекопитающие - Mammalia(taxid 40674). Использовались значения по умолчанию (длина слова 24,E-value 0,05) и максимальное число находок 5000. Всего получено находок 188. Среди находок много представителей класса птиц(Aves) и костных рыб ( Osteichthyes). Также много находок у Thunnus maccoyii (Тунец южный синий).
3.Blastx - алгоритм, который транслирует заданную нуклеотидную последовательность и осуществялет поиск по белковым базам данных, что используется для посика гомологичных белков.
blastx:исключен таксон Млекопитающие - Mammalia(taxid 40674). Использовались значения по умолчанию (длина слова 6,E-value 0,05) и максимальное число находок 5000. Всего получено находок 1892. Очень много находок среди пресмыкающиеся(Reptilia), особенно черепах.
4. Tblastx - агоритм, который транслирует полученную нуклеотидную последовательность в белок и ищет схожие последовательности в нуклеотидной базе данных, транслированной в шести рамках считывания, что используется для нахождения похожих непредсказанных белков.
tblastx
Попытка 1: исключен таксон Млекопитающие - Mammalia(taxid 40674). Использовались значения по умолчанию (длина слова 3,E-value 0,05) и максимальное число находок 5000. Всего получено находок 0 .
Попытка 2: исключен таксон Хищные - Carnivora (taxid:33554).
спользовались значения по умолчанию (длина слова 3,E-value 0,05) и максимальное число находок 500. Всего получено находок 0 .
Попытка 3: исключен таксон Кошачьи - Felidae (taxid:9681).
спользовались значения по умолчанию (длина слова 3,E-value 0,05) и максимальное число находок 500. Всего получено находок 0.
Попытка 4: исключен таксон род Пантеры - Рanthera (taxid:9688).
спользовались значения по умолчанию (длина слова 3,E-value 0,05) и максимальное число находок 500. Всего получено находок 0.
Поиск в геноме эукариота генов основных рибосомальных РНК по далекому гомологу
Последовательность генома Panthera uncia была проиндексирована командой
makeblastdb -in GCF_023721935.1_Puncia_PCG_1.0_genomic.fna -dbtype nucl
Были выданы последовательности 16S рРНК и 23S рРНК
Локальный поиск BLAST отдельно для каждой рРНК по полученной базе данных:
blastn -task blastn -query 16s_e.coli.fasta -db GCF_023721935.1_Puncia_PCG_1.0_genomic.fna -out blastn_16S.out -outfmt 7
blastn -task blastn -query 23s_e.coli.fasta -db GCF_023721935.1_Puncia_PCG_1.0_genomic.fna -out blastn_23S.out -outfmt 7
Для выполнения задачи был использован blastn, так как исходная последовательность не является белок-кодирующей, а виды не близкие. После применения команды были получены файлы
blastn_16S.out и
blastn_23S.out, в первом 5 находок, во втором - 10.
Для 16S РНК из 5 находок гомологов не нашлось. 3 из 5 - неаннотированные. А оставшиеся 2 пренадлежат мРНК. Для всех находок E-value от 0.024 до 3.6 , что говорит о случайности находок.
Для 23S тоже гомологов не нашлось. Из 10 находок 8 не аннотированны, а 2 пренадлежат мРНК.Для всех находок E-value от 0.56 до 6.9 , что говорит о случайности находок.