Осознание баз данных: Начало
Все началось с того, что ночью я нашла переданных мне список 12 белков. Походило все на квест, так как непонятно что делать с этими данными. Но затем я увидела перечень баз данных и решила, что выбор это сложно. Пусть за меня базу данных выберет рандом. Пусть GO - 1, STRING - 2, Human Protein Atlas - 3, KEGG - 4, Reactome - 5, GeneCards - 6, Panther - 7. К сожелению, у меня не получилось сделать рандомайзер на своей страничке 😟. Мне выпало число 2, следовательно я буду работать с базой данных STRING.
STRING
На вход подается файл с ID белков. Затем выплыл запрос про интересующую меня кладу или организм. Так как я все же человек, а человек это млекопитающие, то выбрала я Mammals. Все двенадцать белков были найдены у человека, шимпанзе, орангутана и серой крысы. Обычно все смотрят именно на человека и его "ближайших родственников". А я посмотрю на крысу, они вседь очень милые.

Рис 1. Rattus Norvegicus
| ID | Название |
|---|---|
| ALPI | Щелочная фосфатаза, кишечная |
| ALPL | Щелочная фосфатаза, тканенеспецифический изофермент |
| ALPP | Щелочная фосфатаза, плацентарная |
| ALPPL2(ALPG) | Щелочная фосфатаза, подобная ALPP |
| GCH1 | Циклогидролаза 1 |
| PCBD1 | Птерин-4-альфа-карбиноламиндегидратаза |
| PCBD2 | Кофактор димеризации ядерного фактора гепатоцитов 1 альфа |
| PTS | 6-пирувоилтетрагидробиоптеринсинтаза |
| QDPR | Дигидроптеридинредуктаза |
| SPR | Сепиаптеринредуктаза |
| TPH1 | Триптофан-5-монооксигеназа |
| TPH2 | Триптофан-5-монооксигеназа |
На выдаче был получен граф, содержащий значимо больше взаимодействий, чем случайный. В нём очень много рёбер, обозначающий текс-майнинг, что мешает анализу. Поэтому эти рёбра были убраны. Также можно понять, что ждя всех белков, которые кодируют эти гены существует 3D структура (что приятно).
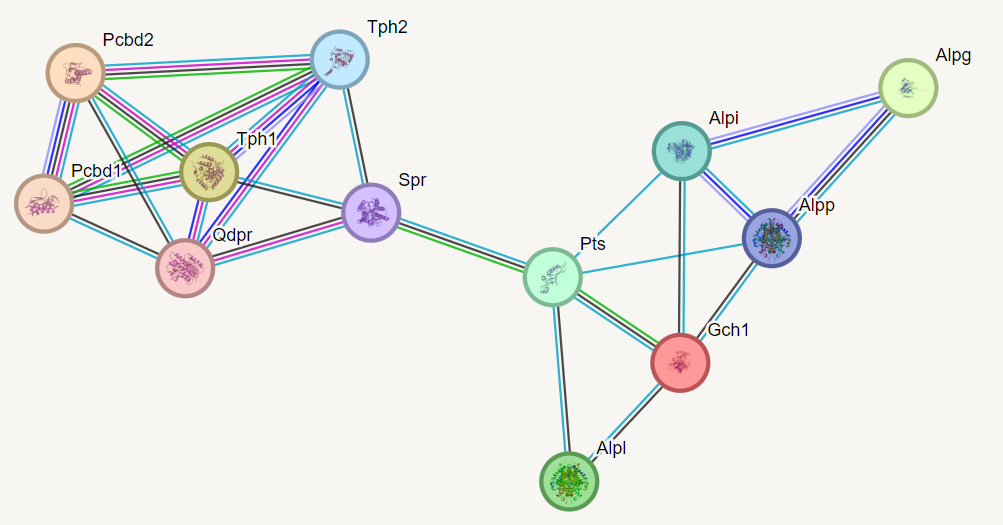
Рис 2. STRING network, включающая текст-майнинг
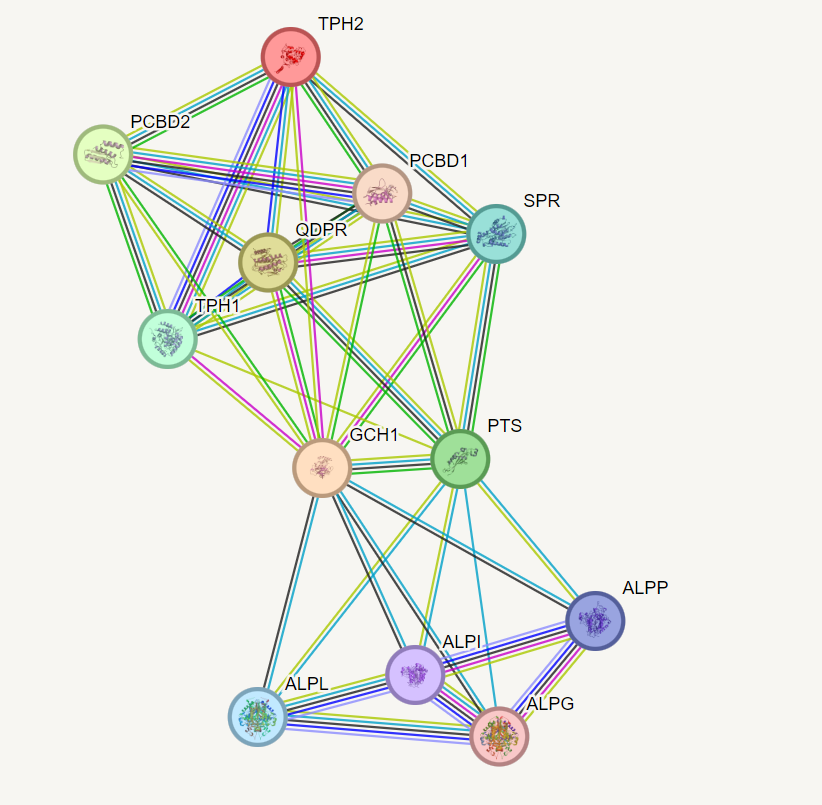
Рис 3. STRING network без текст-майнинга
Также можно заметить, что гены как бы разделены на два кластера, связанных друг с другом взаимодействием SPR и PTS. Это не удивительно, так как оба участвуют в биосинтезе тетрагидробиоптерина , а также показано, что они гомологичны (лиловая линия).
Пусть первый кластер - гены, взаимодействующие с SPR. В нем все гены взаимодействуют со всеми и большенство из них эксперементально потверждено. Интересно посмотреть на пары TPH1/TPH2 и PCBD1/PCBD2. Показано что они гомологичны, а также гены перекрываются (синии линии).
Втрой кластер - гены, взаимодействующие с PTS. Там уже на так все красиво, но можно увидеть, что гены ALPI, ALPP, ALPG, являющиеся щелочными фосфотазами - гомологи и имеют перекрывающиеся участки (хотя это все основано на кукируемых баз данных (бирюзовый )). Странно, что ALPL, которая также является щелочной фосфотазой, не имеет никаких взыимодествий с другими фосфотазами.

Рис 4. Распространенность белков
Стало интересно посмотреть совместное появление генов. Не удивительно, что они все есть у Opisthokonta(человеческие же). Также оказалось, что у меня консервативные гены (неожиданно)
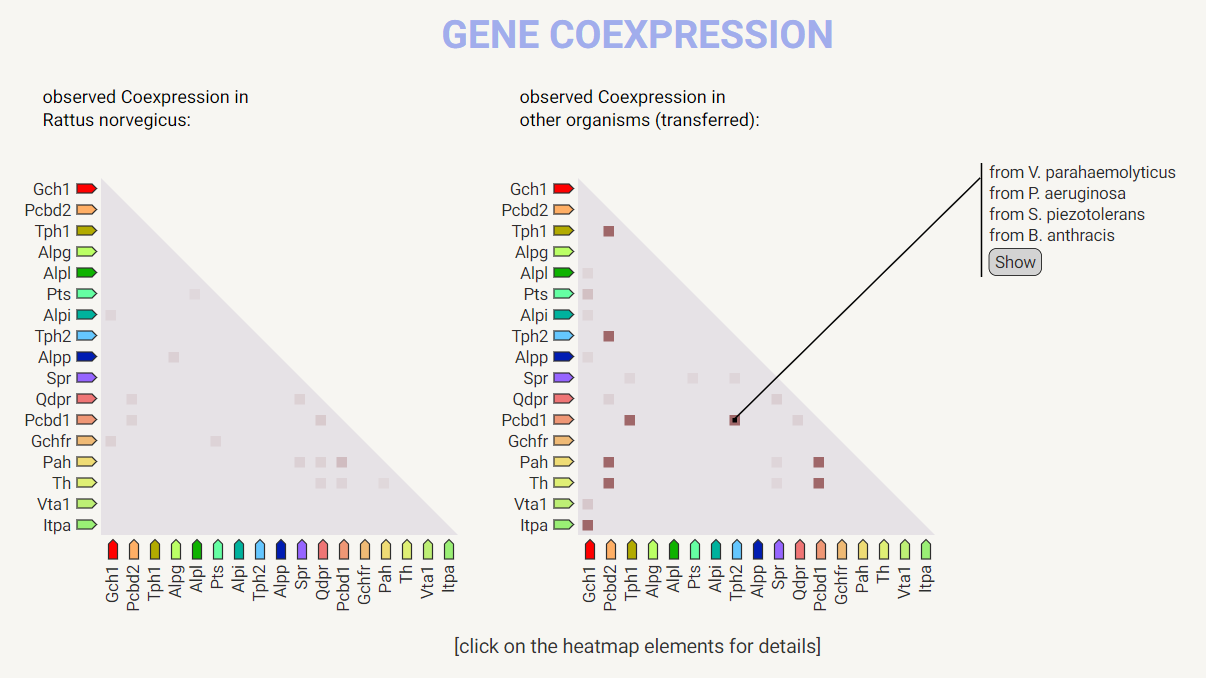
Рис 5. Схема совместной экспрессии
У крысы конечно все грустно, но вот в других организмах (в основном в бактериях) можно увидеть, что у некторых генов совместная экспрессия.