Белок
Мне был выдан белок DES_BACSU. Этот белок катализирует введение двойной цис-связи насыщенных жирных кислот, прикрепленных к мембранным фосфолипидам. у Bacillus subtilis. Для данного белка были скачаны последовательность и трёхмерная структура, предсказанная с помощью AlphaFold.
Задание 1
В базе данных OPM был найден белок, содержащий β‑бочонок: транспортер жирных кислот FadL. Этот белок пронизывает внешнюю мембрану E. coli и переносит длинноцепочечные жирные кислоты из внешней среды в периплазму(Рис.1). Красная - положительно заряженная сторона мембраны. Синяя - отрицательно заряженая сторона мембраны.
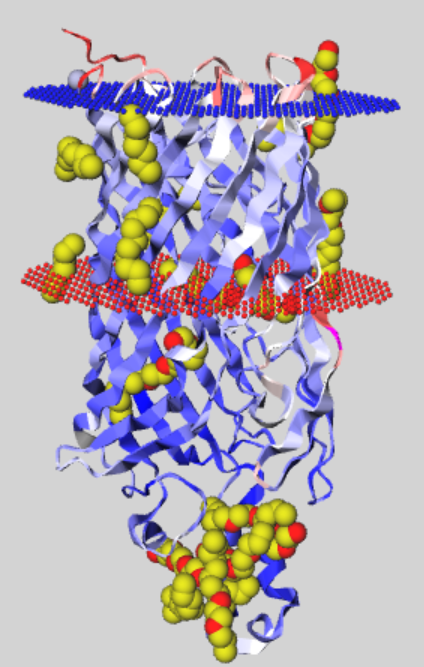
Рис 1. Полная структура FadL
Таблица 1. Параметры из OPM
| Имя белка | Fatty Acid Transporter FadL |
| PDB ID | 3pgs |
| Uniprot ID | FADL_ECOLI |
| Толщина гидрофобной части белка в мембране | 24,6 Å |
| Координаты трансмембранных участков в OPM | 43-53, 78-87, 94-101, 126-135, 142-148, 208-216, 223-229, 274-281, 289-295, 325-334, 341-348, 367-376, 383-390, 411-421 |
| Среднее количество остатков в одном β-тяже белка | 8 |
| Мембрана | Внешняя мембрана бактерии |
Задание 2
С помощью сервиса DeepTMHMM были предсказаны положение альфа-спирального DES_BACSU и бета-листовогого FADL_ECOLI.
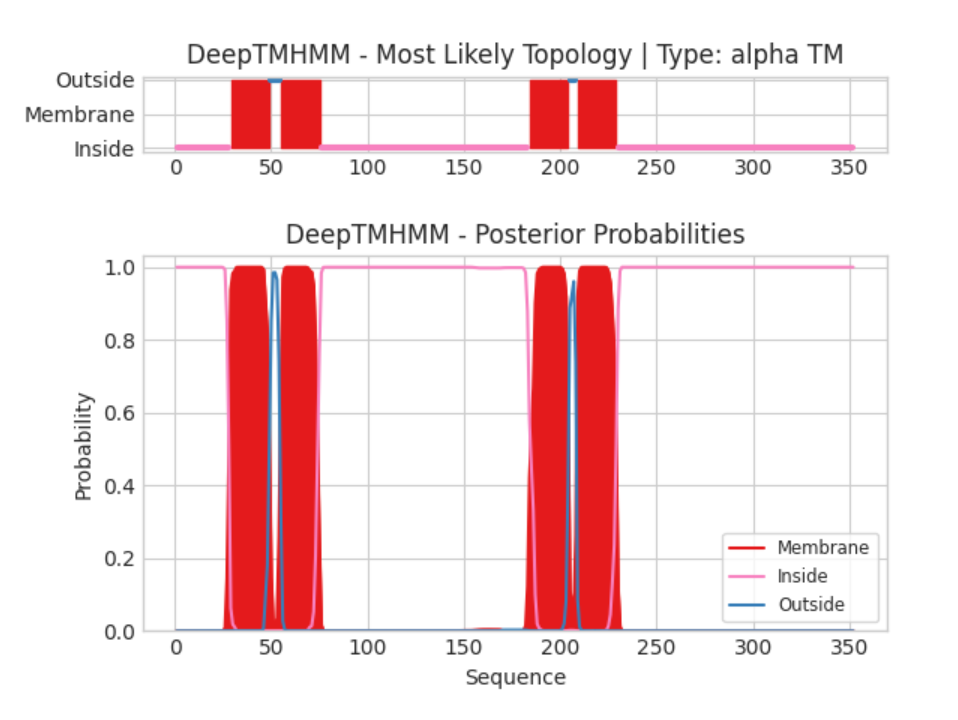
Рис 2. Предсказание трансмембранных элементов альфа-спирального белка DES_BACSU по последовательности белка. По оси x обозначены номера последовательности. По оси y - вероятность нахождения остатка относительно мембраны там, где показано цветом соответсвенно легенде.
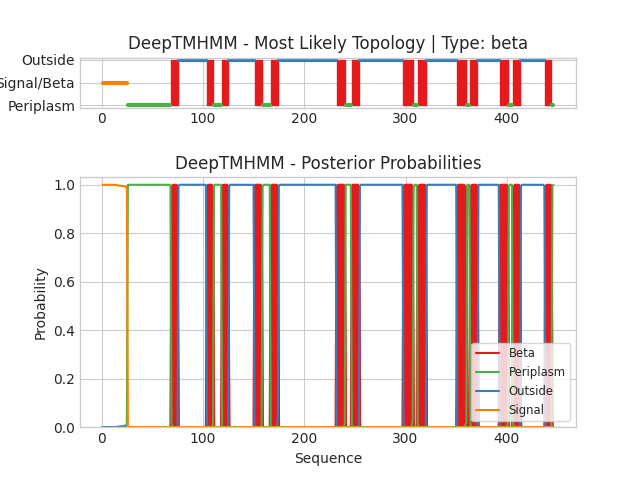
Рис 3. Предсказание трансмембранных элементов бета-листовогого белка FADL_ECOLI по последовательности белка. По оси x обозначены номера последовательности. По оси y - вероятность нахождения остатка относительно мембраны там, где показано цветом соответсвенно легенде.
Задание 3
Алгоритм PMM былы запущен для альфа-спирального белка DES_BACSU с параметрами:
Number of Membranes: 1
Type of membrane: Gram-positive bacteria inner membrane
Allow curvature: no
Topology (N-ter): in (согласно результату DeepTMHMM N-конец внутри)
| Толщина гидрофобной части белка в мембране | 31.3 ± 1.0 Å |
| Координаты трансмембранных участков | 1(28-50), 2(54-72), 3(184-205), 4(206-226) |
| Среднее количество остатков в одном α-спирали белка | 20,3 |
| Локализация белка | Мембрана грам‑положительной бактерии |
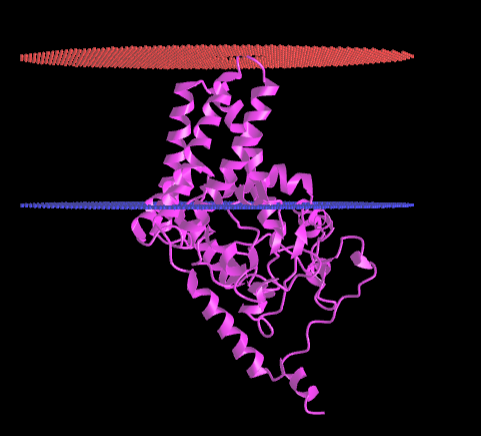
Рис 4. Предсказание положения белка DES_BACSU в мембране с помощью PPM. Красным цветом обозначена положительно заряженная сторона мембраны, направлена во внеклеточное пространство. Синим цветом - отрицательно заряженая сторона мембраны, направлена в сторону цитоплазмы.
Задание 4
При сравнении результатов предсказания для DES_BACSU, полученных с помощью алгоритма PMM и сервиса DeepTMHMM, можно заметить, что количество трансмембранных участков одинаковое, но их координаты отличаются, но не сильно.
PMM: 1(28-50), 2(54-72), 3(184-205), 4(206-226)
DeepTMHMM: 1(29-49), 2(55-75), 3(184-204), 4(209-229)
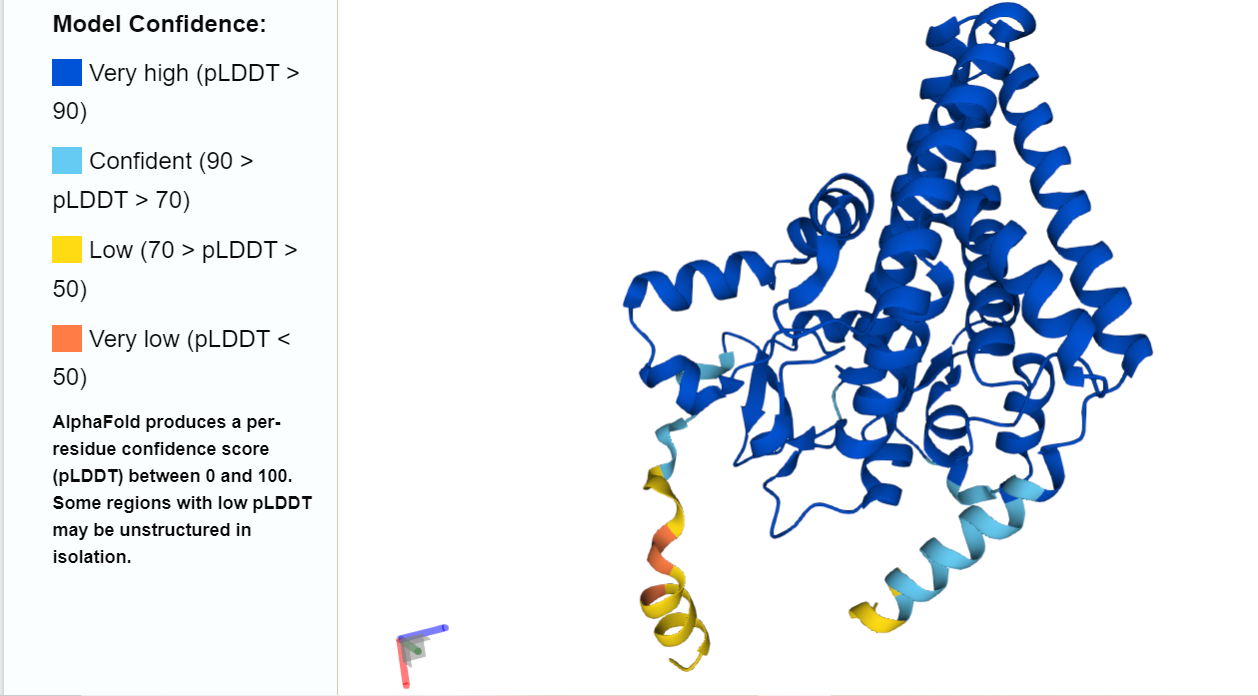
Рис 5. Визуализация исследуемого белка DES_BACSU с выделенными по уровню доверия аминокислотными остатками
Можно заметить, что N и C концы предсказаны с довольно невысоким уровнем доверия, но так как они не входят в ТМ участки, это не должно было повлиять на работу программы PPM. Также можно сказать, что предсказания воспроизводими и предположить, что оба сервера справляются довольно хорошо.