Задание №1: Составление списка гомологичных белков, включающих паралоги
Для нахождения белков, гомологичных
CLPX_ECOLI (АТФ-связывающая субъединица ClpX протеазы Clp Escherichia coli) среди последовательностей протеомов бактерий, выбранных в первом задании, использованы следующие команды:
makeblastdb -dbtype prot -in proteomes.fasta
blastp -query P0A6H1.fasta -evalue 0.001 -outfmt 7 -db proteomes.fasta 1>> proteins.txt
Найдено 28 гомологов.
Таблица 1. Найденные гомологи белка CLPX_ECOLI
| Мнемоника | Белок |
|---|---|
| RUVB_BIFLO | Holliday junction branch migration complex subunit RuvB |
| RUVB_ARTS2 | Holliday junction ATP-dependent DNA helicase RuvB |
| A0K236_ARTS2 | AAA ATPase, central domain protein |
| A0LW31_ACIC1 | AAA ATPase, central domain protein |
| FTSH_ACIC1 | ATP-dependent zinc metalloprotease FtsH |
| Q6NF92_CORDI | Cell division protein |
| FTSH_RUBXD | ATP-dependent zinc metalloprotease FtsH |
| A0LRB8_ACIC1 | ATP-dependent zinc metalloprotease FtsH |
| Q47KU4_THEFY | Peptidase M41, FtsH precursor |
| Q8G3S2_BIFLO | ATP-dependent zinc metallopeptidase involved in cell division |
| A0JR82_ARTS2 | ATP-dependent zinc metalloprotease FtsH |
| Q6ACQ0_LEIXX | Cell division protein |
| B0RHW4_CLAMS | cell division protein ftsH homolog [Clavibacter michiganensis subsp. sepedonicus] |
| Q6NGK1_CORDI | Hypothetical protein |
| Q47MZ2_THEFY | ATPase precursor |
| A0K1M3_ARTS2 | ATPase AAA-2 domain protein |
| Q1AY82_RUBXD | ATPase AAA-2 |
| Q1AU05_RUBXD | ATPase AAA-2 |
| Q8G871_BIFLO | Protease |
| Q6NFB1_CORDI | ATP-dependent Clp protease ATP-binding subunit |
| CLPX_BIFLO | ATP-dependent Clp protease ATP-binding subunit ClpX |
| Q1AVT0_RUBXD | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_CORDI | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_LEIXX | TP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_CLAMS | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_ARTS2 | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_ACIC1 | ATP-dependent Clp protease ATP-binding subunit ClpX |
| Q47MU4_THEFY | ATPase regulatory subunit |
Задание №2: Реконструкция и визуализация
Для построения деревьев была использована программа NGPhylogeny.fr: MAFFT → FastME
Параметры:
Gamma distributed rates across sites - No
Starting tree - BIONJ
No refinement
Number of bootstrap replicates - 100
Скобочная формула.
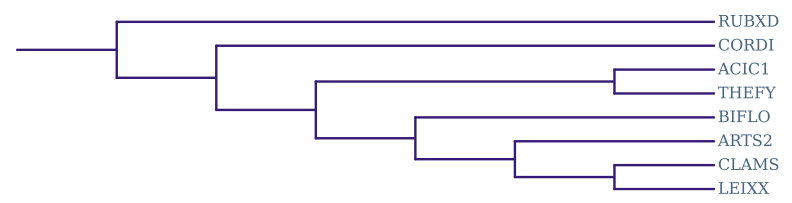
Рис. 1 Филогенетическое дерево бактерий.
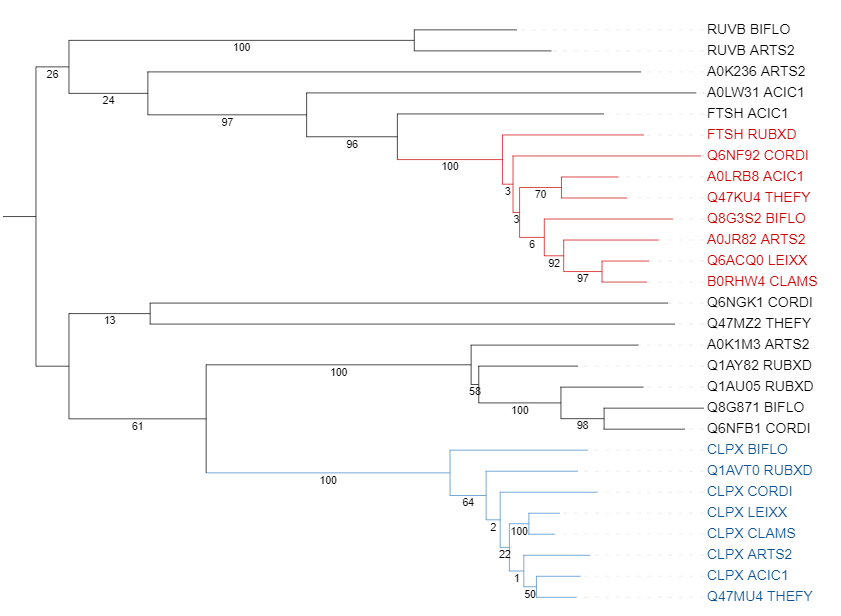
Рис. 2 Филогенетическое дерево гомологов белка CLPX_ECOLI. Ортологические и паралогические группы отмечены разными цветами,
числами показаны bootstrap-поддержки соответствующих ветвей.
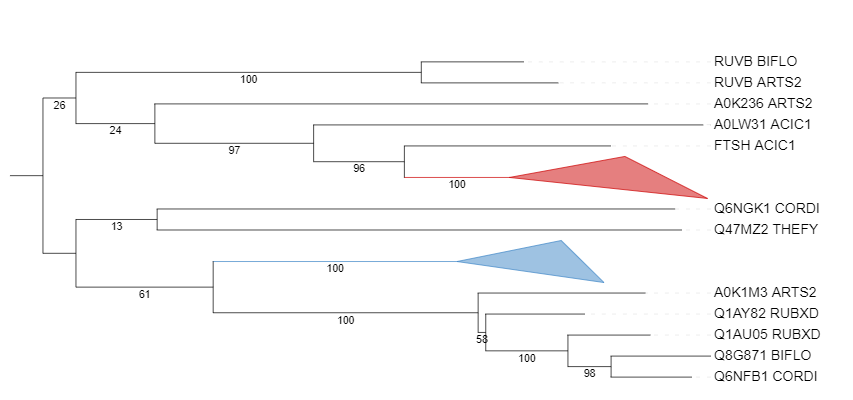
Рис. 3 Филогенетическое дерево гомологов белка CLPX_ECOLI. Ортологические группы "схлопнуты".
Дерево переукренено в среднюю точку.
Пары ортологов: CLPX_LEIXX и CLPX_CLAMS, RUVB_BIFLO и RUVB_ARTS2, A0LRB8_ACIC1 и Q47KU4_THEFY.
Пары паралогов: A0LRB8_ACIC1 и FTSH_ACIC1, Q1AY82_RUBXD и Q1AU05_RUBXD, A0JR82_ARTS2 и A0K236_ARTS2.
На основе описания белгов можно выделить две ортологические группы: ATP-dependent Clp protease ATP-binding subunit ClpX и ATP-dependent zinc metalloprotease FtsH .
Также можно сравнить ортологические группы с эталонным деревом (Рис. 1).
Начала рассмотрим группу ATP-dependent Clp protease ATP-binding subunit ClpX. Можно заметить, что в группу попали белки из всех восьми бактерий, но реконструированная филогения неполностью совпадает с эталонной, но при этом часть нетривиальных ветвей правильные.
Теперь рассмотрим группу ATP-dependent zinc metalloprotease FtsH . В нее попали все белки из восьми бактерий, а также реконструированная филогения полностью совпадает с эталонной.