BLAST
Поиск гомологов
Была взята последовательность YP_003762680.1. На сайти NCBI на страничке последовательности был запущен BLAST -> blastp -> поиск в соответствующей базе данных (Uniprot/SwissProt). В результате было найдено некоторое количество гомологичных последовательностей (рис.1), для каждой из которых в таблице выделена строчка, где, помимо названия последовательности (description - описания), присутствуют такие обозначения, как:
- Max score: наибольший вес выравнивания
- Total score: общий вес выравнивания находки и исходного белка
- Query cover: процент аминокислотных остатков исходной последовательности, покрытых выравниванием
- E-value: expect value - математическое ожидание находки с тем же и большим весом в банке случайных последовательностей. Характеризует то, насколько случайно совпадение найденной последовательности исходной. Чем меньше значение E-value, тем ошибка менее вероятна
- Ident: процент совпадающих аминокислот
- Accession: идентификатор последовательности находки
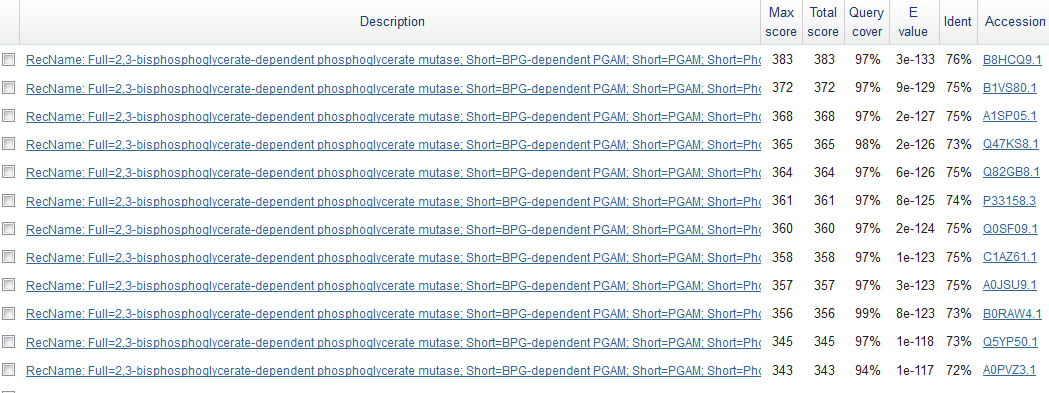
Рис.1: Выдача BLAST
Была выбрана такая находка, чтобы она удовлетворяла параметрам: Identity 40-70%, coverage < 100%, E-value < 0.00001 и не = 0.0. Находка: Identity 68%, Query coverage 92%, E-value = 5e-113, само выравнивание представлено на рисунке 2.
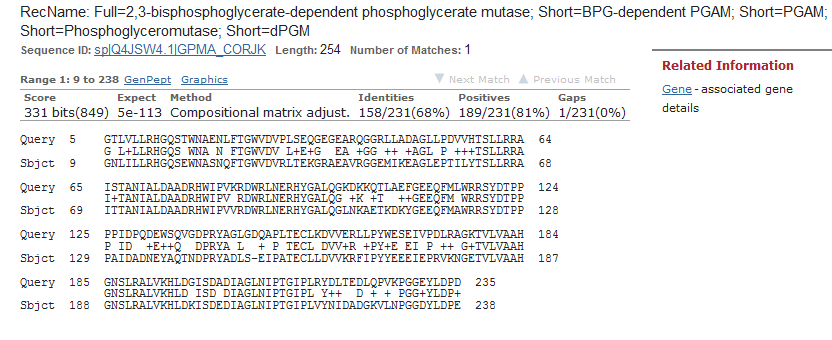
Рис.2: Выравнивание находки и исходной последовательности
Построение карты локального сходства
Для построения карты локального сходства была загружена последовательность находки, описанная выше. Далее на страничке бласта была выбрана опция Align two or more sequences, а в полученном результирующем окне - Dot Matrix. Результат на рисунке 3.
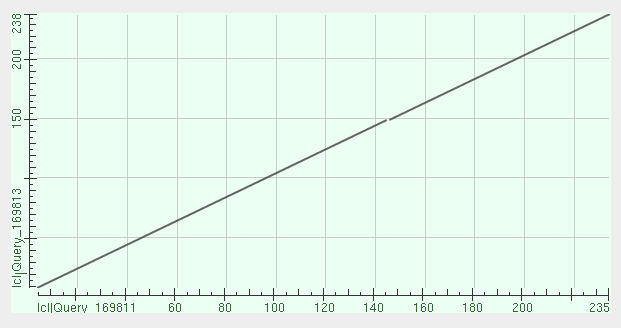
Рис.3: Карта локального сходства
По Х-оси находится нумеровка остатков исходной последовательности, по У - находки. Прямо посередине прямой мы можем увидеть разрыв, который соответствует гепу в 149 позиции последовательности находки. По данной карте хорошо видно, что последовательности гомологичны, негомологичных участков нет.
Эукариотические гомологи последовательности
Проведем поиск BLAST по эукариотам. Для это в поле организм выберем Eukaryota. В результате нашлось 27 находок (E-value меньше 0.001), фрагмент таблицы находок представлен на рисунке 4.
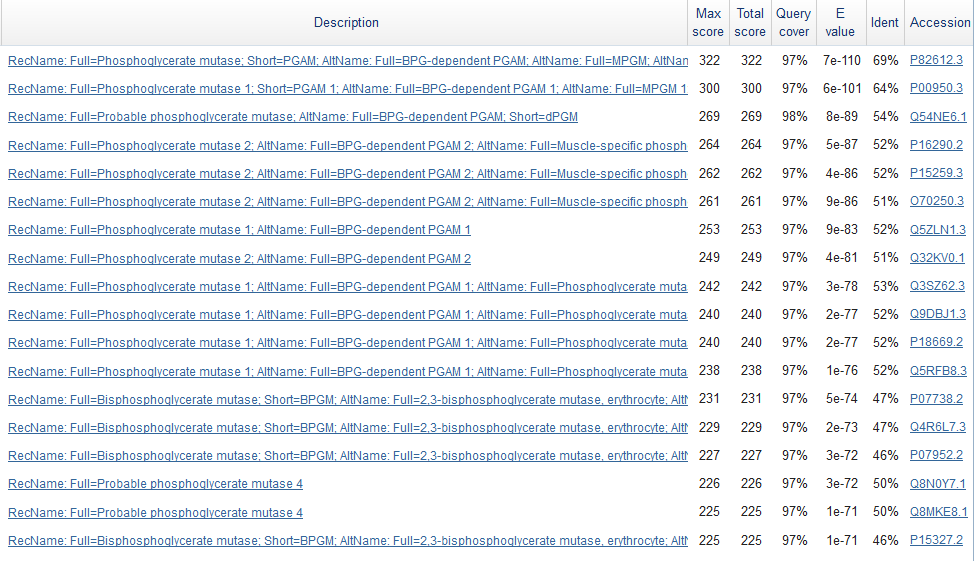
Рис.4: Фрагмент таблицы находок гомологов белка в эукариотах
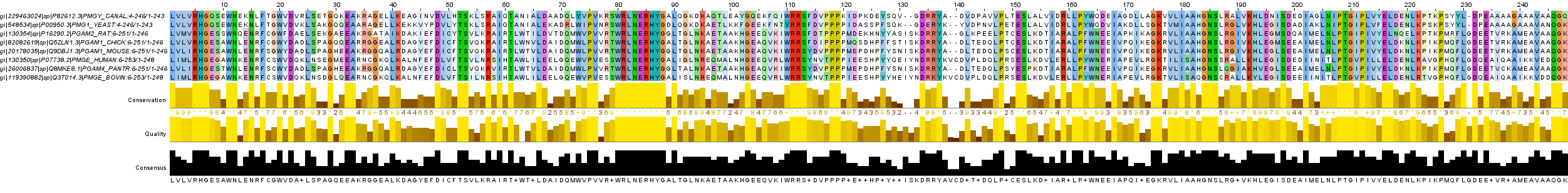
Далее были скачаны 8 найденных гомологов (файл). Общая длина выравнивания составила 248 аминокислотных остатков. Из них консервативных: 73 (29,4%) и функционально консервативных:115 (46,4%). Ниже представлено выравнивание этих восьми последовательностей и проект.