Множественное выравнивание
Выборка
При помощи программы BLAST и PSI-BLAST была собрана выборка из 6 белков, гомологичных моему. В файле представлены по 3 последовательности, найденные программами BLAST и PSI-BLAST и первоначальная последовательность моего белка. Идентичные белки выбирались "на глаз" - ID на уровне 50-60%, e-value < 1e-5, все белки принадлежат бд SwissProt.
Построение и сравнения выравниваний
Сперва были построены два множественных выравнивания: первое - при помощи программы muscle на kodomo (команда muscle -in both.fasta -out muscle.fasta);второе - при помощи программы mafft (mafft both.fasta > mafft.fasta). Результаты работы обеих программ были открыты в Jalview и покрашены Clustalx и по консервативности выше 70% (рисунки 1 и 2)

Рис.1: Выравнивание, полученное программой mafft

Рис.2: Выравнивание, полученное программой muscle
Далее одно выраравнивание "подравнивалось" под другое, а именно попыталась множественное выравнивание, полученное muscle, выровнять с таковым, полученным mafft. Для того, чтобы выравнивания полностью совпали, пришлось внести 1 геп в каждую из семи последовательностей из выравнивания muscle. Результат на рисунке 3.
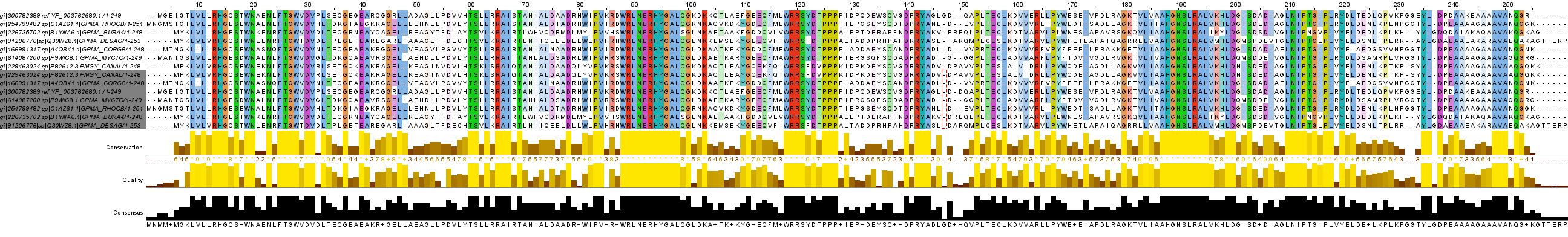
Рис.3: Оба выравнивания
Pfam
Последовательность своего белка запустила на обработку на сайте Pfam для поиска доменов. В итоге было найдено два домена (оба гистодин фосфатазные домены): перывй с 7 по 144 остаток, второй - со 128 по 221. У второго домена достаточно высокое значение E-value 8e-5. Поэтому seed-выравнивание строилось по первому домену. Seed-выравнивание представлено на рисунке 4.

Рис.4: Seed-выравнивание одного из семейств доменов Pfam