Парное выравнивание
Получение парного выравнивания
Было получено множественное выравнивание. Из него получено парное выравнивание таким образом: удалены все последовательности, кроме двух последних, затем удалены колонки с гепами.

Рис.1: Фрагмент парного выравнивания
Построение глобального выравнивания
Для построения глобального выравнивания использовалась команда needle на kodomo. На выходе были получены два файла: один формата needle, другой - fasta. Фрагмент глобального выравнивания представлен на рисунке 2. Как видно, есть небольшие различия между первоначальным выравниванием и таковым, полученным по алгоритму Needleman-Wunsch global alignment.
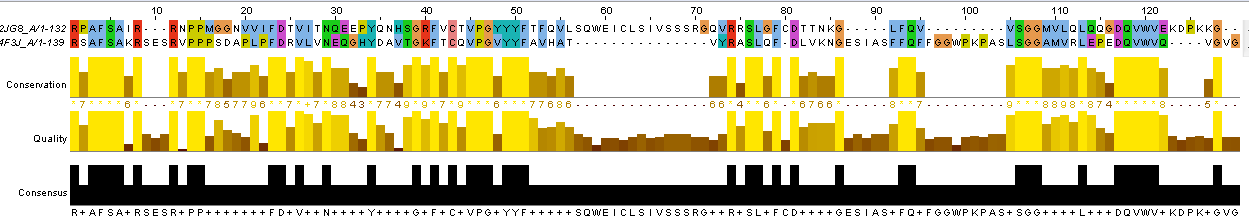
Рис.2: Фрагмент глобального выравнивания
Построение наилучшего локального выравнивания
Для построения локалього выравнивания использовалась команда water на kodomo. Она использует алгоритм Смита-Ватермана для построения локального выравнивания двух последовательностей. Результат в fasta-формате и графическое изображение выравнивания на рисунке 3. Полученное локальное выравнивание не отличается от такого глобального.
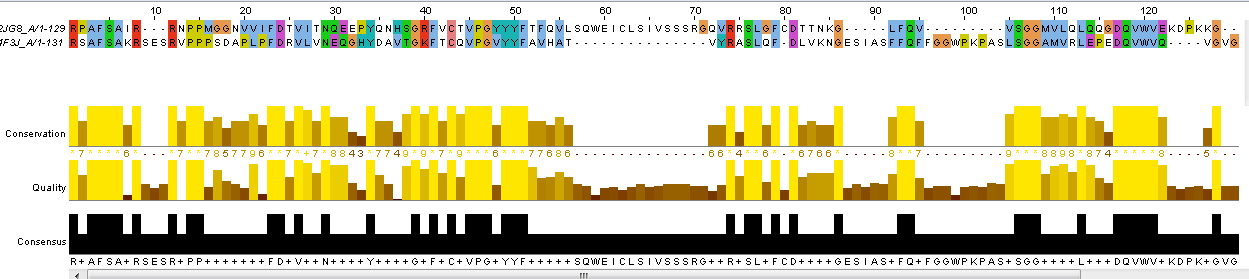
Рис.3: Фрагмент локального выравнивания
Выравнивание двух заведомо негомологичных последовательностей
Были выбраны два случайных белка: белок 1 и белок 2. Для них построено локальное(рис.4) и глобальное(рис.5) выравнивания. По ним ясно видна разница в работе двух алгоритмов. При глобальном выравнивани поиск идет по двух последовательностям целиком, поэтому появляются длинные одноцепочечные участки по краям вырвнивания; в локальном для каджого небольшого выровненного участка происходит его "расширение", поэтому полностью выравнивание состоит как бы из "блоков".
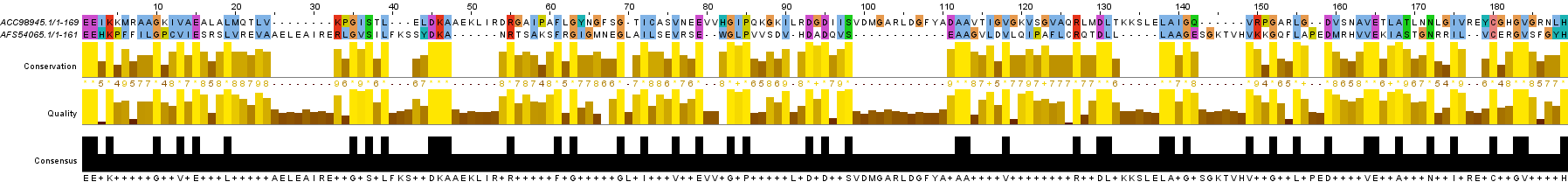
Рис.4:Локальное выравнивание негомологичных последовательностей

Рис.5:Глобальное выравнивание негомологичных последовательностей
Таблица параметров пяти парных выравниваний
| Длина выравнивания | Число и % совпадений | Число и % сходных остатков | Число и % гепов | |
| Начальное парное выравнивание | 144 | 44 = 30,6% | 64 = 44,4% | 17 = 11,8% |
| Глобальное выравнивание | 159 | 50 = 31,4% | 67 = 42,1% | 47 = 29,6% |
| Локальное выравнивание" | 151 | 50 = 33,1% | 66 = 43,7% | 42 = 27,8% |
| Глобальное выравнивание негомологичных белков | 310 | 63 = 20,3% | 101 = 32,6% | 142 = 45,8% |
| Локальное выравнивание негомологичных белков | 189 | 47 = 24,9% | 74 = 39,2% | 48 = 25,4% |
Различия первых трех выравниваний весьма незначительные, причем наибольшее количество совпадений наблюдается в локальном выравниваниии, но это улучшение достигается путем увеличения числа гепов, по сравнению с исходным выравниванием. При сравнении двух последних выравниваний между собой, можно все-таки заметить различия в работ алгоритмов. Как уже описывалось выше, принцип работы отличается, и, хотя процент совпадений различается не так сильно, процент гепов в глобальном выравнии намного превышает таковой в локальном.
Описание отличного участка при компановке двух выравниваний
Необходимо было выровнять результат глобального выравнивания с двумя последовательностями из множественного выравнивания. Первая часть выровнялась хорошо, когда вторая "половина" не "подравнивалась совсем. Сначала был внесен один геп, а затем еще 7. В результате получилось выравнивание, представленно на рисунке 6. После некоторых попыток хорошо выровнять С-конец не получилось.
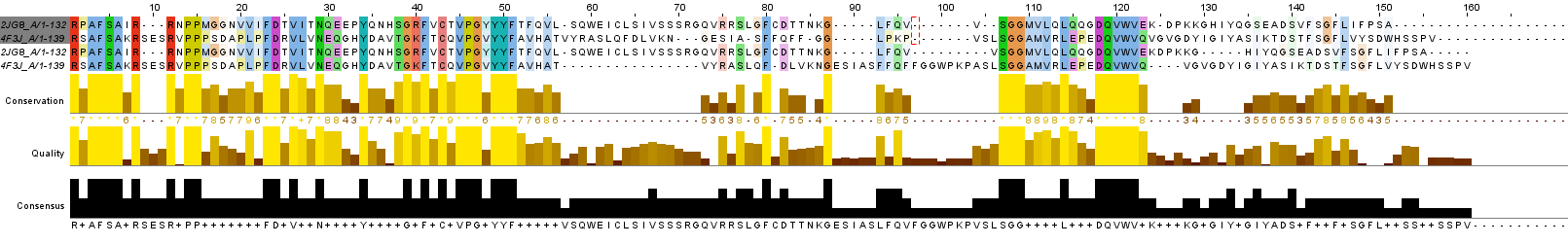
Рис.6:Два выровненных выравнивания
Проверка правильности выравнивания
Следующим заданием предлагалось проверить правильность парного выравнивания из множественного (полученного первоначально) и глобального выравнивания. Для этого в двух окошках JalView были отображены эти два выравнивания, для каждого из них добавлено по две строчки аннотации: первая символами "+" показывает достоверный, по моему мнению, участок последовательности (есть значительное сходство на достаточно протяженном участке); во второй - знаком "S" отмечены те колонки, которые ссответствуют пространственному совмещению структур. Для этого использовалась программа SupCheck: из папки aln_10.fasta были скачаны все файлы, запущена программа sup_check.exe, открыт файл aln_10.pdb в RasMol и запущен скрипт aln_10.scr для первого выравнивания и aln_10_2.scr - для второго. Результаты представлены на рисунках 7 и 8.