Пограмма getorf пакета EMBOSS
entret embl:D89965 - файл с последовательностью матричной РНК из желудка крысы Rattus norvegicus. С помощью командыentret embl:D89965 -auto getorf d89965.entret -table 0 -minsize 90 -find 1
получили набор из 5-ти рамок считывания:
>D89965_1 [66 - 155] Rattus norvegicus mRNA for RSS, complete cds. MQFHPRLPAVLQVCAACDRYASLLPAQRRL >D89965_2 [56 - 169] Rattus norvegicus mRNA for RSS, complete cds. MISDAVSSATASSASSLRSMRSVRQSFASSTAALTRWP >D89965_3 [163 - 432] Rattus norvegicus mRNA for RSS, complete cds. MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHM AVTAYAYYSCHELTPWLRIQSTNPVQKYGA >D89965_4 [218 - 3] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds. MLLRCSNCLNVNWKCIRAIWSKPPLSWQKTGVPIACCANLKHCWQSRMKLHRLSSPVTVT WCSQKTILLLSA >D89965_5 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds. MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGSТретья рамка соответствует записи EMBL для белка. Используя команду seqret sw:POA7B8 получим файл. эта запись соответствует белку из кишечной палочки.Приведенная в записи последовательность сотносится с пятой рамкой считывания, найденной нами ранее.
>HSLV_ECOLI P0A7B8 ATP-dependent protease subunit HslV (3.4.25.2) (Heat shock protein HslV) MTTIVSVRRNGHVVIAGDGQATLGNTVMKGNVKKVRRLYNDKVIAGFAGGTADAFTLFEL FERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVADETASLIITGNGDVVQPENDL IAIGSGGPYAQAAARALLENTELSAREIAEKALDIAGDICIYTNHFHTIEELSYKA >D89965_5 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds. MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGSКак можно объяснить сложившуюся ситуацию?
Возможно, это можно объяснить тем, что во время эксперимента по ошибке была отсеквенирована последовательность гена бактерии, которая может населять различные участки кишечного тракта крысы. Затем было предположено, что это и есть крысиный ген.
Файлы-списки
В задании требовалось получить файл с последовательностями алкогольдегидрогеназ. С помощью командыseqret sw:adh*_*adh.fasta
были получены все доступные в SwissProt последовательности дегидрогеназ. Для получения файла с их универсальными адресами (USA) этих последовательностей использовалась программа:
infoseq adh.fasta -only -usa > list.txt
Из этого файла-списка был создан меньший файл, с адресами только тех последовательностей, которые взяты из данных мне организмов. Использовалась команда:
grep -f org.txt list.txt > list2.txt
Fasta-файл был получен:
seqret @list2.txt adh_n.fasta
Все файлы, полученные в этом задании, можно увидеть здесь:adh.fasta, list.txt, , adh_n.fasta.
EnsEmbl
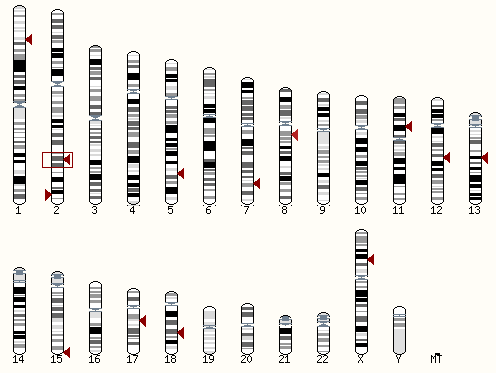
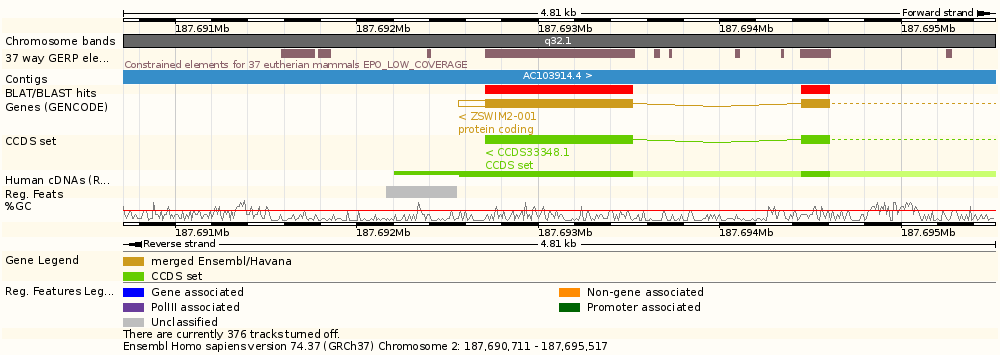
Последовательность гена: gene.fasta. Информацию о гене мы искали на портале EnsEMBL. Для начала, мы воспользовались сервисом BLAST/BLAT. В итоге мы получили несколько блоков.В блоке "Alignment Location vs. Karyotype" можно увидеть расположение участка генома, который выровнялся с данным геном, участок выделен рамочкой. Стрелочками выделены менее удачные выравнивания.
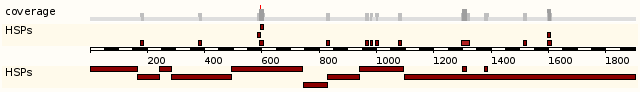
В следующем разделе "Alignment Locations vs. Query" в графическом виде приведена информация о полученном выравнивании.
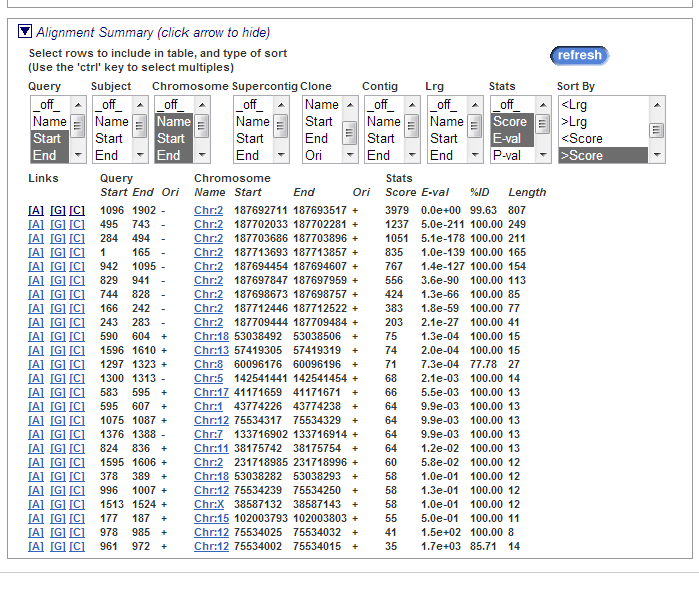
В последнем блоке представлена общая информация о выравнивании.
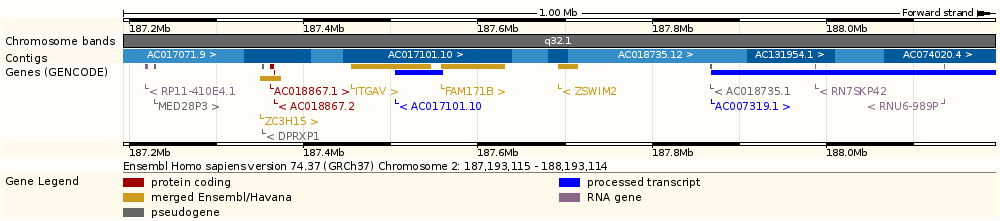
Пройдя по ссылке [C], можно посмотреть информацию о расположенных на участке хромосомы генах, о экзонах и интронах, контигах (Region in detail).Мы можем посмотреть 3 блока.
- Положение на хромосоме
- Более детально описанную область с геном
- Локус хита

© Желудкевич Анна, 2013