Реконструкция филогенетических деревьев.
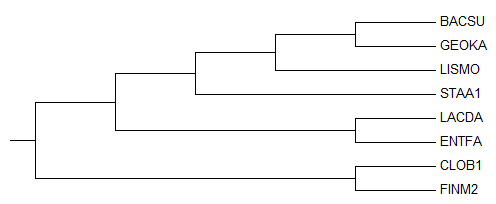
На первом занятии мы отобрали 8 бактерий и построили филогенетическое дерево:
Пользуясь таксономическим сервисом NCBI: http://www.ncbi.nlm.nih.gov/taxonomy/, определили, к каким таксонам относятся выбранные бактерии:
| Название | Мнемоника | Тип | Класс | Отряд | Семейство |
| Clostridium botulinum | CLOB1 | Firmicutes | Clostridia | Clostridiales | Clostridiaceae |
| Finegoldia magna | FINM2 | Firmicutes | Clostridia | Clostridiales | Clostridiales Family XI. |
| Enterococcus faecalis | ENTFA | Firmicutes | Bacilli | Lactobacillales | Enterococcaceae |
| Lactobacillus delbrueckii | LACDA | Firmicutes | Bacilli | Lactobacillales | Lactobacillaceae |
| Bacillus subtilis | BACSU | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Geobacillus kaustophilus | GEOKA | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Listeria monocytogenes | LISMO | Firmicutes | Bacilli | Bacillales | Listeriaceae |
| Staphylococcus aureus | STAA1 | Firmicutes | Bacilli | Bacillales | Staphylococcaceae |
На нашем дереве можно найти ветви, выделяющие отдельные таксоны. Так, например, можно заметить, что классы Bacilli и Clostridia разделены ветвью {CLOB1,FINM2} против {LACDA,ENTFA,STAA1,LISMO,GEOKA,BACSU}. Отряд Lactobacillales(включающий два листа: ENTFA, LACDA) как и отряд Bacillales(включающий четыре листа: BACSU, GEOKA, LISMO, STAA1) выделены соответсявующми ветвями. Семейство Bacillaceae также выделено отдельной ветвью:{GEOKA,BACSU} против {CLOB1,FINM2,LACDA,ENTFA,STAA1,LISMO}.
Множественные выравнивания.
Из списка функций белков была выбрана функция "Шаперонин" (HSLO). Из Swiss-Prot были получены последовательности белков с данной функцией из отобранных ранее бактерий. Все последовательности помещены в единый fasta файл.
Выравнивание отобранных белков можно провести несколькими способами. Я использовала программу JalView.Ниже приведено изображение выравнивания в "блочной" форме с блоками шириной 100 остатков и ракраской по проценту идентичности.

Построенное выравнивание отобранных белков, а также проект JalView были сохранены в соответствующих файлах.
Реконструкция филогенетических деревьев.
В JalView доступны четыре метода для реконструкции филогенетического дерева ( меню Calculate - Calculate tree). Приведенные ниже изображения созданы с помощью программы Mega.
Average Distance Using % Identity.

Neighbour Joining Using % Identity.
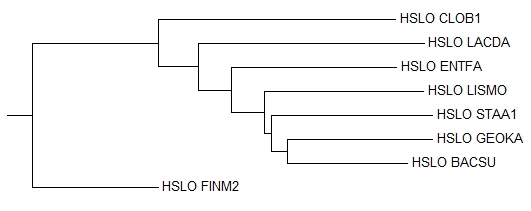
Average Distance Using BLOSUM62.

Neighbour Joining Using BLOSUM62.
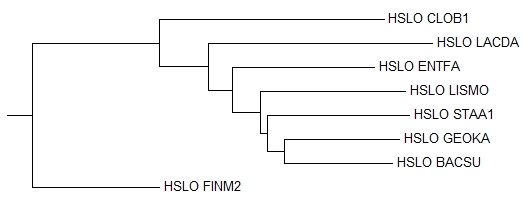
Сравним топологию полученных деревьев с топологией правильного дерева. Курсивом выделим те ветви, которых нет в правильном дереве.
Average Distance Using % Identity.
Нетривиальные ветви:{'BACSU','GEOKA'}vs{'FINM2', 'LACDA','ENTFA','STAA1','CLOB1','LISMO'},
{'BACSU', 'GEOKA', 'STAA1'}vs{'FINM2','LACDA', 'ENTFA','CLOB1','LISMO'},
{'BACSU', 'GEOKA', 'STAA1', 'LISMO'}vs{'FINM2', 'LACDA', 'ENTFA','CLOB1'},
{'BACSU', 'GEOKA', 'STAA1', 'LISMO', 'ENTFA'}vs{'FINM2', 'LACDA','CLOB1'},
{'BACSU', 'GEOKA', 'STAA1', 'LISMO', 'ENTFA', 'LACDA'}vs{'FINM2','CLOB1'},
Neighbour Joining Using % Identity.
Нетривиальные ветви:{'BACSU','GEOKA'}vs{'FINM2', 'LACDA','ENTFA','STAA1','CLOB1','LISMO'},
{'BACSU', 'GEOKA', 'STAA1'}vs{'FINM2','LACDA', 'ENTFA','CLOB1','LISMO'},
{'BACSU', 'GEOKA', 'STAA1', 'LISMO'}vs{'FINM2', 'LACDA', 'ENTFA','CLOB1'},
{'BACSU', 'GEOKA', 'STAA1', 'LISMO', 'ENTFA'}vs{'FINM2', 'LACDA','CLOB1'},
{'BACSU', 'GEOKA', 'STAA1', 'LISMO', 'ENTFA', 'LACDA'}vs{'FINM2','CLOB1'},
Average Distance Using BLOSUM62.
Нетривиальные ветви:{'BACSU','GEOKA'}vs{'FINM2', 'LACDA','ENTFA','STAA1','CLOB1','LISMO'},
{'BACSU', 'GEOKA', 'STAA1'}vs{'FINM2','LACDA', 'ENTFA','CLOB1','LISMO'},
{'BACSU', 'GEOKA', 'STAA1', 'LISMO'}vs{'FINM2', 'LACDA', 'ENTFA','CLOB1'},
{'BACSU', 'GEOKA', 'STAA1', 'LISMO', 'ENTFA'}vs{'FINM2', 'LACDA','CLOB1'},
{'BACSU', 'GEOKA', 'STAA1', 'LISMO', 'ENTFA', 'LACDA'}vs{'FINM2','CLOB1'},
Neighbour Joining Using BLOSUM62.
Нетривиальные ветви:{'BACSU','GEOKA'}vs{'FINM2', 'LACDA','ENTFA','STAA1','CLOB1','LISMO'},
{'BACSU', 'GEOKA', 'STAA1'}vs{'FINM2','LACDA', 'ENTFA','CLOB1','LISMO'},
{'BACSU', 'GEOKA', 'STAA1', 'LISMO'}vs{'FINM2', 'LACDA', 'ENTFA','CLOB1'},
{'BACSU', 'GEOKA', 'STAA1', 'LISMO', 'ENTFA'}vs{'FINM2', 'LACDA','CLOB1'},
{'BACSU', 'GEOKA', 'STAA1', 'LISMO', 'ENTFA', 'LACDA'}vs{'FINM2','CLOB1'},
При сравнении этих деревьев, можно заметить, что они практически не отличаются друг от друга, и, при этом, отличаются от правильного дерева. В правильном дереве CLOB1 и FINM2 выделяются в отдельную кладу, также как и LACDA и ENTFA.
Реконструкция дерева из выравнивания белков в программе MEGA.
Импортируем выравнивание (файл в fasta-формате) в программу Mega (при импорте выберем "Analyze"). Реконструируируем дерево методом "Maximum Parsimony" (кнопка Phylogeny). Укореним дерево в наиболее правильную ветвь (меню Subtree > Root).
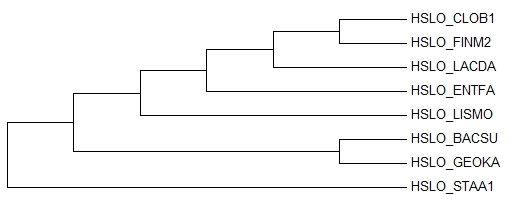
Ниже представлено правильное дерево.

Отличия от правильного:
{CLOB1,FINM2,LACDA} против {ENTFA,LISMO,BACSU,GEOKA,STAA1} {CLOB1,FINM2,LACDA, ENTFA} против {LISMO,BACSU,GEOKA,STAA1}.
В правильном дереве LACDA и ENTFA выделялись в отдельную кладу.