Алгоритмы реконструкции деревьев. Укоренение и бутстреп.
Укоренение в среднюю точку
Укореним в среднюю точку дерево, потроенное на прошлом занятии методом Neighbour-Joining. Для этого воспользуемся программой retree пакета PHYLIP.
1)Скопируем файл с деревом в Newick-формате в файл с именем intree без расширения;
2)Запустим retree;
3)Мы увидим окно, в котором мы должны проверитьт правильность параметров. В нашем случае все верно, поэтому вводим 'Y' и нажимаем Enter. После этого программа выдает нам дерево (пока без изменений) и строчку с вопросом 'NEXT?';
4)Для получения информации введем '?'. В появившемся окне мы видим, что для укоренения дерева в среднюю точку (Midpoint root the tree) нужно нажать 'M', для записи дерева в файл (Write tree to a file) - 'W'. Дерево записалось в файл outtree.
Филогенетическое дерево, построенное методом Neighbour Joining Using % Identity
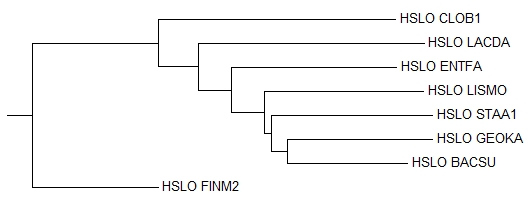
Укорененное в среднюю точку дерево, построенное методом Neighbour Joining Using % Identity
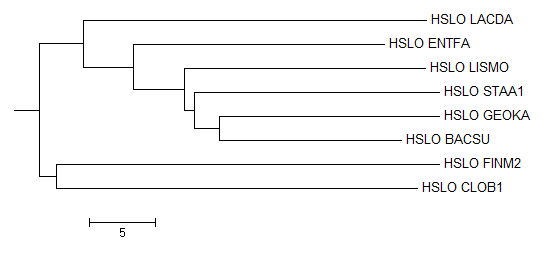
Правильное дерево бактерий
Укоренение можно считать правильным, поскольку оно соотносится с правтльным корнем. Но сами деревья (укорененное и правильное) имеют небольшие различия в ветвях.
Использование внешней группы
Воспользуемся укоренением с использованием в качестве внешней группы белок из кишечной палочки (HSLO_ECOLI). Построим выравнивание последовательностей белков всех бактерий (включая e.coli), отредактируем имена, оставив лишь мнемонику видов и импортируем в MEGA. После реконструкции дерева нужно в меню Subtree выбрать Root и указать в качестве корня ветвь, ведущую к ECOLI. Наконец, чтобы получить изображение укоренённого дерева без ECOLI, нужно воспользовавться кнопкой "Show Subtree Separately" (изображение голубой лупы на фоне дерева на левой панели окна MEGA).
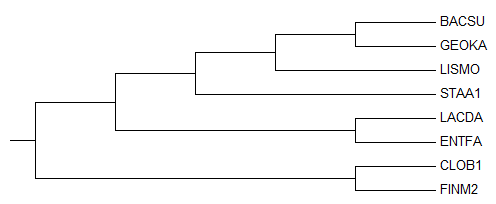
Ниже представлено получившееся дерево:
Укоренение произошло в ветвь {FINM2, CLOB1} против {LACDA, ENTFA, LISMO, STAA1, GEOKA, BACSU}. Такое укоренение не отличается от корня правильного дерева бактерий. Есть небольшое различие в ветвях: на правильном дереве LACDA и ENTFA образуют кладу.
Бутстреп
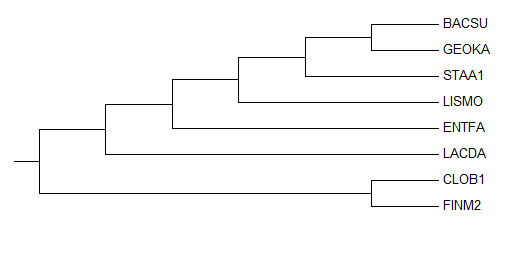
Проведем бутстреп-анализ (количество реплик равное 100) при построении филогенетического дерева. Для этого используем один из методов программы MEGA. Ниже представлено полученное дерево ("Original tree" и "Bootstrap consensus tree" не отличаются).
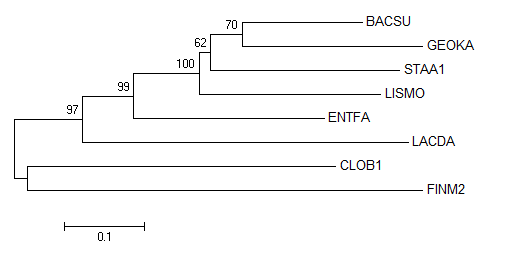
Как можно заметить, неправильные ветви не имеют меньшую поддержку, чем правильные. Отличием от правильного дерева является то, что LACDA и ENTFA не выделяются в отдельную кладу.