Эволюционные домены
Для данной работы на сайте Pfam я выбрала домен .TMP-TENI
Описание выбранного домена из Pfam
TMP-TENI (Thiamine monophosphate synthase/TENI): тиамин монофосфат синтаза (TMP) катализирует замещение пирофосфата в 2-метил-4-амино-гироксиметилпиримидин пирофосфате на 4-метил-5-тиазол фосфата с образованием тиамин фосфата. Это семейство также включает в себя регуляторный белок TENI.
- AC: PF02581
- ID: TMP-TENI
- Ссылка на страницу домена в Pfam
Для домена известна 31 архитектура, для которой доступно 5722 последовательности.
Скачиваем выравнивание из Pfam в JalView. Раскрашиваем выравнивания по консервативности (ClustalX, By conservation 20%). Сохраним полученное выравнивание как проект и файл fasta
Получаем таблицу с информацией об архитектуре всех последовательностей, содержащих выбранный домен PF02581. Использум скрипт: python swisspfam-to-xls.py -z swisspfam.gz -m AC.txt -o teni.xls. Полученная таблица teni.xls
Для работы были выбранны две доменные архитектуры:
1. TMP-TENI - 4922 последовательности
2. Phos_pyr_kin, TMP-TENI - 111 последовательность
В список последовательностей добавим колонки с информацией о таксономической принадлежности. Для этого скачаем полные записи всех последовательностей. Зайдем на сайт Uniprot, пройдем по вкладке Retrieve, введем список AC наших последовательностей из таблицы и нажмем Retrieve. Сохраним файл flat file.
Можно заметить, что некоторые записи были перемещены или вовсе удалены из Uniprot.
Replaced(заменены): H1FS24, H1FZH8, O23128, O23128, Q8L7M9, Q8L7M9, P66916
Deleted(удалены): A6SCD0, A6SCD0, A9EMF5, A9EPY5, A9FNA5, A9GD47, B5J3S1, B5J4Z0, B5K0R8, B5K3X8, B5K4I1, B5RYF5, B5S7I7, B9NIT7, C1NH01, C4FYZ6, D0CHQ2, D1NB69, D1S181, D2MIB4, D5QKB4, D5QR10, D6KVW7, E1Y2L9, E1Y2L9, E4M4U7, E4M4W9, E4MN97, E5BW01, E5YNH1, E7R3M8, E7R3M8, E8KXU7, E8KXU7, E8KXU7, E8KZ08, F0DK18, F0DNV5, F3RH87, F5K201, F5KBU1, F5KBU1, F5KM16, F5KM16, F5KU81, F7KAZ0, F7KAZ0, F7KAZ1, F7PHH9, F7PRR2, F7Q6E4, F7Q6E4, F7QCL4, F9QK29, F9TWX7, F9TWX7, F9TY54, G3YRL5, G4D8P0, G4D8P0, G4D9Q1, G4DEW0, G4DEW0, G4DF47, G4DPE6, G4DPE6, G4DUW7, G4EJV5, G4EJV5, G4FZR9, G4G622, G4GIK8, G4IMR6, G4INC4, G4J898, G4JZ61, G6G8C1, G6GLR5, G6GMN3, G6HL89, G6I1Q4, G6IKF6, G9X6A9, G9X6A9, H0FH84, H0FKQ6, H1HZF5, H1I8D2, H1IMH9, H1IYG3, H1IYG3, H1IYG3, H1J9F1, H1K2H8, H1M2N0, H1M3Z0, H1MQB1, H1MQB1, H1MQB1, H1NWE9, H1NWE9, H1P4S8, H1U9M2, H1UAF0, H1UAF0, H1UAF0, H2WUY4, H4FGT1, H5RQ61, H5W0B0, H7DWJ0, H7E326, I0XYP6, I0YH74, I0YH74, Q1VQ87, Q1VWC8, Q1ZHE4, Q1ZHE4, Q3R202, Q3R4H6, Q3R6I1, Q3R6I1, Q3RB97, Q3RB97
Получим таксономию: python uniprot-to-taxonomy.py -i tax_flat.txt -o tax.txt
Из таксона Bacteria выберем два подтаксона Actinobacteria (A) и Bacteroidetes (B) для архитектуры TMP-TENI и два подтаксона Firmicutes (F) и Proteobacteria (P) для архитектуры Phos_pyr_kin, TMP-TENI.
Из каждой архитектуры были отобраны последовательности из заданных подтаксонов. Они представлены на листе chosen. Получим выравнивания, используя заданный скрипт. Файл с выраниваниями запустим в JalView, удалим пустые колонки. Ниже приведено выравнивание в JalView.
Полное выравниваниеВ домене видны несколько консервативных участков, хотя, по болльшей части, можно увидеть много неконсервативных участков.
Построение филогенетического дерева по выравниванию представителей домена.
Закодируем наши доменные архитектуры: цифрой 1 - TMP-TENI (однодоменную), цифрой 2 - Phos_pyr_kin, TMP-TENI (двудоменную). Закодируем также и наши таксоны Actinobacteria (A), Bacteroidetes (B), Firmicutes (F) и Proteobacteria (P). Отформатируем последовательности для построения дерева.
Дерево построено в программе MEGA методом Neighbour- Joining with % Identity.
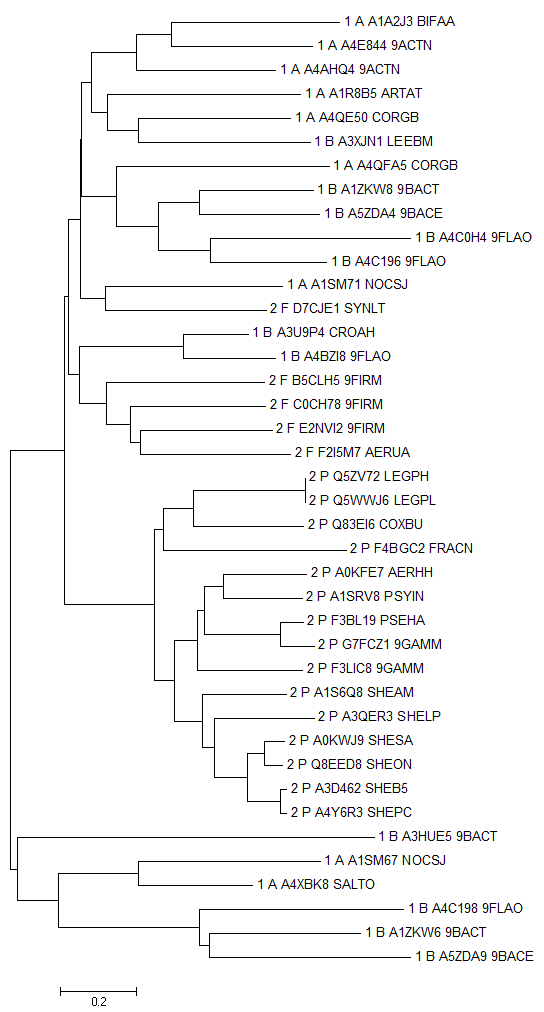
Скобочная формула дерева представлена в файле tree.nwk
С использованием сервиса ITOL была создана следующая окраска листьев:
1_A синий
1_B красный
2_F розовый
2_P зеленый
В файле приведено филогенетическое дерево Circular mode ITOL.
Поскольку первоначально были выбраны разные подтксоны для двух архитектур, то мы не можем увидеть, как шла эволюция архитектур с выбранным доменом внутри одного таксона. По полученному дереву можно предположить, что двудоменная архитектура произошла от однодоменной. При этом двудоменные белки четко распределяются по кладам (2F и 2P). Сильно разветвленные ветви среди однодоменных белков, вероятно, говорят о высокой степени полиморфности.