Вторичная структура тРНК(PDB ID: 1IL2) была предсказана тремя способами: наиболее точным способом, с использованием программ find_pair, analyze пакета 3DNA описан в предыдущем практикуме, с помощью программы einverted из пакета EMBOSS, позволяющей найти инвертированные участки в нуклеотидных последовательностях, а также с помощью программы RNAfold из пакета Viena Rna Package реализующей алгоритм Зукера. Данные приведены ниже, в Таблице 1. | ||||||||||||||||||||||||
| ||||||||||||||||||||||||
| На рисунке ниже представлена визуализация единственной находки прогаммы. | ||||||||||||||||||||||||
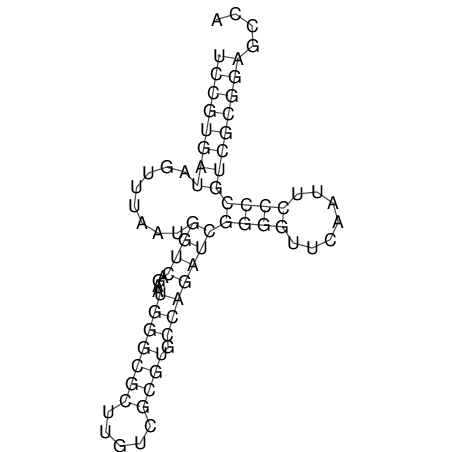 | ||||||||||||||||||||||||
ПОИСК ДНК-БЕЛКОВЫХ КОНТАКТОВПомимо этого для был осуществлен поиск ДНК-белковых контактов в комплексе ДНК-белок (PDB ID: 1ODH), также описанном в предыдущем практикуме. Связи между атомами белка и ДНК были найдены с помощью программы для визуализации молекул JMol, позволяющей выделять определенные группы атомов в заданной структуре. Ниже приведен Jmol-апплет, наглядно демонстрирующий примеры выделения различных групп атомов в структуре 1ODH. | ||||||||||||||||||||||||
В Таблице 2 представлены итоги поисков ДНК-белковых контактов. С помощью выделения различных групп атомов в составе комплекса (аналогично представленным в апплете выше) были определены различного рода полярные и неполярные взаимодействия между атомами белка и нуклеиновой кислоты. При этом полярными считались атомы азота и кислорода, неполярными - атомы углерода, фосфора и серы. Под полярным контактом подразумевается ситуация, когда полярные атомы белка и ДНК расположены на расстоянии не более 3.5 Å, неполярные - не более 4.5 Å. Поиск контактов осуществлялся с помощью программы JMol, c помощью данного скрипта. | ||||||||||||||||||||||||
| ||||||||||||||||||||||||
Из таблицы 2 видно, что наибольшее количество контактов с белком образуют остатки фосфорной кислоты, несколько меньше остатки дезоксирибозы. В целом из этого можно сделать вывод, что во взаимодействии с белковой молекулой преимущественно вступает сахоро-фосфатный остов ДНК. При этом в данном случае молекулы азотистых основыний образовывали существенно меньше контактов, атомы, обращенные в сторону малой бороздки не образовывали их вообще. При этом четкого перевеса в количестве полярных или неполярных контактов не наблюдается. | ||||||||||||||||||||||||
ВИЗУАЛИЗАЦИЯ ДНК-БЕЛКОВЫХ КОНТАКТОВС помощью программы nucplot из пакета EMBOSS была полученна популярная схема, иллюстрирующая ДНК-белковые контакты для комплекса 1ODH (Рис.1). 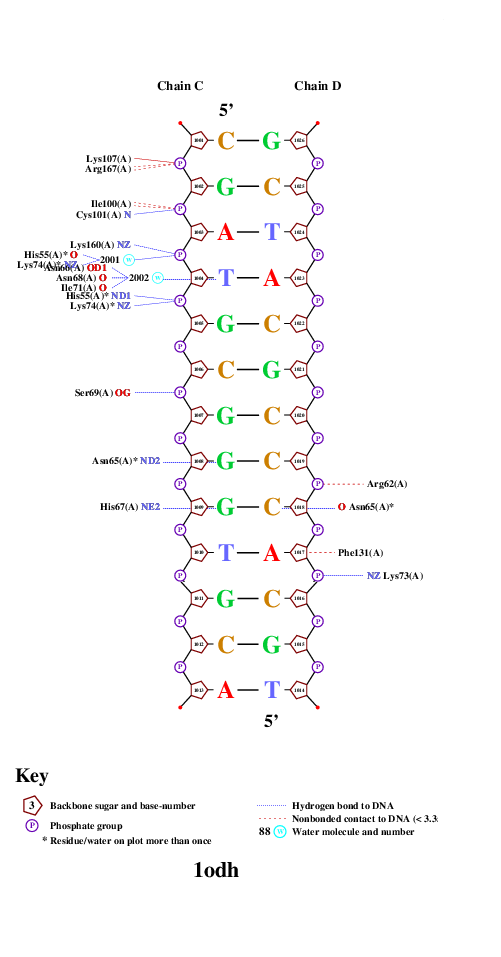 Рис.1 Схема ДНК-белковых контактов комплекса 1ODH Рис.1 Схема ДНК-белковых контактов комплекса 1ODH | ||||||||||||||||||||||||
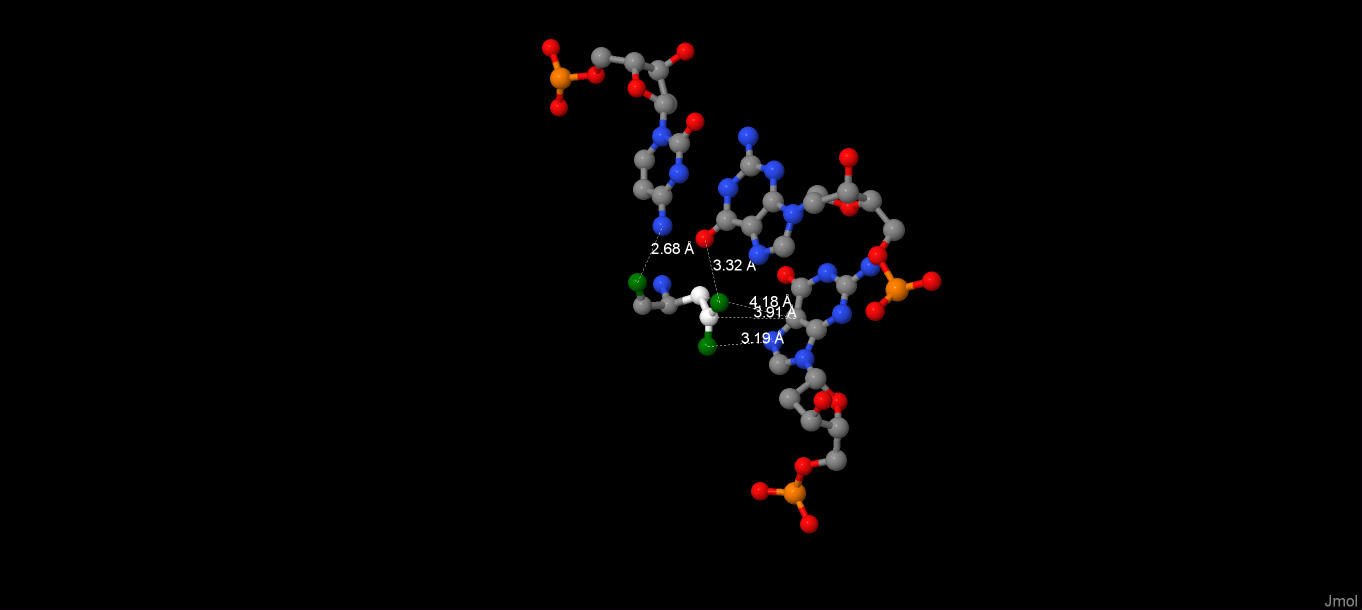
НЕКОТОРЫЕ КОНТАКТЫНа Рис. 2 показаны контакты аминокислоты [ARG]65 с ДНК. Как видно из схемы на Рис.1, данная аминокислота взаимодействует с двумя азотистыми основаними на двух различных цепочках ДНК. С помощтю Jmol ранее была выявлено, что данная аминокислоты вступает в пять взвимодейтсвий ( 3 полярных и 2 неполярных) с атомами малой бороздки. Однако испльзуя приложенный выше скрипт, были получены результаты, утверждающие, что данная аминокислота взаимодействует с тремя парами азотистых оснований (одно взаимодействие с [DC]1018, одно с [DG]1008 и три с [DG]1007). Из чего можно сделать вывод, что [ARG]65 вступает в наибольше число контактов. | ||||||||||||||||||||||||
Рис.2 Контакты [ARG]65 c ДНК Зеленым отмечены атомы аргинина, участвующие в полярных взаимодейтсвиях,
белым - в неполярных. | ||||||||||||||||||||||||
В узнавании ДНК, данным доменом (GCM) учавствует фрагмент 5'-ATGCGGGT-3' , при этом наиболее значимы позиции 2, 3, 6, 7. | ||||||||||||||||||||||||
Рис.3 Взаимодействие [HIS]67 c гуанином.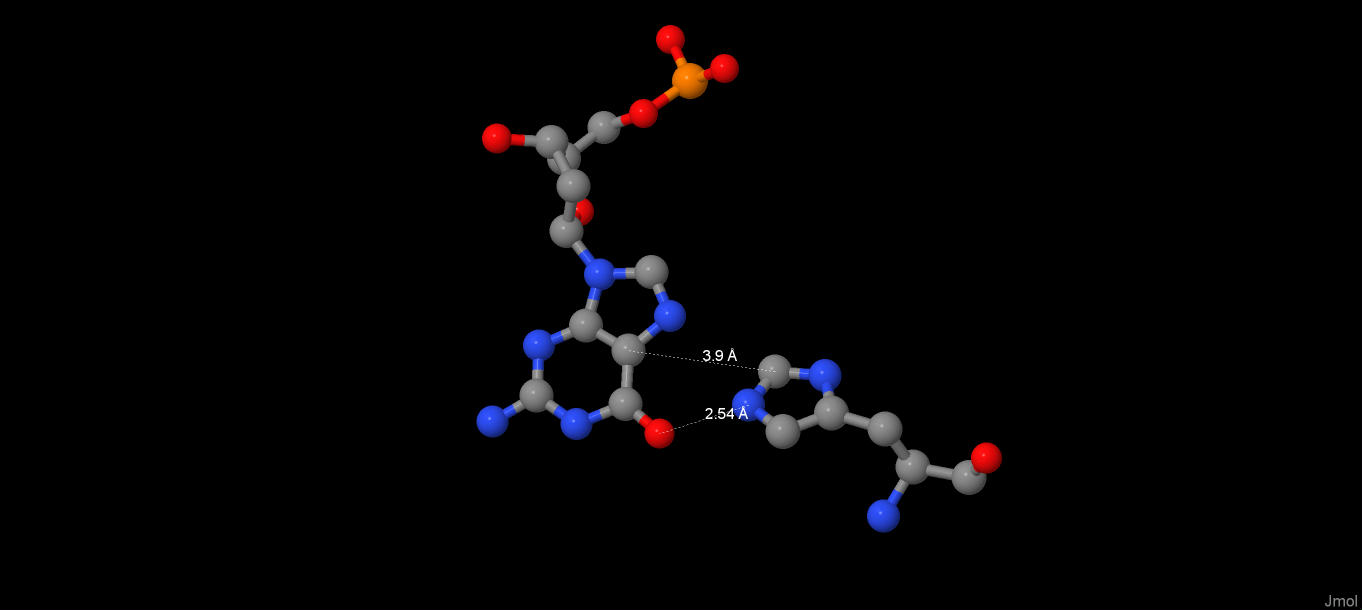 | ||||||||||||||||||||||||
ССЫЛКИ |