| A- И В- ФОРМЫ ДНК. СТРУКТУРА РНК. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
МОДЕЛИ СТРУКТУР ДНКНа данной страничке представлены структуры различных форм ДНК, построенных с помощью инструментов пакета 3DNA, одного из популярных пакетов программ для анализа и простейшего моделирования структур нуклеиновых кислот, работающего под операционной системой LINUX. С помощью программы fiber пакета 3DNA были построены A-, B- и Z-форму дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "gatc" Результат работы программы приложен в файлах ниже, а также отображен с помощью Jmol-апплетов. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Выход программы А-форма ДНК | Выход программы B-форма ДНК | Выход программы Z-форма ДНК | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 |  |  | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 |  | 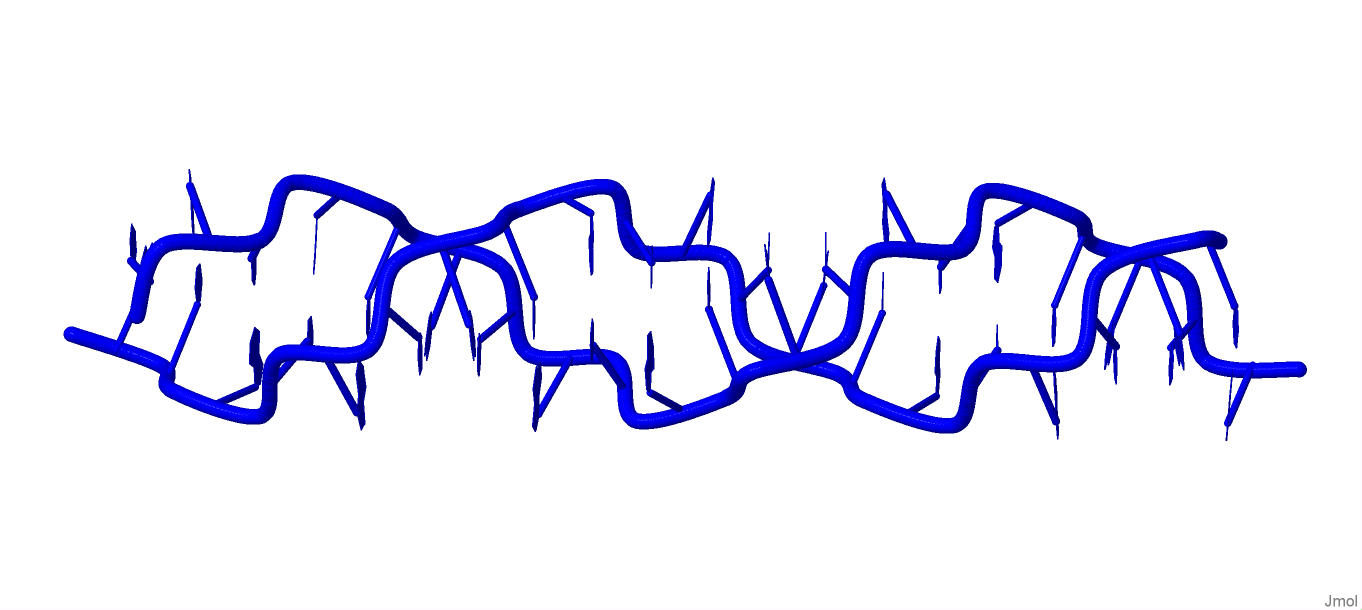 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
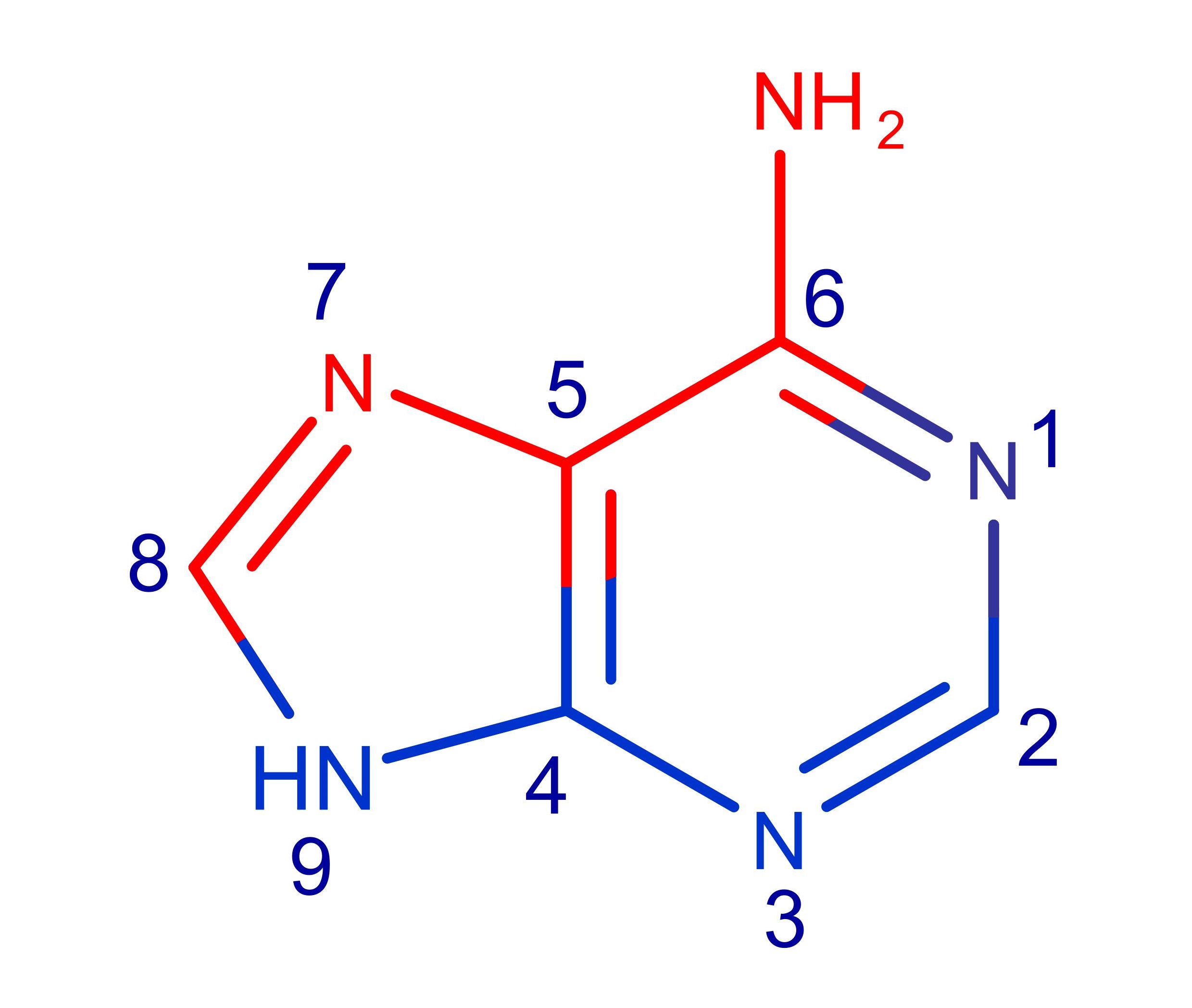
СРАВНЕНИЕ ФОРМ ДНК С ПОМОЩЬЮ JMOLC помощью программы Jmol удалось определить, какая из бороздок является малой и большой. Для аденина ([DA]10:A), входящего в состав структуры В-формы ДНК, полученной с помощью программы fiber (см. выше), было выявленно, какие из атомов этого азотистого основания смотрят в сторону большой бороздки (отмечены красным на Рис.1), а какие в сторону малой (отмечены синем на Рис.1). В результате получилось: | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Рис.1 Изображение азотистого основания аденина (справа). Синим цветом обозначены атомы, обращенные в сторону малой бороздки, красным цветом обозначены атомы, обращенные в сторону большой бороздки. |
 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Также с помощью программы Jmol, было проведено сравнение 3-х полученных форм ДНК по основным параметрам. Результаты сравнения предствлены в Таблице 1. Таблица 1. Сравнение 3-х форм ДНК.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
ОПРЕДЕЛЕНИЕ ПАРАМЕТРОВ НУКЛЕИНОВЫХ КИСЛОТС помощью пакеты 3DNA были определены различные параметры заданных нуклеиновых кислот (торсионные углы, структура водородных связей, стекинг взаимодейтвия). Были рассмотрены структура тРНК, взятой из комплекса аспартил-тРНК синтетазы (PDB ID:1IL2) и ДНК, из комплекса домена GCM, связанного с ДНК (PDB ID: 1ODH). Апплеты с данными комплексами представлены ниже. Cинем цветом окрашены нуклеиновые кислоты, черным белковая часть комплекса. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
ОПРЕДЕЛЕНИЕ ТОРИОННЫХ УГЛОВ НУКЛЕОТИДОВС помощью программ пакета 3DNA find_pair и analyze, были определены значения торсионных углов для данных тРНК и ДНК, а также структур А-, В-, Z-форм ДНК, полученных выше. С помощью функций СРЗНАЧ и ОКРУГЛ Мicrosoft Excel были посчитаны средние значения для каждого из углов, значения которых преведены в Таблице 2. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Таблица 2. Средние значения торсионных углов.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Как видно из Таблицы 2, усредненные значения торсионных углов обеих тРНК ближе по значению к А-форме ДНК, ДНК (1ODH) наоборот больше походит на В-форму. Также были определены нуклеотиды с наиболее отклоняющимися от среднего значениями торсионных углов. С помощтю Excel для каждого из нуклеотидов удалось посчитать отклонения от среднего значения угла для каждого из семи углов. Нуклеотиды с наибольшими значениями суммы отклонений от каждого угла были признаны самыми "деформированными". Cамые деформированные нуклеотиды тРНК:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Таблица 3. Наиболее деформированные нуклеотиды для strand1.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Таблица 4. Наиболее деформированные нуклеотиды для strand2.
Самые деформированныq нуклеотид ДНК: 2G | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Таблица 4. Наиболее деформированные нуклеотиды для ДНК.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Таблицас вычислениями | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
СТРУКТУРА ВОДОРОДНЫХ СВЯЗЕЙБлагодаря использованию программ find_pair и analyze были определены водородные связи, образующие стебли вторичной структуры тРНК. Ниже преведенны фрагменты документа, иллюстрирующие водородные связи между парами стеблей, наглядное изображение которых представлено на Рис. 1. Разными цветами окаршены разные стебли. Также можно ознакомится с документом. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Strand I Strand II Helix
1 (0.003) ....>C:.901_:[..U]U-----A[..A]:.972_:C<.... (0.005) |
2 (0.004) ....>C:.902_:[..C]C-----G[..G]:.971_:C<.... (0.009) |
3 (0.004) ....>C:.903_:[..C]C-----G[..G]:.970_:C<.... (0.011) |
STEM1 4 (0.012) ....>C:.904_:[..G]G-----C[..C]:.969_:C<.... (0.004) |
5 (0.008) ....>C:.905_:[..U]U-*---G[..G]:.968_:C<.... (0.010) |
6 (0.006) ....>C:.906_:[..G]G-----C[..C]:.967_:C<.... (0.008) |
7 (0.006) ....>C:.907_:[..A]A-----U[..U]:.966_:C<.... (0.004) |
8 (0.017) ....>C:.949_:[5MC]c-----G[..G]:.965_:C<.... (0.008) |
9 (0.005) ....>C:.950_:[..G]G-----C[..C]:.964_:C<.... (0.005) |
STEM2 10 (0.006) ....>C:.951_:[..G]G-----C[..C]:.963_:C<.... (0.006) |
11 (0.012) ....>C:.952_:[..G]G-----C[..C]:.962_:C<.... (0.005) |
12 (0.009) ....>C:.953_:[..G]G-----C[..C]:.961_:C<.... (0.008) |
15 (0.005) ....>C:.938_:[..C]C-**--P[PSU]:.932_:C<.... (0.044) |
16 (0.006) ....>C:.939_:[..G]G-----C[..C]:.931_:C<.... (0.006) |
17 (0.003) ....>C:.940_:[..U]U-*---G[..G]:.930_:C<.... (0.009) |
STEM3 18 (0.010) ....>C:.941_:[..G]G-----C[..C]:.929_:C<.... (0.004) |
19 (0.010) ....>C:.942_:[..C]C-----G[..G]:.928_:C<.... (0.004) |
20 (0.003) ....>C:.943_:[..C]C-----G[..G]:.927_:C<.... (0.005) |
21 (0.005) ....>C:.944_:[..A]A-**--G[..G]:.926_:C<.... (0.005) |
22 (0.009) ....>C:.910_:[..G]G-*---U[..U]:.925_:C<.... (0.004) |
STEM4 23 (0.003) ....>C:.911_:[..U]U-----A[..A]:.924_:C<.... (0.009) |
24 (0.007) ....>C:.912_:[..U]U-----A[..A]:.923_:C<.... (0.007) |
25 (0.044) ....>C:.913_:[PSU]P-*---G[..G]:.922_:C<.... (0.005) |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Рис.1 Стебли тРНК. В состав структуры данной тРНК входят также неканонические пары оснований. Ниже предствлены 9 из 30 существующих пар, являющихся неканоческими (U-G, P-G, C-P, A-G, A-A, U-u). | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
5 (0.008) ....>C:.905_:[..U]U-*---G[..G]:.968_:C<.... (0.010) | 14 (0.043) ....>C:.955_:[PSU]P-**+-G[..G]:.917_:C<.... (0.005) x 15 (0.005) ....>C:.938_:[..C]C-**--P[PSU]:.932_:C<.... (0.044) | 17 (0.003) ....>C:.940_:[..U]U-*---G[..G]:.930_:C<.... (0.009) | 21 (0.005) ....>C:.944_:[..A]A-**--G[..G]:.926_:C<.... (0.005) | 22 (0.009) ....>C:.910_:[..G]G-*---U[..U]:.925_:C<.... (0.004) | 25 (0.044) ....>C:.913_:[PSU]P-*---G[..G]:.922_:C<.... (0.005) | 27 (0.003) ....>C:.914_:[..A]A-**--A[..A]:.921_:C<.... (0.008) | 29 (0.115) ....>C:.916_:[H2U]u-**+-U[..U]:.959_:C<.... (0.006) x | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Помимо этого в состав данной структуры входят также пары оснований, поддурживающие третичную структуру молекулы и не учавствующие в образовании стеблей. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
14 (0.043) ....>C:.955_:[PSU]P-**+-G[..G]:.917_:C<.... (0.005) x 26 (0.003) ....>C:.908_:[..U]U-**--A[..A]:.946_:C<.... (0.005) | 29 (0.115) ....>C:.916_:[H2U]u-**+-U[..U]:.959_:C<.... (0.006) x 30 (0.021) ....>C:.918_:[..G]G-----C[..C]:.956_:C<.... (0.007) + | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Стеккинг-взаимодейтвияВ том же документе, выход программ find_pair и analyze были выявлены пары с наибольшей и наименьшей площадью перекрывания 2-х последовательных пар азотистых оснований (данные приведены ниже). С помощью программ ex_str и stack2img , были получены изображения стеккинг-взаимодействия для этих двух пар (Рис.2 и Рис.3). | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
41 Gt/AC 8.53( 3.99) 0.00( 0.00) 0.00( 0.00) 5.60( 2.62) 14.13( 6.61)
50 AG/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Рис. 2 Стеккинг взаимодействие с максимальным перекрыванием (пара 41)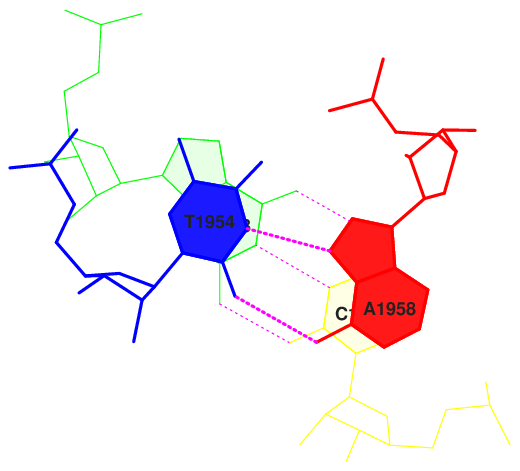 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Рис. 3 Стеккинг взаимодействие с минимальным перекрыванием (пара 50)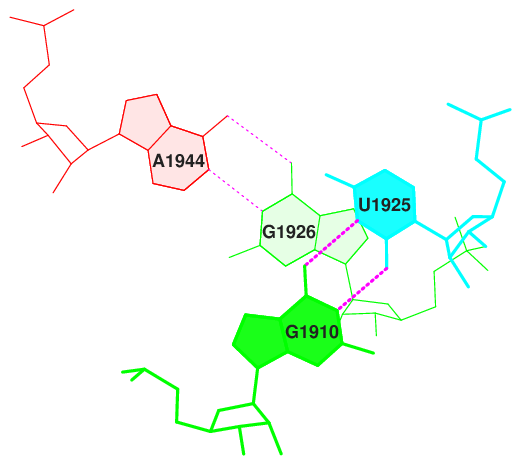 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
