КАЧЕСТВО СБОРКИ ГЕНОМА ЭУКАРИОТИЧЕСКОГО ОРГАНИЗМА |
| Для охарактеризования качетсво сборки генома эукариотического организма был выбран вид Шистосома Маносони (Schistosoma mansoni).
Шистосома Мансони - вид плоских червей, один из основных возбудителей шистосомоза человека. Паразитирует в брыжеечных венах около кишечника, возбуждая тем самым кишечный
шистосомоз. Данный вид обладает признаками, выделяющими шистосом из ряда других плоских червей: раздельнополостью, наличием в жизненном цикле свободноплавающией личинки
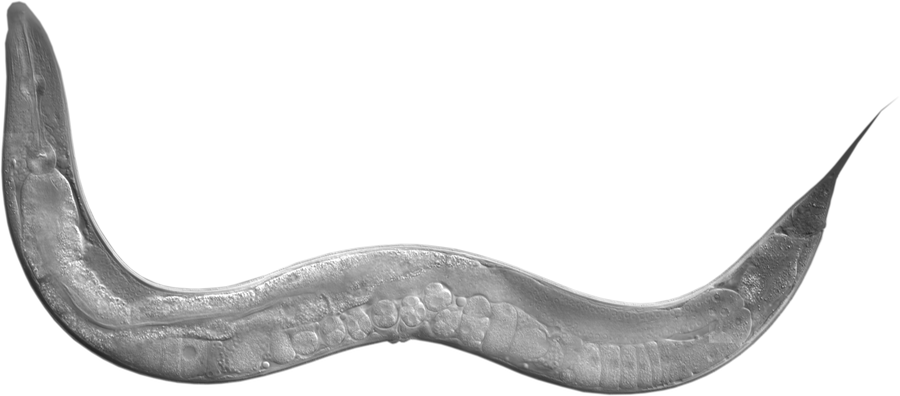
церкарии с характерным раздвоенным хвостом (Рис.1), способностью церкарии проникать в организм человека через кожу и слизистые оболочки, а не только при проглатывании (Рис.2).
Последним свойством шистосома обязана своему широкому распространению. Шистосомы Мансони обитают в водоемах стран Карибского бассейна,
Южной Америке, Африке, на Ближнем Востоке. По скромным подсчетам в 2015 году 218 млн. человек в мире нуждались в профилактическом лечении
шистосомоза. И примерно в половине этих случаев виновником являлась шистостома Мансони. Наибольшую опасность представляют шипы на яйцах шистосом.
Часть яиц с помощью шипа с легкостью проникает сквозь стенку кровеносных сосудов, попадают в кишечник и выходят с калом во
внешнюю среду. Остальные же попадают в кровоток и имеют свойство застревать в тканях хозяина – это и есть основной вред от шистосомза.
Так повреждается в первую очередь печень и стенки кишечника – развивается
фиброз.[1],[2],[3] |
Таблица 1. Таксономическое положение Schistosoma mansoni.[4]
| Superkingdom | Eukaryota |
| Kingdom | Metazoa |
| Phylum | Platyhelminthes |
| Class | Trematoda |
| Subclass | Digenea |
| Order | Strigeidida |
| Superfamily | Schistosomatoidea |
| Family | Schistosomatidae |
| Genus | Schistosoma |
Рис.1 Церкария шистосомы  Рис.2 Жизненный цикл шистосомы Рис.2 Жизненный цикл шистосомы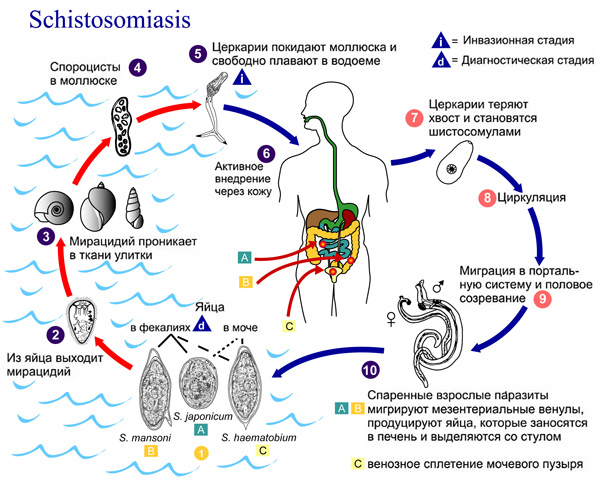 |
| Таким образом шистосома представляет из себя довольно интрересный объект для изучений, в связи с чем существует множество проектов по секвенированию
генома данного организма. |
ЧИСЛО СБОРОК, ПРОЕКТОВ И ОБРАЗЦОВ |
| BIOPROJECT | 82 |
| BIOSAMPLE | 1846 |
| ASSEMBLY | 2(1)* |
|
Существует 82 проекта, связанных с геномом Schistosoma mansoni, большинство их этих проектов направлены на изучение транскриптомов (54) и лишь
один на сборку генома (3 проекта об эпигеноме, 1 - экзом, 1 -секвенирование генома и др.). Из них лишь один попадает в категорию Refseq.
Образцов данного вида насчитывается 1846, число сборок - 1.
|
| *Поиск для сборок по данному организму выдает две записи, однако вторая по-видимому является более поздней,
проверенной версией первой, относящейся к Refseq, хоть и датированы они обе одним числом. |
ДАННЫЕ ОБ ОБРАЗЦЕ
В данном случае была выбрана одна из двух сборок (более поздняя). Ниже в таблице приведены характеристики образца, из которого был извлечен генетический
материал для сборки генома. |
Таблица 2. Характеристика биообразца Schistosoma mansoni
| Параметр | Значение | Пояснение |
| Identifiers | BioSample: SAMEA2272516; SRA: ERS379934 | Идентификатор образца |
| Organism | Schistosoma mansoni
cellular organisms; Eukaryota; Opisthokonta; Metazoa; Eumetazoa; Bilateria; Platyhelminthes; Trematoda; Digenea; Strigeidida; Schistosomatoidea; Schistosomatidae; Schistosoma | Таксономия образца |
| Attributes |
| sample name | ERS379934 |
strain | Puerto Rico |
ENA-FIRST-PUBLIC | 2013-12-19 |
ENA-LAST-UPDATE | 2013-12-09 |
| Идентификатор образца Название штама Дата первой публикации в ENA Дата послденего обновления |
| BioProjects |
PRJNA39937 Schistosoma mansoni
Retrieve all samples from this project
PRJEA36577 Schistosoma mansoni strain:Puerto Rico
Retrieve all samples from this project | Проекты с участием данного образца |
| Submission | EBI; 2013-12-20 | Организация и дата предоставления в общее пользованние |
|
ГЕНОМНЫЙ ПРОЕКТ |
| Выбранная сборка фигурирует сразу в двух проектах, один из которых относится к секвенированию и сборке генома, а другой к геному Refseq. Учитывая,
что параметры обоих проектов и фигурирующая информация о проектах довольна близка, делаю вывод, что одна из этих записей возникла после того, как геном
Schistosoma mansoni был добавлен в базу данных Refseq. Они не одинаковы, но близки. Оба посвящены исключительно геному данного вида.
Описание гласит: "кровяная трематода, шистосома Мансони, одна из трех трематод, ответственных за возникновение шистоматоза. Геном этого организма
состоит из 7 соматических хромасом и 2 половых."
Идентификаторы данных проектов PRJNA39937, PRJEA36577
Секвенирование проводилось с использованием Whole Genome Shotgun стратегии секвенирования. Около 3.8 миллионов ридов было произведено и собрано с
алгоритма сборки Phusion. Обоим проектам сопутствует одна публикация: Le TH et al., "Phylogenies inferred from mitochondrial gene orders-a cautionary tale from the parasitic flatworms.",
Mol Biol Evol, 2000 Jul;17(7):1123-5
|
ОПИСАНИЕ КЛЮЧЕЙ ТАБЛИЦ ОСОБЕННОСТЕЙ |
Банки нуклеотидных последовательностей EMBL, DDBJ, GenBank используют таблицы сайтов и особенностей для описания роли и местоположения
доменов последовательностей и их элементов более высокого порядка в пределах генома организма. Диапозон таблиц особенностей обширен и включает в себя регионы которые:
- Выполняют какую-либо биологическую функцию
- Влияют или явлеяются результатом выражения биологической функции
- Взаимодействуют с другими молекулами
- Влияют на репликацию последовательности
- Влияют на или являются результатом рекомбинации различных последовательностей
- Являются узнаваемой повторяющейся единицей
- Имеют вторичную или третичную структуру
- Демонстрируют вариабильность или были пересмотрены и исправлены
|
Структура таблиц особенностей состоит из следующих элементов:
- Feature key - слово или аббревиатура, обозначающая функциональную группу
- Location - инструкции по поиску данной особенности
- Qualifiers - вспомогательная информация об объекте
|
Существует множество ключей для таблиц особенностей, ниже приведены несколько примеров их использования:
CDS |
| ОПРЕДЕЛЕНИЕ: кодирующая последовательность; последовательность нуклеотидов, которые соответствует
последовательности аминокислот в белке (location включает стоп-кодон). |
| ПРИМЕР: (GenBank: AJ405982.1; Polyclad flatworm sp. THRL1 partial mitochondrial cytochrome oxidase c subunit I (coxI gene)) |
CDS <1..>555
/gene="coxI"
/codon_start=1
/transl_table=14
/product="cytochrome oxidase c subunit I"
/protein_id="CAC14006.1"
/db_xref="GOA:Q9G6X0"
/db_xref="InterPro:IPR000883"
/db_xref="UniProtKB/TrEMBL:Q9G6X0"
/translation="EVYILILPGFGMISQVVTFYSGKDSAFGHMGMVYAILGIGLLGF
IVWAHHMYTVGLDIDTRAYFTGATMIIAVPTGIKIFSWLATFYGRPLSQSVDSVGPAW
ATGFIFLFTLGGLTGVVLASASLDISLHDTYYVVAHFHYVLSMGAVFSIFAGLVHWWP
LFTGTGLNGKMAMGQFWVMFTGVNL" |
| ПОЯСНЕНИЕ: Данный фрагмент последовательности кодирует белок, называемый cytochrome oxidase c subunit I (ID: CAC14006.1),
имеющий следующие идентификаторы в различных базах данных: GOA:Q9G6X0, InterPro:IPR000883, UniProtKB/TrEMBL:Q9G6X0; кодирует указанную
выше последовательность аминокислот, входит в состав гена coxI, который кодирует данный белок. /codon_start указывает на смещение в
рамке считывания данной последовательности; /transl_table определяет используемую таблицу генетического кода в случае если универсальная таблица не
подходит для данной последовательности. При этом первый нуклеотид данного гена находится за пределами данного фрагмента, аналогично последнему (в
данной записи приведен лишь фрагмент всего гена). |
GENE |
| ОПРЕДЕЛЕНИЕ: область биологического интереса, определенная как ген, для которого присвоено собственное
название. |
| ПРИМЕР: (GenBank: AJ405982.1; Polyclad flatworm sp. THRL1 partial mitochondrial cytochrome oxidase c subunit I (coxI gene)) |
gene <1..>555
/gene="coxI" |
| ПОЯСНЕНИЕ: Данный фрагмент входит в состав гена, называемого "coxI".
При этом первый нуклеотид данного гена находится за пределами рассматриваемой последовательности, аналогично последнему (в
данной записи приведен лишь фрагмент всего гена). |
SOURCE |
| ОПРЕДЕЛЕНИЕ: Данный ключ идентифицирует биологический источник определенного участка рассматриваемой последовательности.
Этот ключ является обязательным. Возможно использование более одного ключа данного типа для каждой последовательности.
Каждая запись в любом случае имеет либо один ключ source, охватывающий всю последовательность, либо несколько source ключей, которые вместе,
охватывают последовательность целиком. |
| ПРИМЕР: (GenBank: MG387089.1 ; Schistosoma mansoni isolate H511DB6-parasite small subunit ribosomal RNA gene and internal transcribed spacer 1, partial sequence) |
source 1..485
/organism="Schistosoma mansoni"
/mol_type="genomic DNA"
/isolate="H511DB6-parasite"
/host="Biomphalaria smithi (snail)"
/db_xref="taxon:6183"
/country="Tanzania: Bugamba"
/collection_date="28-Sep-2010"
/collected_by="Jared Bakuza" |
| ПОЯСНЕНИЕ: Данный фрагмент принадлежит геномной ДНК вида Schistosoma mansoni,
обитавщей в хозяине Biomphalaria smithi (snail). Образец был получен в Танзании, Джаредом Бакуза 28 сентября 2010 года. Данный организм в базе данных taxon
имеет ID 6183. |
REPEAT_REGION |
| ОПРЕДЕЛЕНИЕ: Область генома, содержащая повторяющиеся элементы. |
| ПРИМЕР: (GenBank: AJ746373.1; Taraxacum officinale microsatellite DNA, clone msta145r |
repeat_region 1..136
/experiment="experimental evidence, no additional details
recorded"
/rpt_type=TANDEM
/rpt_unit_range=26..41
/rpt_unit_range=79..90
/satellite="microsatellite" |
| ПОЯСНЕНИЕ: Данный фрагмент содержит повторяющиеся участки по типу тандемных повторов, относящихся к микросателлитам (длиной меньше 9 нуклеотидов). Повторяющиеся фрагменты содержатся
на участке с 26 по 41 нуклеотид и с 79 по 90. |
PRIMER_BIND |
| ОПРЕДЕЛЕНИЕ: Нековалентный сайт связывания праймера для инициирования репликации, транскрипции или обратной транскрипции.
Включает в себя так же сайт(ы) для синтетических реакций, например, праймеров PCR. |
| ПРИМЕР: (GenBank: AJ746373.1; Taraxacum officinale microsatellite DNA, clone msta145r) |
primer_bind 1..19
primer_bind complement(116..136) |
| ПОЯСНЕНИЕ: Данный фрагмент содержит повторяющиеся участки по типу тандемных повторов, относящихся к микросателлитам (длиной меньше 9 нуклеотидов). Повторяющиеся фрагменты содержатся
на участке с 26 по 41 нуклеотид и с 79 по 90. |
REGULATORY |
| ОПРЕДЕЛЕНИЕ: Любая область последоватльности, учавствующая в регуляции транскрипции, трансляции, репликации
или в организации структуры хроматина. |
| ПРИМЕР: (GenBank: AY519853.1; Caenorhabditis elegans acetylcholine receptor (62.5 kD) (acr-23) complete mRNA) |
regulatory 1711..1716
/regulatory_class="polyA_signal_sequence"
/gene="acr-23"
/locus_tag="5E130"
/note="standard AATAAA" |
| ПОЯСНЕНИЕ: Последовательность с 1711 по 1716 позиции содержит
регуляторный учатсток, поли-А сигнальную последовательность, содержащую стандартный фрагмент AATAAA. Последовательность входит в состав гена acr-23. |
TRNA |
| ОПРЕДЕЛЕНИЕ: Участок кодирует зрелую транспортную РНК, небольшую молекулу РНК (длинной 75-85 оснований), которая
опосредует трансляцию последовательности нуклеиновой кислоты в аминокислотную последовательность. |
| ПРИМЕР: (NCBI Reference Sequence: NC_001328.1; Caenorhabditis elegans mitochondrion, complete genome) |
tRNA 5621..5677
/product="tRNA-Leu"
/codon_recognized="CUN" |
| ПОЯСНЕНИЕ: Последовательность с 5621 по 5677 позиции кодирует
тРНК, переносящую аминокислоту лейцин. Данная тРНК узнает антикодоном последовательность CUN. |
|
МАССОВЫЕ ГЕНОМНЫЕ ПРОЕКТЫ1000 FUNGAL GENOMES |
| Существует множество массовых геномных проектов, одним из которых является геномный проект 1000
fungal genomes(1KFG). Данный проект является международной коллаборацией и направлен он на восполнение пробела в нашем знании о разнообразии грибных
организмов. Грибы играют крупномасштабную роль в функционирвании экосистем, выступая в роли многочисленных симбионтов, паразитов и редуцетов, играя
основополагающую роль в углеродном цикле, обнаруживая способность разлагать большинство природных полимеров и большинство ангтопогенного происхождения.
Таким образом грибные организме в перспективе могут быть использованы в разработке биотоплива, биоремеедиации загрязненных экосистем и др. Однако для этого
необходимо понимание взаимодействия грибов в естественных и искусственных средах.
Всвязи с этим международная исследовательская группа во главе с DOE Joint genome institute приступила к проекту по упорядочиванию 1000 геномов со
всего Грибного дерева жизни. Общий план состоит в том, чтобы в течение 5 лет заполнить пробелы в Грибном древе Жизни путем секвенирования по
меньшей мере двух эталонных геномов из более чем 500 признанных семейств грибов. При этом этот проект имеет ключевую цель - предоставить справочную
информацию для исследования взаимодействий растений и микробов, микробной эмиссии и сбора парниковых газов.
Как уже было указано выше данный проект интернациональный, среди его основателей есть французские и нидерландские исследователи, однако большинство
участников граждане США.[5]
Как уже было описанно выше данный проект ставит себе целью за пять лет секвенировать по меньшей мере 1000 геномов грибных организмов, на данный момент
уже секвенировано более 800. Разработка плана проекта происходила в 2011 году. Первые упоминания о нем появляются в ноябре 2011. К декабрю 2012 было
было секвенировано более 50 геномов. Последний секвенированный образец:
2017-04-07 Phyllosticta capitalensis CBS 128856 v1.0[6] |
Таблица 4. Осписание проекта 1КFG
| Ссылка на сайт | http://1000.fungalgenomes.org/home/ |
| Страна | Интернациональный проект (большинство исследователей из США) |
| Организация | Международная исследовательская команда в коллаборации с DOE JGI |
| Планируемой число геномов | 1000+ |
| Число геномов на данный момент | более 800 |
| Год начала проекта | 2011 - планирование проекта 2012 - информация о первых секвенированных геномах |
| Последняя публикация | Информация о последнем секвенированном геноме: 2017-04-07 Phyllosticta capitalensis CBS 128856 v1.0[6] |
bgcolor = "d4b806" |
ТАБЛИЦА МИТОХОНДРИАЛЬНЫХ ГЕНОВ |
Для организма Caenorhabditis elegans, свободноживущего круглого червя (нематоды)длиной около 1 мм, популярного модельного
организма, были найдены все полные митохондриальные геномы. Поиск по запросу: |
| (Caenorhabditis elegans[Organism] AND Mitochondrion[Title]) AND (complete genome[Title] OR complete sequence[Title]) |
Выдает
две находки
Поиск в Refseq по запросу: |
| ((Caenorhabditis elegans[Organism] AND Mitochondrion[Title]) AND (complete genome[Title] OR complete sequence[Title])) AND Refseq[Keyworld] |
находит одну запись. В таблице 5 приведена информация о ней.
Также можно ознакомится с таблицей митохондриальных генов данного оргнизма, в которой представлена информация о белках, закодированных в митохондрионе,
их координатах и ориентации в геноме, их идентификаторов: |
| ТАБЛИЦА ГЕНОВ МИТОХОНДРИАЛЬНЫХ БЕЛКОВ |
Не удалось аналогичным образом найти информацию о митохондриальных генах организмов данного типа (Nematoda), а также для организмов и
таксонов Roundworm и Flatworm. Поэтому не удалось сравнить митохондриальные гены организмов различных таксонов. Таблица содержит
исключительно данные о генах, кодирующих белки. Количество генов, кодирующие разные типы молекул РНК было посчитано с помощью
записи в Refseq и описано в Таблице 5. |
 Рис.2 Жизненный цикл шистосомы
Рис.2 Жизненный цикл шистосомы