БАЗА ДАННЫХ OPM |
С помощью базы данных OPM был найден белок, содержащий в своей трансмембранной части альфа спирали
(Таблица 1) и белок, сожержащий в своей трансмембранной части бета листы (Таблица 2).
В качестве белка, содержащего только альфа-спирали был выбран Мускариновый ацетилхолиновый рецептор M4 человека. Он функционирует как ингибирующий ауторецептор для ацетилхолина.
Активация рецепторов M4 ингибирует выделение ацетилхолина в полосатом теле. Мускариновые ацетилхолиновые рецепторы обладают регулятивным действием на дофаминергическую нейротрансмиссию.[1] |
Таблица 1. Характеристика 5dsg (Muscarinic acetylcholine receptor M4).
| Название | Muscarinic acetylcholine receptor M4 |
| Идентификатор OPM | 5dsg |
| Организм | human |
| Толщинa гидрофобной части | 34.6 ± 2.1 A |
| Координаты трансмембранных спиралей | A - Tilt: 10° - Segments: 1( 28- 55), 2( 70- 95), 3( 100- 129), 4( 148- 174), 5( 195- 220), 6(401- 425), 7( 432- 456) |
| Среднее количество остатков в спирали | 22 |
| Мембрана | Эукариотическая плазматическая мембрана |
|
Рис.1. Структурная формкла мускаринового рецептора М4 (5dsg). 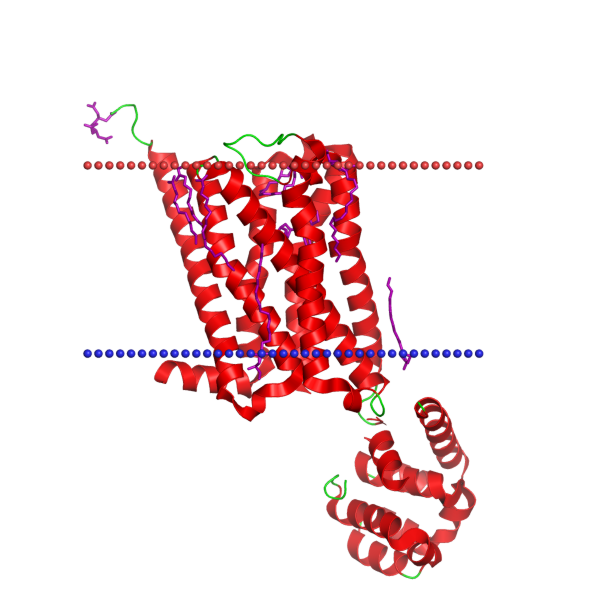 |
Рис.2. Структурная формкла мускаринового рецептора М4 (5dsg). 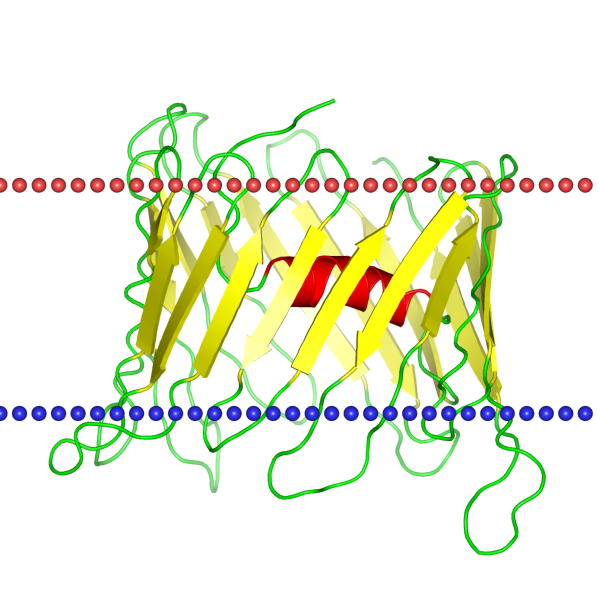 |
|
В качестве белка, содержащего в трансмембранной части только бета-листы был вабран потенциал-зависимый анионный канал, порин, расположенный на внешней
митохондриальной мембране человека. |
Таблица 2. Характеристика 2jk4 (VDAC-1 channel, structure 1).
| Название | VDAC-1 channel, structure 1 |
| Идентификатор OPM | 2jk4 |
| Организм | human |
| Толщинa гидрофобной части | 23.4 ± 2.3 A |
| Координаты трансмембранных спиралей | A - Tilt: 6° - Segments: 1(29-36), 2(42-51), 3(58-65), 4(72-79), 5(84-90), 6(98-105), 7(115-122), 8(126-134), 9(141-148), 10(152-160), 11(170-176), 12(182-187), 13(193-199), 14(206-213), 15(222-228), 16(235-241), 17(246-252), 18(258-266), 19(277-285) |
| Среднее количество остатков в спирали | 7 |
| Мембрана | Внешняя митохондриальная мембрана |
|
АНАЛИЗ ПРЕДСКАЗАНИЯ ТРАНСМЕМБРАННЫХ СПИРАЛЕЙ |
| Последовательность белка мусакринового рецептора (5dsg) была дана на вход серивисам TMHMM и Phobius. Результаты работы программ приведены ниже. |
| TMHMM | Phobius |
Рис.3. График результата работы TMHMM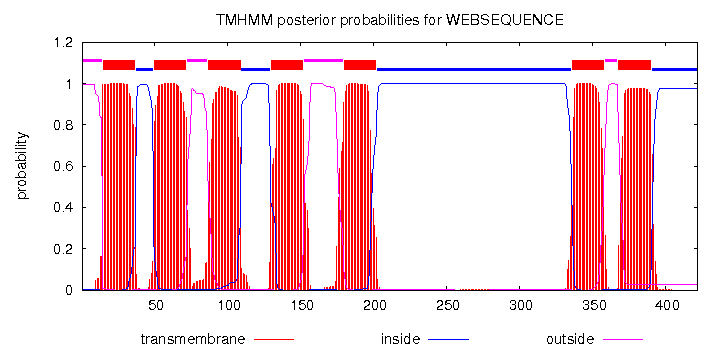 |
Рис.4 График результата работы Phobius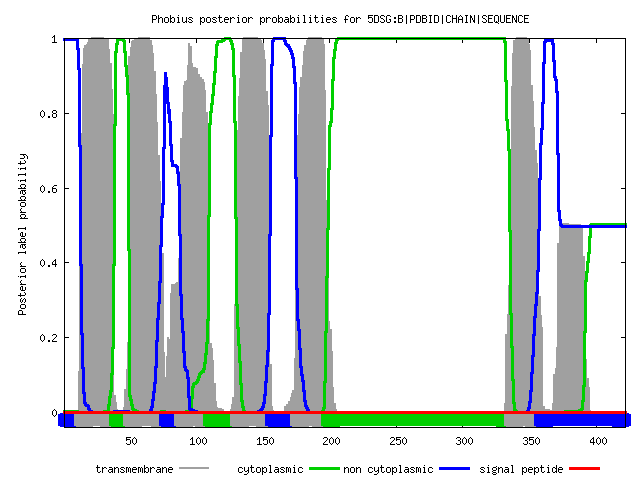 |
| По оси абсцисс отложены координаты аминокислотных остатков белковой последовательности, по оси ординат - вероятность того,
что в данном месте есть трансмембранная спираль. Линии разных ветов соответсвуют: красные - трансмембранные участки, синии - выходящие в цитоплазму,
розовые - выходящие наружу от цитоплазмы. |
По оси абсцисс отложены координаты аминокислотных остатков белковой последовательности, по оси ординат - вероятность того,
что в данном месте есть трансмембранная спираль. Линии разных ветов соответсвуют: серые - трансмембранные участки, зеленые - выходящие в цитоплазму,
синие - не присутсвующие в цитоплазме, красные - сигнальная последовательность |
Текстовая выдача TMHMM
outside 1 14
TMhelix 15 37
inside 38 49
TMhelix 50 72
outside 73 86
TMhelix 87 109
inside 110 129
TMhelix 130 152
outside 153 179
TMhelix 180 202
inside 203 335
TMhelix 336 358
outside 359 367
TMhelix 368 390
inside 391 422 |
Текстовая выдача Phobius
TOPO_DOM 1 12 NON CYTOPLASMIC.
TRANSMEM 13 38
TOPO_DOM 39 49 CYTOPLASMIC.
TRANSMEM 50 76
TOPO_DOM 77 87 NON CYTOPLASMIC.
TRANSMEM 88 109
TOPO_DOM 110 129 CYTOPLASMIC.
TRANSMEM 130 155
TOPO_DOM 156 174 NON CYTOPLASMIC.
TRANSMEM 175 197
TOPO_DOM 198 335 CYTOPLASMIC.
TRANSMEM 336 357
TOPO_DOM 358 422 NON CYTOPLASMIC. |
|
Из приведенных выше графиков видно, что сервером TMHMM были найдены все 7 спиралей, присутствующие в OPM, однако координаты не совпадают
с координатами из OPM, сдвиг наблюдается где-то на 20 аминокислот. Сервером Phobius были найдены лишь 6 спиралецй (с 1 по 6). Седьмой пик присутствует
на графике, однако его вероятность около 0.5. Координаты спиралей не совпадают с координатами с OPM, однако близки к координатам, предсказанным TMHMM
за исключением последней спирали. |
БАЗА ДАННЫХ TCDB |
В базе данных TCDB не было обнаружено белка мускоринового рецептора M4 человека. Наиболее похожий из обнаруженных белков был мускариновый
рецептор M1 мыши.Его идентификатор в этой базе данных 9.A.14.3.2.
- 9. - Incompletely Characterized Transport Systems
- 9.A. - Transporters of unknown biochemical mechanism
- 9.A.14. - The G-protein-coupled receptor (GPCR) Family
- 9.A.14.3.2. - Muscarinic acetylcholine receptor M1
|
Данный белок можно отнести к следующим терминам GO:
- GO:0030054 - Cell junction
- GO:0016021 - Integral to membrane
- GO:0045211 - Postsynaptic membrane
- GO:0040012 - Regulation of locomotion
- GO:0005886 - Plasma membrane
- GO:0016907 - G-protein coupled acetylcholine receptor activity
|
Белок учавствует в следующих метаболических путях KEGG: - mmu04020 Calcium signaling pathway
- mmu04024 cAMP signaling pathway
- mmu04080 Neuroactive ligand-receptor interaction
- mmu04151 PI3K-Akt signaling pathway
- mmu04725 Cholinergic synapse
- mmu04810 Regulation of actin cytoskeleton
|
CCЫЛКИ |
- https://en.wikipedia.org/wiki/Muscarinic_acetylcholine_receptor_M4
|
|
Главнaя страница
|



