ВЫРАВНИВАНИЕ ГЕНОМОВ
АНАЛИЗ КРУПНЫХ ГЕНОМНЫХ ПЕРЕСТРОЕК |
В данном практикуме рассматривались геномы бактерии Streptococcus thermophilus.Streptococcus thermophilus - вид грамположительных
факультативно анаэробных бактерий. Streptococcus thermophilus относится к группе молочнокислых бактерий, сбраживающих углеводы с
образованием молочной кислоты. Благодаря этому свойству он широко используется в пищевой промышленности при приготовлении различных молочных
продуктов, включая ряженку, йогурты, варенцы, сметаны, моцареллы и других сыров. |
В Таблице 1 приведена основная информация о трех геномах Streptococcus thermophilus, исполльзовавшихся в потсроении выравнивания. |
Таблица 1. Информация об используемых геномах Streptococcus thermophilus.
| ID | Штамм | Длина генома | Тип |
| CP000024 | CNRZ1066 | 1796226 bp | кольцевой |
| CP022547 | B59671 | 1821173 bp | кольцевой |
| CP012588 | MN-BM-A01 | 1876516 bp | кольцевой |
|
Данные хромосомы были выравнены при помощи blast2seq, алгоритма blastn. Параметры задавались по умолчанию. Ниже представлены
карты локального сходтва для трех выравниваний геномов: |
Рис.1 Карта локального сходства геномов CP000024.1 и CP022547.1 |
Рис.2 Карта локального сходства геномов СP012588.1 и CP000024.1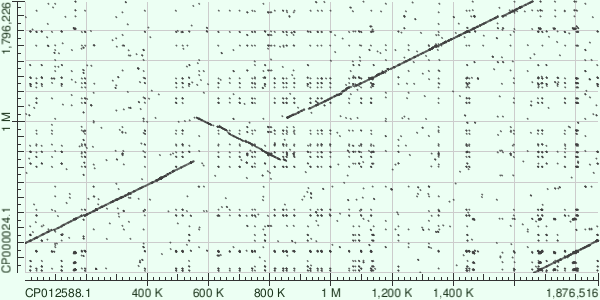 |
Рис.3 Карта локального сходства геномов СP012588.1 и СP022547.1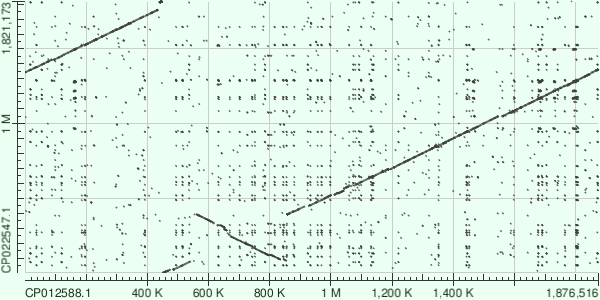 |
Ниже в Таблице 2 представлена основаная информация о трех построенных выравниваниях: |
Таблица 2. Характеристика выравниваний
| Выравнивание | CP012588.1-CP000024.1 | СP012588.1 - СP022547.1 | CP000024.1 - CP022547 |
| Ident% | 99% | 99% | 99% |
| E-value | 0.0 | 0.0 | 0.0 |
| Max score | 1.821e+05 | 1.982e+05 | 2.014e+05 |
| Query cover | 93% | 91% | 94% |
| Чиссло находок | 3285 | 3508 | 2965 |
| Карта |  |  |  |
| Наибольшая находка |
| Длина | 102490 | 111975 | 114287 |
| Ident% | 99.420 | 99.296 | 99.113 |
| E-value | 0.0 | 0.0 | 0.0 |
| Max score | 1.821e+05 | 1.982e+05 | 2.014e+05 |
| q. start - q. end | 1662107 - 1764548 | 1599495 - 1711426 | 501605-615811 |
|
Также в таблицах 3-5 можно ознакомится с находками для выравниваний, обладающих наибольшей длинной: |
Таблица 3. Оновные находки для выравнивания CP012588.1 и CP000024.1
| % identity | alignment length | mismatches | gap opens | q. start | q. end | s. start | s. end | evalue | bit score |
| 99.420 | 102490 | 531 | 29 | 1662107 | 1764548 | 1 | 102475 | 0.0 | 1.821e+05 |
| 99.141 | 84285 | 661 | 29 | 1238679 | 1322935 | 1390743 | 1474992 | 0.0 | 1.487e+05 |
| 99.052 | 72030 | 640 | 29 | 319179 | 391179 | 501800 | 573815 | 0.0 | 1.267e+05 |
| 99.070 | 58595 | 438 | 22 | 1488070 | 1546639 | 1635030 | 1693542 | 0.0 | 1.032e+05 |
| 97.614 | 53429 | 989 | 117 | 392577 | 445848 | 573816 | 627115 | 0.0 | 90335 |
| 99.282 | 48877 | 310 | 21 | 494787 | 543645 | 680952 | 729805 | 0.0 | 86503 |
| 99.262 | 47940 | 321 | 22 | 251354 | 299269 | 436708 | 484638 | 0.0 | 84788 |
| 98.637 | 46426 | 507 | 39 | 786923 | 833237 | 796504 | 750094 | 0.0 | 80808 |
| 99.190 | 44428 | 304 | 25 | 1805450 | 1849852 | 144379 | 188775 | 0.0 | 78435 |
| 99.412 | 44066 | 191 | 15 | 1194639 | 1238686 | 1346661 | 1390676 | 0.0 | 78294 | |
Таблица 4. Оновные находки для выравнивания CP012588.1 и CP022547.1
| % identity | alignment length | mismatches | gap opens | q. start | q. end | s. start | s. end | evalue | bit score |
| 99.296 | 111975 | 704 | 53 | 1599495 | 1711426 | 1086390 | 1198323 | 0.0 | 1.982e+05 |
| 98.986 | 105317 | 874 | 72 | 194051 | 299269 | 1524726 | 1629946 | 0.0 | 1.850e+05 |
| 99.042 | 74748 | 608 | 40 | 1255970 | 1330671 | 757912 | 832597 | 0.0 | 1.315e+05 |
| 98.998 | 72082 | 620 | 35 | 319179 | 391179 | 1648263 | 1720323 | 0.0 | 1.267e+05 |
| 99.565 | 64758 | 259 | 19 | 121071 | 185815 | 1455489 | 1520236 | 0.0 | 1.154e+05 |
| 99.229 | 54184 | 344 | 19 | 1492473 | 1546639 | 994361 | 1048487 | 0.0 | 95810 |
| 99.520 | 52865 | 208 | 19 | 1711706 | 1764548 | 1199824 | 1252664 | 0.0 | 94146 |
| 99.160 | 50566 | 371 | 16 | 1135852 | 1186374 | 644501 | 695055 | 0.0 | 89250 |
| 99.316 | 50014 | 275 | 21 | 38269 | 88260 | 1378555 | 1428523 | 0.0 | 88618 |
| 98.467 | 50096 | 665 | 35 | 562336 | 612391 | 391324 | 341292 | 0.0 | 86815 | |
Таблица 4. Оновные находки для выравнивания CP012588.1 и CP022547.1
| % identity | alignment length | mismatches | gap opens | q. start | q. end | s. start | s. end | evalue | bit score |
| 99.113 | 114287 | 906 | 41 | 501605 | 615811 | 1648069 | 1762327 | 0.0 | 2.014e+05 |
| 99.219 | 112274 | 802 | 37 | 114220 | 226441 | 1260580 | 1372830 | 0.0 | 1.984e+05 |
| 99.435 | 78922 | 401 | 14 | 1639432 | 1718322 | 994361 | 1073268 | 0.0 | 1.403e+05 |
| 99.510 | 73118 | 311 | 17 | 1060915 | 1133997 | 425946 | 499051 | 0.0 | 1.302e+05 |
| 99.442 | 72101 | 348 | 31 | 987610 | 1059694 | 353903 | 425965 | 0.0 | 1.281e+05 |
| 99.470 | 67589 | 337 | 13 | 242255 | 309831 | 1386105 | 1453684 | 0.0 | 1.202e+05 |
| 99.292 | 66977 | 405 | 26 | 1408044 | 1474992 | 757912 | 824847 | 0.0 | 1.186e+05 |
| 99.492 | 66397 | 280 | 26 | 1568652 | 1635034 | 921669 | 988022 | 0.0 | 1.182e+05 |
| 99.459 | 60306 | 295 | 17 | 436708 | 497004 | 1582024 | 1642307 | 0.0 | 1.072e+05 |
| 99.424 | 59343 | 286 | 17 | 49632 | 108955 | 1199824 | 1259129 | 0.0 | 1.054e+05 | |
АНАЛИЗ КРУПНЫХ ГЕНОМНЫХ ПЕРЕСТРОЕК |
Рис.1 Карта локального сходства геномов CP000024.1 и CP022547.1 |
На данной карте по горизонтали расположен геном CP000024.1, по вертикали CP022547.1. Перестройка, изображенная на карте, соответствует транслокации участка длинной
приблизительно в 600 тыс пн. (около трети генома) из начала генома в конец. Однако т.к. рассматриваемые геномы - кольцевые, следовательно участи начала и конца
геномов на картах совпадают друг с другом, а значит перестройки не происходило. Видимо, точки начала секвенирования для двух геномов не совпали. |
| На карте по горизонтали расположени геном СP012588.1, по вертикали CP000024.1. Трансолокация начального участка генома CP000024.1.
объясняется аналогично предыдущему случаю. Помимо этого на карте можно наблюдать инверисию крупного участка приблизительно с 550 тыс. нуклеотида по 850 тыс.
(по горизонтали). |
Рис.2 Карта локального сходства геномов СP012588.1 и CP000024.1 |
Рис.3 Карта локального сходства геномов СP012588.1 и СP022547.1 |
По горизонтали расположен геном СP012588.1, по вертикали СP022547.1. Транслокация объясняется аналогично первому случаю. Также на карте присутствует уже
виденная ранее инверсия - инвертированный участок приблизительно такой же длинный что и рассмотренный на второй карте. Больше крупных
перестроек на карте не наблюдается. |
|
Таким образом, основываясь на картах локального сходства, можно сделать вывод, что геномы CP000024.1 и CP022547.1 достаточно
близки друг к другу, между ними не происходило крупных перестроек. А геном СP012588.1, наоборот, отличает от обоих крупная инверсия участка длиной
приблизительно в 300 тыс. пн. Таким образом, можно предположить, что штамм MN-BM-A01 эволюционно более новый. |
|
|
|
|
|
|
|
|
|
|
Главнaя страница
|


