Метод секвенирования по Сэнгеру был разработан Фредериком Сэнгером в 1977г. На сегодняшний день секвенирование ДНК по Сэнгеру полностью автоматизировано и проводится нa секвенаторах. Использование дидезоксинуклеотидов с флуоресцентными метками с разными длинами волн испускания позволяет проводить реакцию в одной пробирке. Реакционную смесь разделяют капиллярным электрофорезом в растворе, фрагменты ДНК, выходящие из капиллярной колонки регистрируются детектором флуоресценции. Результаты анализируют с помощью компьютера и представляют в виде последовательности разноцветных пиков, соответствующих четырём нуклеотидам - хроматограмм.[1] | ||||||||||||||||
В рамках выполнения данной работы необходимо было прочитать последовательность ДНК на основании данных, полученных из капиллярного секвенатора по Сэнгеру и осветить проблемы, возникшие при чтении хроматограмм. Капилярный секвенатор выдает файл с автоматически прочтенной последовательностью в формате .ab1. При этом иногда у программы возникают проблемы с прочетинием последовательности в неоднозначных местах, где затруднено распознавание пиков, что ведет к возникновению ошибок или к появлению в последовательности буквы N, означающей любой из 4 возможных нуклеотидов. | ||||||||||||||||
| В данном случае чтение последовательности производилось с помощью программы Chromas lite. Чтение производилось с двух файлов в формате .ab1, соответствующих прямой и обратной цепям одной молекулы ДНК: | ||||||||||||||||
ИСХОДНЫЕ ФАЙЛЫ | ||||||||||||||||
ХАРАКТЕРИСТИКА ИСХОДНОЙ ХРОМАТОГРАММЫ | ||||||||||||||||
| Качетсво хроматограммы довольно сильно варьирует на разных участках, так как секвенированные фрагменты довольно крупные по размеру (около 1000 нуклеотидов). На удивление существенно более высоким качетсвом обладала обратная цепь. С помощью программы Chromas, она была реверсирована (был совершен переход к комплементарной цепочке) - в дальнейшем нуклеотиды нумеруются отностиельно реверсированной последовательности. Участок же на прямой цепи с хорошим сигналом очень невелик (см. Таблицу 1), поэтому довольно крупная часть прямой последовательности восстанавливалась по обратной цепи. На прямой цепи хороший участок с началом последовательности, на обратной - середина и конец. Поэтому при формировании итоговой последовательности из прямой цепочти брался участок с 1 по 289 (не считая первых 16 вырезаных), а 299-914 с обратной цепи (нуклеотиды считаются по файлу итоговой последовательности - см.ниже). В Таблицах 1 и 2 приведены более подробные данные по качетву хроматограмм. | ||||||||||||||||
| ||||||||||||||||
Таблица 2. Характеристика исходной хроматограммы обратной цепи при переходе к обратной цепочке(Ae6_18SII_R_H06_WSBS-Seq-1-08-15.ab1). | ||||||||||||||||
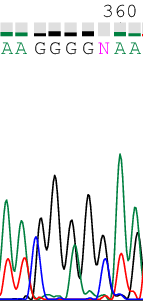
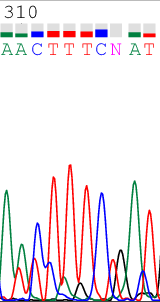
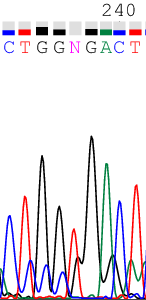
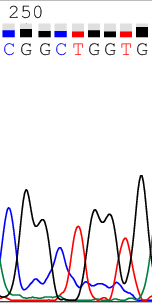
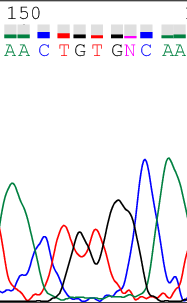
ПРОБЛЕМНЫЕ НУКЛЕОТИДЫВ ходе анализа хроматограмм возникали проблемы с трактованием последовательности - неоднозначные пики или расхождение во мнениях с программой. Ниже приведены некоторые примеры подобных ситуаций и принятые по ним решения. | ||||||||||||||||
| ||||||||||||||||
ПРОЕКТ ВЫРАВНИВАНИЯ ПРЯМОЙ И ОБРАТНОЙ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | ||||||||||||||||
| После прочтения хроматограмм были получены последовательности прямой и обратной(реверсированной цепи) в FASTA формате:
Проблемные нуклеотиды, которые были исправлены вручную помечены строчными буквами. Нечитаемые фрагменты последовательностей удалены (1-16; 750-905 для прямой цепи, 1-89; 950-962 для обратной цепи). С помощью программы needle из пакета EMBOSS было получено глобальное парное выравнивание двух последовательностей, визуализированное в Jalview. С файлами, полученными на выходе этих двух программ, а также с визуализированным выравниванием можно ознакомиться ниже: | ||||||||||||||||
 Глобальное парное выравнивание прямой и обратной последовательностей. | ||||||||||||||||
РЕЗУЛЬТАТ ПРОЧТЕНИЯКомбинируя две полученные последовательности прямой и обратной цепи был получена финальная последовательность. Учитывая особенности обеих хроматограмм, описанные выше, я добавила в финальную последовательность лучшие из двух вариантов фрагменты при наличии такового выбора. Таким образом 1-298 нуклеотид последовательности был взят из прямой цепи, 299-914 из обратной. | ||||||||||||||||
РЕЗУЛЬТАТ ПРОЧТЕНИЯ: | ||||||||||||||||
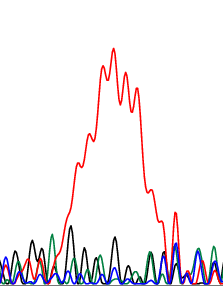
ПРИМЕР ПЛОХОЙ ХРОМАТОГРАММЫ
| ||||||||||||||||
| Главнaя страница |

.png)

.png)
.png)

.png)