ВЫРАВНИВАНИЕ КАК ОТРАЖЕНИЕ ЭВОЛЮЦИИ |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
ПОСТРОЕНИЕ ВЫРАВНИВАНИЯ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ГОМОЛОГИЧНЫХ БЕЛКОВ | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
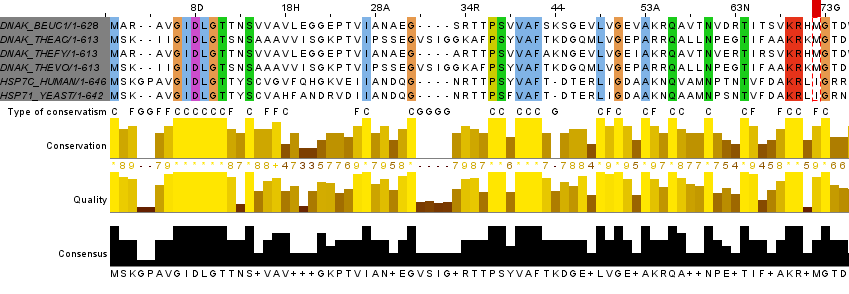
| В данной работы было пострено выравнивание последовательностей для шести гомологичных белков из семейства HSP70 (два из эукариот (PDB ID HSP7C_HUMAN_1; HSP71_YEAST_1), два из бактерий (PDB ID: DNAK_THEFY_1; DNAK_THEVO_1), два из архей (PDB ID: DNAK_BEUC1_1: DNAK_THEAC_1). Выравнивание было потстроено с помощью программы Jalview. Ниже приведен фрагмент результата ее работы (Рис. 1). | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Рис.1 Выравнивание для шести последовательностей гомологичных белков
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
На Рис.1 одинаковыми цветами показаны абслютно консервативные позиции (колонки). Под выравнием на Рис.1 добавлена строка с обозначением типа консерватизма для различных позиций, где "C" - позиции, консервативные более чем на 80%, "G" - позиции, содержащие гэп, "F" - абсолютно функциональные позиции (позиции, содержащие аминокислотные остатки из одних функциональных групп). В данном случае пометки "F" были присвоены нескольким позициям, в том числе позиции 3, где расположены аналогичные по свойствам положительно заряженные аминокислотные остатки аргенина и лизина (аналогично позиции 61); в позициях 6 и 7 чередуются алифатические аминокислоты аланин, валин и изолейцин (аналогично в позициях 56, 72, 75) и т.д.. В таблице 1 содержатся информация о выравнии. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Таблица 1. Информация о выравнивании.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
В таблице 1 приведена информация о длинах последовательностей и длине выравнивания, числе гэпов, числе абсолютно и функционально консервативных позиций, числе позиций, консервативных на 70%, и их процент от длины. Для рассчетов результатов из таблицы была использована программа infoalign пакета EMBOSS. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
ЭВОЛЮЦИЯ БЕЛКА | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
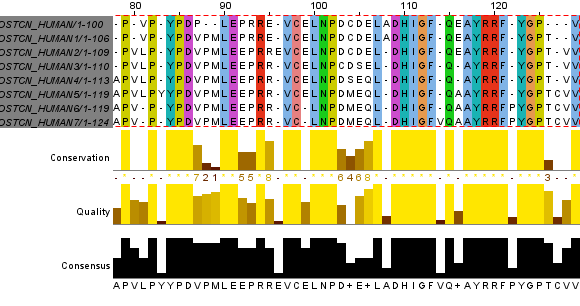
Эволюция белка изучалась на примере белка остеокальцина человека (Uniprot ID: OSTCN_HUMAN; составляет 1-2% от всех белков кости, прочно связывается с кальцием в костях), взятым из базы данных Uniprot. Длина данного белка 100 а.к.. При помощи команды msbar пакета EMBOSS было проведено семь раундов мутаций различных типов (делеции, вставки, замены, дупликации, перемешения). При этом исходная аминокислотная последовательность белка остеокальцина человека выступает в роли предка, а каждая последующая, полученная при помощи msbar последовательность - потомок, в котором за условно взятую единицу времени (100 лет) произошло 7 новых мутации. В дальнейшем поледовательности именуются p - предковая, p1, p2, p3... - полученные последовательным мутагенезом. Ниже можно ознакомится со сценарием, при использовании которого были получены все последовательности и полученным на его выходе файле.* На Рис.2 представлен результат выравнивания, предложенного Jalview для данных последовательностей. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Рис.2 Выравнивание восьми поколений последовательностей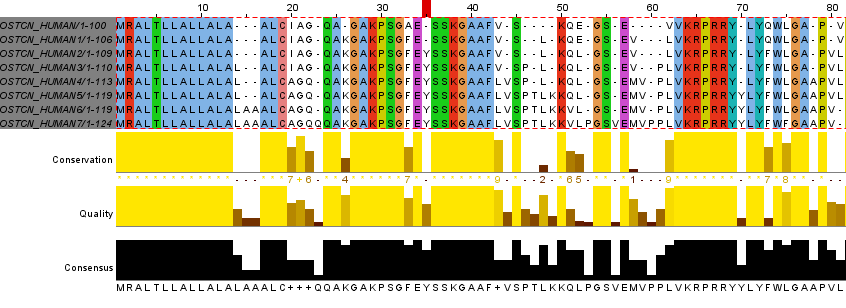 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
ИСПРАВЛЕННОЕ ВЫРАВНИВНИЕ | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Не всегда выравнивания построенные автомататически, по заданным параметрам абслолютно верны. Некоторые могут содержать очевидные огрехи. В данном случае выравнивание, полученное в Jalview было несколько изменено, с целью убрать очевидные ошибки. Исправленное выравнивание продемонстрировано на Рис.3. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Рис.3 Исправленное выравнивание восьми поколений последовательностей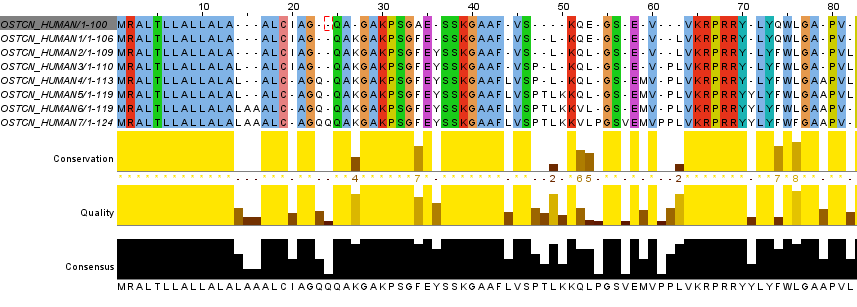 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| *Сценарий получения семи поколений последовательности, каждое из которых получает по семь новых точечных мутаций. Файл, содержащий получившиеся после мутирования последовательности, используемые в дальнейшем для выравнивания. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
ЭВОЛЮЦИЯ НУКЛЕОТИДНОЙ ПОСЛЕДОВАТЕЛЬНОСТИ | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Для изучения того, как влияет изменение нуклеотидной последоваветльности на эволюцию белка, был взят белок ацетил-носитель (UniProt ID: ACP_BACSU)из протеома Bacillus subtilis (strain 168), изучавшийся в более ранней работе. Этот белок участвует в пути биосинтеза жирных кислот, являющимся частью липидного метаболизма. Длина данного белка 77 а.к.. Аналогично предыдущему экспериметну было проведено семь раундов мутаций различных типов (делеции, вставки, замены, дупликации, перемешения), однако на этот раз нуклеотидной последовательности. С помощью программы transeq из мутированных нуклеотидных последовательностей были получены белковые последовательности продуктов трансляций. Аналогично в дальнейшем поледовательности именуются p - предковая, p1, p2, p3... - полученные последовательным мутагенезом. Ниже можно ознакомится со сценарием, при использовании которого были получены все последовательности и полученным на его выходе файле, а также с исходной нуклеотидной последовательностью* На Рис.4 представлен результат выравнивания, предложенного Jalview для данных последовательностей. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Рис.4 Выравнивание восьми поколений последовательностей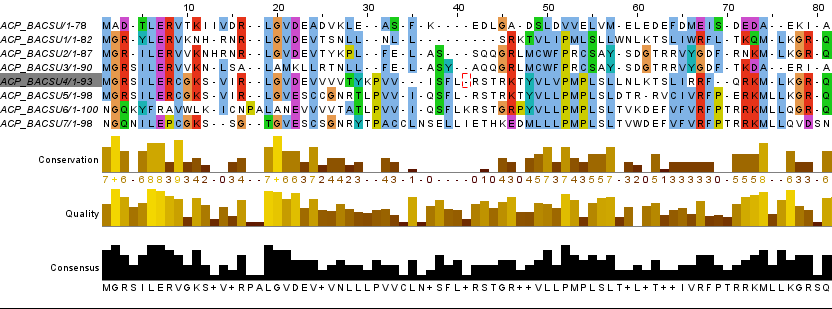 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
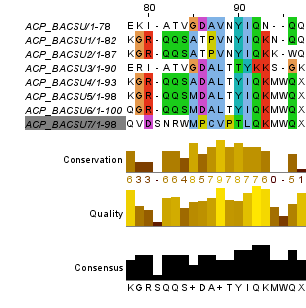
ИСПРАВЛЕННОЕ ВЫРАВНИВНИЕ | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Рис.5 Исправленное выравнивание восьми поколений последовательностей | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| *Сценарий получения семи поколений последовательности, каждое из которых получает по семь новых точечных мутаций. Файл, содержащий получившиеся после мутирования последовательности, используемые в дальнейшем для выравнивания. Файл, содержащий исходную нуклеотидную последовательность. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
TAKE HOME MESSAGE"Только мутации в половых клетках наследуются" - Этот тезис применим к группе организмов, размножающихся только половым путем. В случае бесполого и вегетативного размножения, свойственного многим живым организмам, в том числе большинству растениям, грибам, некоторым животным и т.д, утверждать подобное не является целесообразное. Контрпример: существуют виды, не размножающиеся половым путем, и тем не менее нельзя утверждать, что в их геномах не происходят мутации. Зеленая водоросль хлорелла, размножающаяся лишь вегетативно, делением клетки пополам, будучи одноклеточной, передает все возникающие соматические мутации потомству. При бесполом размножении у грибов, мутации возникающие не в половых клетках, будут также переданы потомству. "В гомологичных последовательностях живущих сегодня организмов мы видим почти исключительно мутации, прошедшие отбор" - многие вредные мутации, не являющиеся летальными, все же снижают жизнеспособность особи-носителя, понижают ее шансы на выживание, и, вероятно, в дальнейшем приведут к гибели. Во-первых, если в тезисе идет речь о нуклеотидных последовательностях, часть вредных мутаций, могут не проявлятся в фенотипе, но все же накапливаться генетическим грузом в генотипе. Пример - рецессивные мутации. Такие мутации безусловно могут присутствовать в геномах живущих организмов, но про них не в кое мере нельзя сказать, что они прошли отбор. При серповидно-клеточной анемии, гоммологичная последовательность живущих на сегодняшний день нездоровых людей также существует, однако нельзя сказать, что эта последовательность прошла отбор. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Главнaя страница | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||