PSI-BLAST. СОСТАВЛЕНИЕ СЕМЕЙСТВА ГОМОЛОГОВ. | ||||||||||||||||||||||||||||||
В расках данного задания для белковой последовательности последовательности с идентификатором AC P74518 при помощи psi-blast было найдено семейство гомологов путем нескольких итераций поиска, результаты которых приведены в Таблице 1. Рассматриваемый белок является фактором стимулирования гибернации рибосомы (Ribosome hibernation promotion factor) организма Synechocystis sp. (strain PCC 6803 / Kazusa). Он необходим для димеризации активных 70S рибосом в 100S рибосомы в стационарной фазе; 100S рибосомы трансляционно неактивны и иногда присутствуют при экспоненциальном росте.[1] | ||||||||||||||||||||||||||||||
Таблица 1. Результаты итераций поиска PSI-BLAST P74518.
| ||||||||||||||||||||||||||||||
Как видно из таблицы, всего было произведено 4 итерации поиска (поск по банку Swiss-Prot, параметры по умолчанию). Уже на второй выборка стабилизировалась, на последующих итерациях количество находок выше порога не изменялось и составило 28. При этом на последних двух итерациях e-value худщей находки выше порога также не изменялось, а значение e-value лучшей находки ниже порога на четвертой итерации совпало со значением на второй. Так как разница между e-value лучшей находки ниже порога и худшей находки выше порога довольно высокое (порядка e-18), можно сказать, что найденная выборка действительно состоит из семейства гомологов. | ||||||||||||||||||||||||||||||
PROSITE | ||||||||||||||||||||||||||||||
| В данном задании расматривались паттерны, описывающие семейство бактериальных белков терминации трансляции 1, из нескольких
видов бактерий, работа с которыми велась во втором практикуме.
банке Prosite был осуществлен поиск по последовательности RF1_BURCA, который выявил наличие в последовательности одного паттерна. c идентификатором PS00745 (RF_PROK_I) и следующей консенсусной последовательностью: | ||||||||||||||||||||||||||||||
[ARH]-[STA]-x-G-x-G-G-Q-[HNGCSY]-[VI]-N-x(3)-[ST]-[AKG]-[IV] | ||||||||||||||||||||||||||||||
Далее было рассметоренно выравнивание 18 белков из 18 различных видов бактерий (fasta-файл), представленное на Рис.1. По 228-244 позициям выравнивания была построена более строгая консенсусная последовательность с добавлением рядомстоящих консервативных столбцов: | ||||||||||||||||||||||||||||||
R-[SA]-[SQ]-G-A-G-G-Q-H-[IV]-N-[TK]-T-[DE]-S-A-[IV]-R-[IVL]-T-H-[IL]-P-[TS]-G | ||||||||||||||||||||||||||||||
Рис.1 Выравнивание белков терминации трансляции 1 из 18 различных видов бактерий. 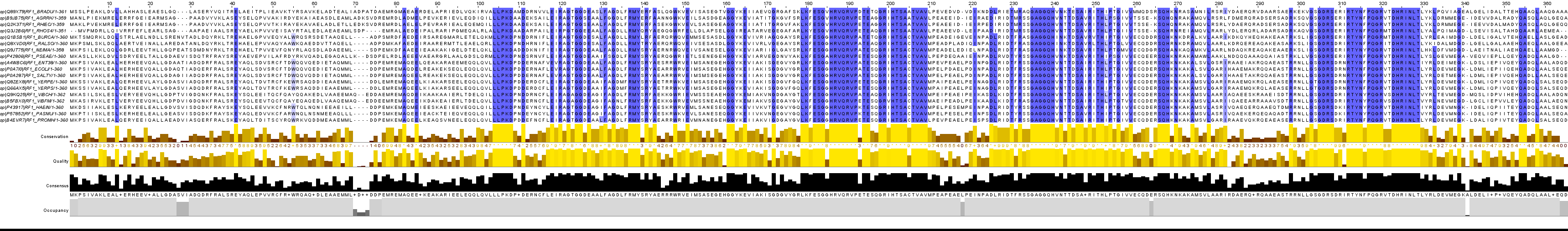 | ||||||||||||||||||||||||||||||
Далее в банке prosite был осуществлен поиск по новой консенсусной последовательности, который выявил 367 находок. После из банка Uniprot (c помощью запроса: mnemonic:rf1_* taxonomy:proteobacteria) были найдены всевозможные последовательности факторов терминации трансляции 1, число которых составило 408. Их сравнение производилось с помощью следующего скрипта. В результате сравнения были определены следующие параметры:
Те же самые действия были произведены для строгой консенснусной последовательности, построенной по выравниванию Рис.1, однако быз добавления рядомстоящих консервативных позиций. Число находок составило 480.
| ||||||||||||||||||||||||||||||
R-[SA]-[SQ]-G-A-G-G-Q-H-[IV]-N-[TK]-T-[DE]-S-A-[IV] | ||||||||||||||||||||||||||||||
CCЫЛКИ | ||||||||||||||||||||||||||||||
| Главнaя страница |