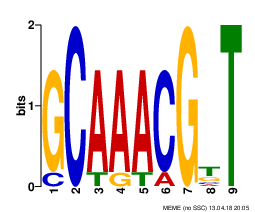ОТОБРАННЫЕ БАКТЕРИИ | |||||||||||||||||||||||||||||||||||||||||||||
В рамках данного практикума работа велась с видом бактрии Shigella flexneri или Шигелла Флекснера, одним из видов шигелл, вызывающих бактериальную дизентерию у человека. На сайте базы данных Uniprot с помощью запроса: | |||||||||||||||||||||||||||||||||||||||||||||
organism:"shigella flexneri" keyword:"purine biosynthesis" | |||||||||||||||||||||||||||||||||||||||||||||
| Были найдены все белки данного вида относящиеся к биосинтезу пуринов. Поиск выявил 254 записи о белках, из них 19 относились к категории Reviewed. Среди этих 19 записей, 8 принадлежали к штамму Shigella flexneri serotype 5b (strain 8401). С ними и велась дальнейшая работа. Информация об этих 8 записях, а таже о координатах кодирующих данные белки генов представлена в Таблице 1. | |||||||||||||||||||||||||||||||||||||||||||||
Таблица 1. Информация о записях 8 белков бактрии Shigella flexneri serotype 5b (strain 8401)
| |||||||||||||||||||||||||||||||||||||||||||||
Далее была найдена запись полного генома данной бактерии в ENA/EMBL (AC: CP000266.1). Из этой записи были получены Upstream-регионы для всех восьми данных генов из Таблицы 1, по координатам, указанным в Таблице 1 с помощью команды descseq. В качества Upstream-регионов брались 100 нуклетидов до начала гена. Файл в fasta-формате со всеми вырезанными последовательностями прилагается: cp000266.fasta | |||||||||||||||||||||||||||||||||||||||||||||
| Далее был осуществлен поиск мотивов среди последовательностей полученного файла с помощью программы MEME следующей командой (параметры задют поиск на прямой и комплементраной цепи, а также число мотивов, которые хотим найти): | |||||||||||||||||||||||||||||||||||||||||||||
ememe -dataset cp000266.fasta -revcomp -nmotifs 3 | |||||||||||||||||||||||||||||||||||||||||||||
| С результатами работы программы можно ознакомиться по ссылке, а также в Таблице 2. | |||||||||||||||||||||||||||||||||||||||||||||
Таблица 2. Результат работы MEME.
| |||||||||||||||||||||||||||||||||||||||||||||
Таким образом, ихз таблицы 3 можно заметить, что все 3 найденних программой мотива отличаются низким значением E-value (>0.001). Наиболее заслуживающим внимания безусловно является первый мотив, с наименьшим значением E-value (6.7e-002) и небольшой длинной. Однако так как поиск велся на прямой и комплементарной цепи, в 3 из 8 последовательностях, в которых был найден данный мотив, мотив был найден на комплементарной для Upstream-региона цепочке.Второй мотив обладает очень высокой длиной и высоким e-value(1.5e+003) при этом в 4 из 8 последовательносте1, в которых встретился этот мотив, он был найден на цепочке, комплементарной цепи с Upstream-регионом (и геном соответственно). Третий мотив обладает высоким e-value и был встречен только в двух последовательностях, в одной из которых на цепочке, комплементарной Upstream-региону. | |||||||||||||||||||||||||||||||||||||||||||||
Также можно ознакомится, с Таблицей 3, в которой суммированы все данные по практикуму. | |||||||||||||||||||||||||||||||||||||||||||||
Таблица 3. Отчет.
| |||||||||||||||||||||||||||||||||||||||||||||
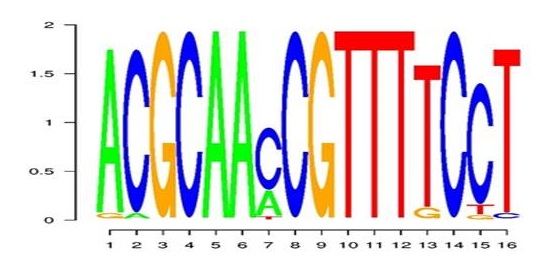
Также было произведено срапвнение LOGO для сайта связывания пуринового репрессора E.coli с первым мотивом: | |||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||
Из Рис.1 и Рис.2 видно, что первый мотив найденный программой MEME полностью содерижится в LOGO для сайта связывания пуринового репрессора E.coli в позициях с 3 по 11. При этом небольшое отличие наблюдается в 5(7) позиции, где в LOGO E.coli вместо A преобладает С. | |||||||||||||||||||||||||||||||||||||||||||||
Также был произведен поиск найденных с помощью meme мотивов по всему геному бактерии. Поиск производился с помощью программы emast. На вход подвавлся файл, полученный на выходе работы программы meme - meme.txt, с измененной строчкой "DATAFILE= MEME/meme.fasta" на "DATAFILE= сp000266.fasta", где сp000266.fasta - название файла, содержащего геном бактерии. С результатами работы программы можно ознакомиться по Ссылке. Команда была задана следующим образом: | |||||||||||||||||||||||||||||||||||||||||||||
emast meme.txt | |||||||||||||||||||||||||||||||||||||||||||||
Видно, что все три мотива были найдены по всему геному в количествах превыщающих 8 генов связанных с пуриновым синтезом. | |||||||||||||||||||||||||||||||||||||||||||||
| Главнaя страница |