|
Карань Анна |
|||
| Главная | О себе | Учеба | ФББ МГУ |
Поиск сигналов. Chip-seq
Цель: Определите сайты связывания данного транскрипционного фактора в данном участке хромосомы человека
1. Контроль качества чтений
В данном практикуме необходимо определить сайты связывания данного транскрипционного фактора в данном участке хромосомы. Файлы .fastq с ридами Illumina, полученные в результате ChIP-seq эксперимента, разделены на отдельные файлы, соответствующие участкам хромосом. Предложенный мне файл для анализа: chipseq_chunk47.fastq, лежащий в папке /srv/databases/ngs/annakaran. Сначала нужно сделать контроль качества прочтений с помощью программы FastQC. Если необходимо, то результаты следует отфильтровать с помощью программы Trimmomatic.
| fastqc chipseq_chunk47.fastq |
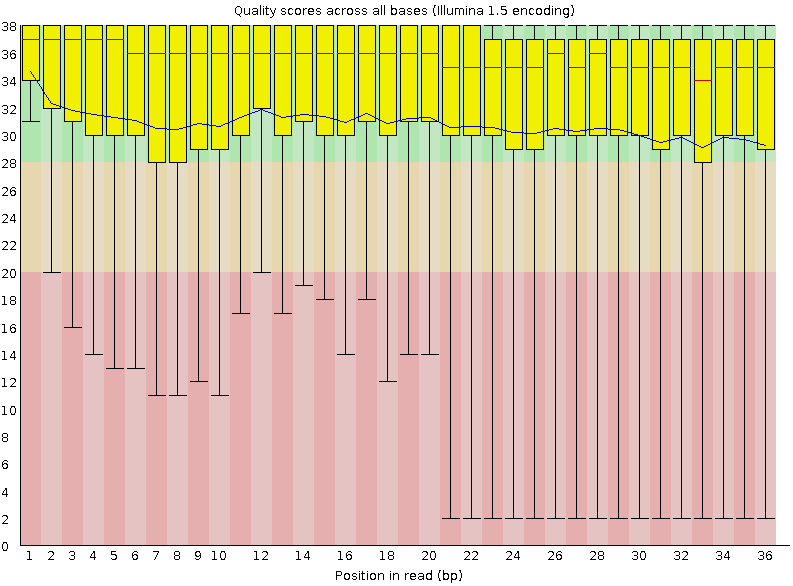
chipseq_chunk47_fastqс.html - выходной файл программы в виде html страницы. Как видно на Рис. 1 среднее значения качества чтений приемлимо, однако слишком много выпадений со значениями качества ниже 20 по всем позициям. Поэтому я считаю нужным воспользоваться программой Trimmomatic для отфильтровывания результатов.
| java -jar /usr/share/java/trimmomatic.jar SE -phred33 chipseq_chunk47.fastq trimm.fastq TRAILING:50 MINLEN:36 chipseq_chunk47.fastq - входной файл trimm.fastq - выходной файл MINLEN:36 - удаляет прочтения короче 36 TRAILING:50 - удаляют риды с качеством ниже 50 |
Изначально было 19622 рида, после очитски осталось 16512, т.е. 84,15%. Далее с помощью программы FastQC проверим качество после очистки.
| fastqc trimm.fastq |
|
|
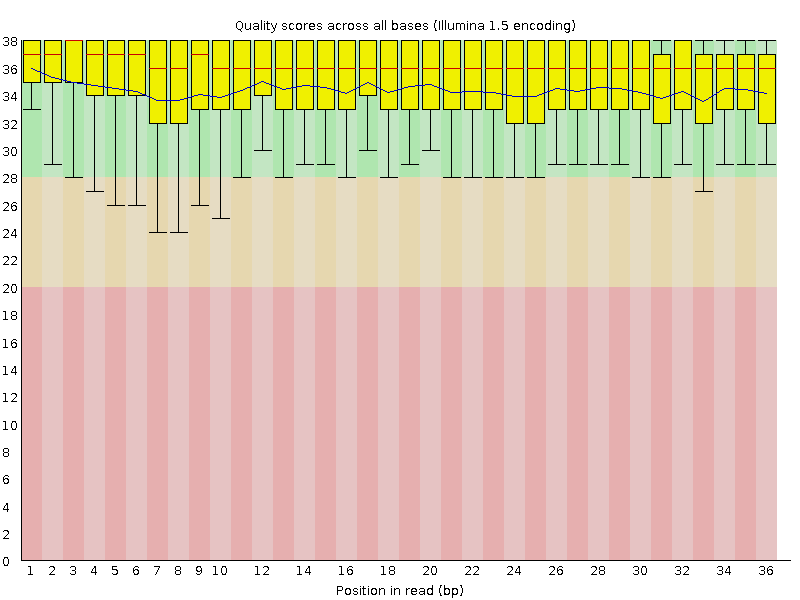
Видно, что качество после очистки значительно улучшилось, и средние значения стали выше и нет выпадений с качеством ниже 20.
2. Картирование прочтений на геном человека hg19
Картирование прочтений на геноме человека hg19 осуществлялось с помощью программы BWA.
| bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk41_out.fastq > chipseq_chunk41.sam |
Для анализа полученных с помощью BWA результаты использовались следующие команды:
| samtools view chipseq_chunk47.sam -bSo chipseq_chunk47.bam - файл с выравниванием переводится в бинарный формат .bam samtools sort chipseq_chunk47.bam -T chip_temp -o chipseq_chunk47_sort.bam - сортирует выравнивание ридов и референса по координате рида в референсе samtools index chipseq_chunk47_sort.bam - полученный файл сортируется samtools idxstats chipseq_chunk47_sort.bam > count.out - считает, сколько ридов откартировалось |
В результате получен файл, где риды откартированы на геном: count.out Откартировались все 16512 прочтений, 14618 (88,53%) из которых откартировались на 11 хромосому. Из чего можно сделать вывод, что для работы мне были предложены риды с 11 хромосомы.
3. Поиск пиков (Peak calling)
Для поиска пиков будет использовать программы MACS
| macs2 callpeak -n chipseq_chunk47 -t chipseq_chunk47_sort.bam --nomodel |
В результате было получено 3 файла: chipseq_chunk47_summits.bed, chipseq_chunk47_peaks.xls, chipseq_chunk47_peaks.narrowPeak. Всего было найдено 9 пиков, 7 из которых в хромосоме 11. Их визуалиция в UCSC Genome Browser показана на Рис. 3. Для визуализации в браузер загружался файл chipseq_chunk47_peaks.narrowPeak с дописанной в начало строкой - "track type=narrowPeak visibility=3 db=hg19 name="my_peaks" description="Peaks from chunk 47" browser position chr11:76747540-78287893" и без пиков не из 11 хромосомы.
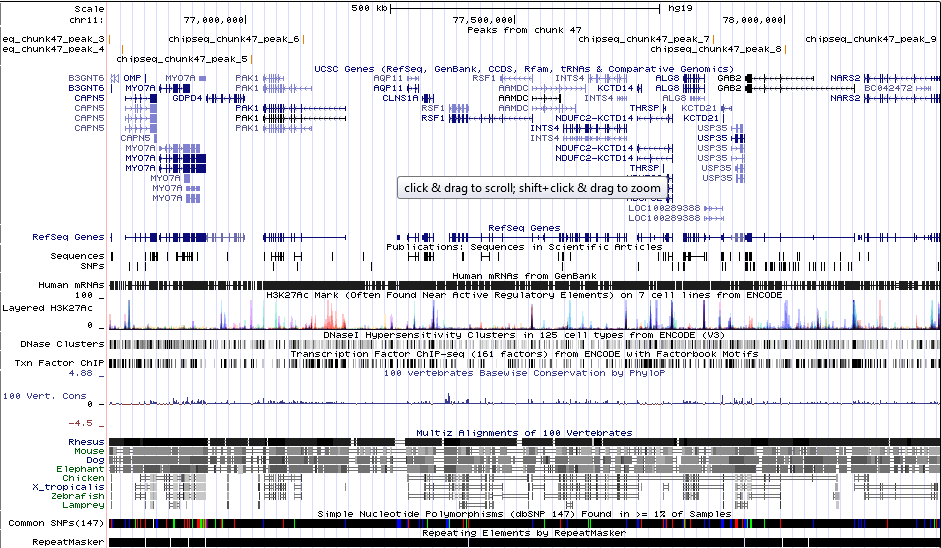
| Таблица 1. Характеристики пиков, найденных в 11 хромосоме | ||||
| Номер | Координаты | Длина | Расстояние от старта пика до его вершины | -log10(p-value) |
| 1 | 76747536 - 76747766 | 231 | 174 | 15.03431 |
| 2 | 76772234 - 76772490 | 257 | 94 | 22.79714 |
| 3 | 77010689 - 77010928 | 240 | 93 | 26.48665 |
| 4 | 77107440 - 77107664 | 225 | 66 | 12.65187 |
| 5 | 77867476 - 77867701 | 226 | 166 | 26.40125 |
| 6 | 78001328 - 78001618 | 291 | 43 | 10.66569 |
| 7 | 78287669 - 78287893 | 225 | 114 | 17.28921 |
Каждому найденному пику соответствует число — -log10(p-value),по которому можно оценить достоверность находки. При этом чем больше это число, тем меньше p-value и, следовательно, тем выше достоверность находки. Рассмотрим в более крупном масштабе пики с наименьшим p-value, т.е. пики по номерами 3 и 5 в Таблице 1.
Рис.4 Пик 3 в более крупном масштабе
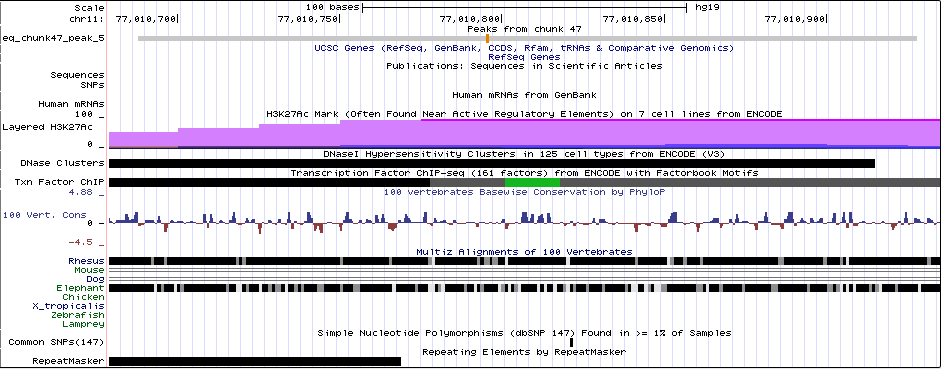
Рис.5 Пик 5 в более крупном масштабе
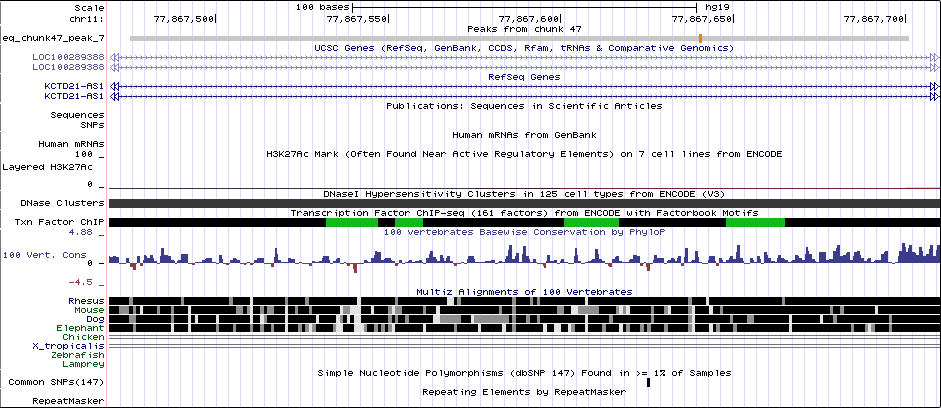
KCTD21-AS1 (KCTD21 Антисмысловая РНК 1) - это РНК ген и связаг с некодирующим классом РНК.
©Карань Анна, 2015