|
Карань Анна |
|||
| Главная | О себе | Учеба | ФББ МГУ |
Работа с KEGG ORTHOLOGY
Задание 1. Выбор пары ортологических рядов для дальнейшей работы
| Название выбранного пути | Nicotinate and nicotinamide metabolism (Метаболизм никотината и никотинамида) |
| Выбранная реакция | Nicotinamide <-> NAD+ |
| Число EC для данной реакции | 3 |
| Был выбран тот ЕС, у которого 2 ортологических ряда и чуть больше генов, чем у другого, иначе следующие задания будут менее интересны | |
| Число ортологических рядом выбранного EC (3.2.2.6) | 2 |
| Идентификаторы этих рядов | K01242, K18152 |
| Число генов выбранных ортологических рядов, представленных в базах данных | K01242 - 118, K18152 - 72 |
| Число белков выбранных ортологических рядов, представленных в базах данных | K01242 - 41 (36 в Uniprot), K18152 - 22 (19 в Uniprot) |
| Число метаболических путей, в которых участвууют белки выбранного ортологического ряда | K01242 - 16, K18152 - 8 |
| Число реакций, выполняемых белками выбранного ортологического ряда | K01242 - 12, K18152 - 12 |
| Ссылка на эту реакцию и ортологические ряды | |
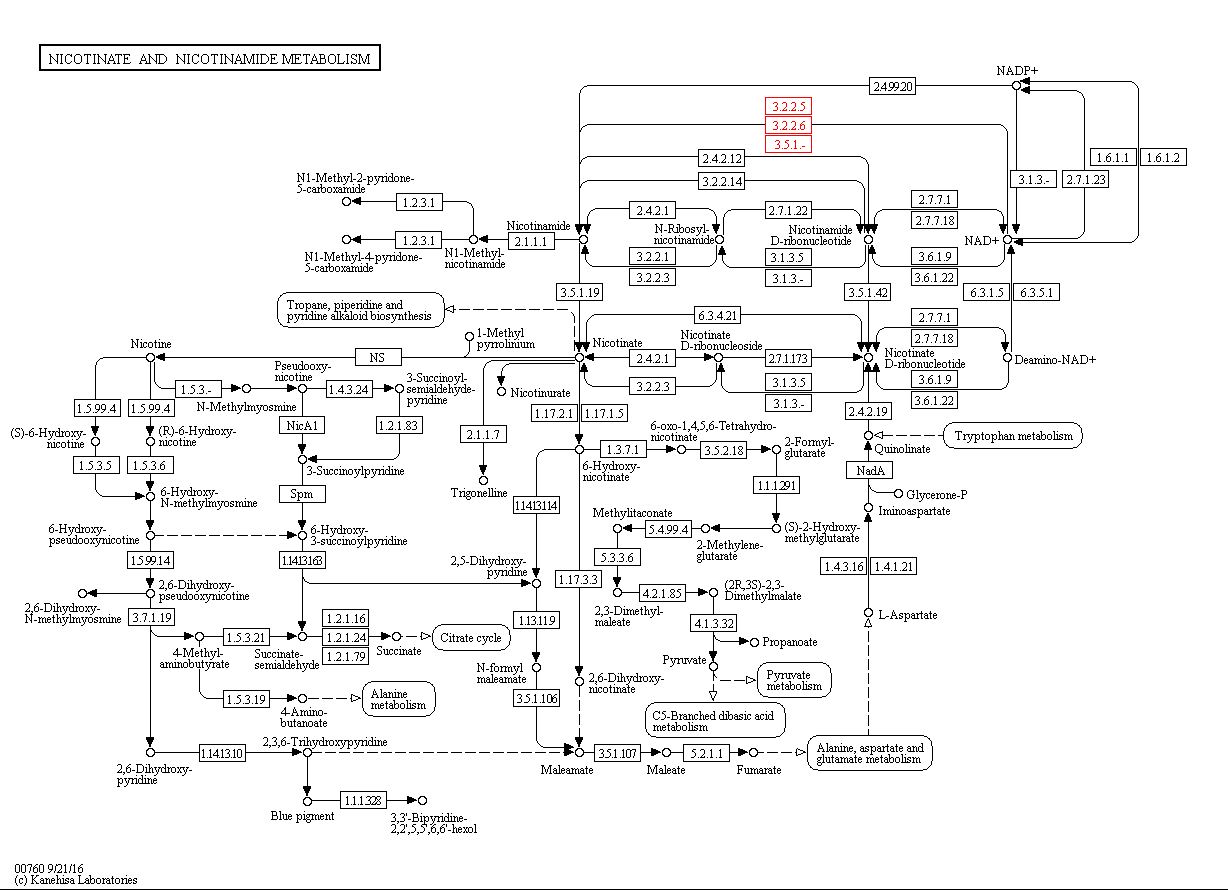
Рис.1 Метаболический путь никотината и накотинамида (красным выделена выбранная реакция, выбранный EC - 3.2.2.6).
Задание 2. Получение совместного множественного выравнивания
Сначала я получила последовательности белков этих ортологических рядов, представленных в базе данных Uniprot - последовательности. Далее с помощью Musle в Mega сделала множественное выравнивание. А дальше импортировала выравнивание в Jalview и покрасила Clustalx - проект Jalview.
Задание 3. Проверка гомологичности белков в выравнивании
Проверка выравнивания
В выравнивнивании есть один плохо выравненный белок - A0A0D9RV45|K01242, есть участки, не авыравненные
совсем с остальными, т.е. у остальных белков гэпы, а есть наоборот, т.е. он явно сильно выделяется.
Также есть белки - B3RIY1|K01242, V4AMV5|K01242, P28907|K18152, P56528|K18152, Q5VAN0|K18152, Q64244|K18152,
Q9MZ03|K18152 с большим числом гэпов в конце, и вообще белки ортологического ряда K18152 в конце
выравниваются не очень хорошо с большим числом гэпов, а в начале выравнивания наоборот. По
всему выравниваю белки ряда K18152 выравнелись хуже.
Гомологичность белков в выравнивании: Но во-первых, рассматривать только один ортологический ряд
не имеет смысла, поэтому я удалю только самые плохие последовательнсоти, а во-вторых, все-таки
и у этих белков есть не мало вертикальных блоков (если говорить не об абсолютно консервативных позициях,
а об абсолютно функционально консервативных, и не для всех последовательностей сразу, а больше, чем
для 60%, т.е. не только для хорошо выравненного ряда K01242). В таком случае тут около 13 вертикальных
блоков на выравнивание длиной 401 нуклеотид.
Думаю, в данном случае можно сделать предположение о наличии множественного выравнивания, но учитывать,
что ортологический ряд K18152 выравнился сильно хуже.
Рис.2 Выравнивание раскрашенное по схеме Clustalx c
удаленными плохо выравненными последовательностями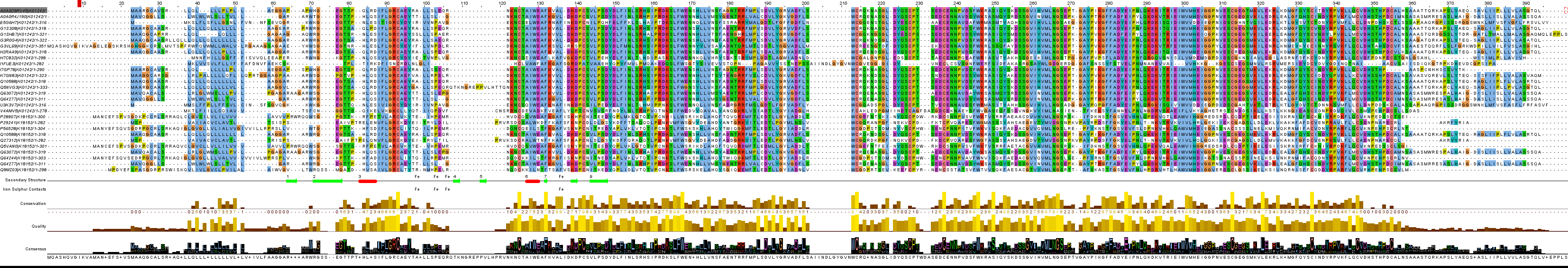
Задание 4. Построение филогенетического дерева
 Рис.3 Полученное дерево |
 Рис.4 Дерево после укоренения |
На Рис.3 изображено построенное методом Neighbor-Joining дерево. Чтобы разделить ряды я попробовала его укоренить (Рис.4), однако дерево тогда получается несбалансированным. А еще на Рис.4 красным выделены белки ряда K18152, попавшие в группу белков ряда K01242, таких 3 из 10, т.е. 30% белков попали не в свою кладу. И что самое логично, это именно те белки, которые я считала выравненными лучше (сзади нету гэпов и все выравнивание лучше, Рис. 2). Я решила выравнять отдельно последовательности K18152 - выравнивание K18152.

Рис.5 Выравнивание раскрашенное по схеме Clustalx только с последовательностями белков ряда K18152
На Рис.5 изображено выравнивание только белков ряда K18152, и если сравнить его с частью Рис.2, то видно, что оно совсем не улучшилось. Значит, белки ряда K18152 почему-то далеки друг от друга, а некоторые даже ближе к белкам ряда K01242. Попробуем сравнить эти два ортологических ряда.
Сравнение ортологических рядов K18152 и K01242
Белки ряда K01242 участвуют в намного большем числе метаболических путей и в очень разных системах организма, и в иммунной, и эндокринной, и в пищеварительной, а K18152 только в пищеварительной. С одной стороны разнообразие функций и место наоборот должно приводить к большим отличиям в ряду K01242, чем в K18152, но все как раз наоборот. Можно, конечно, сказать, что белки ряда K01242 важнее, поэтому более консервативны, однако все-таки белки этих рядов выполняют схожие функции и являются схожими ферментами, поэтому пока этого объяснения недостаточно. Я нашла статью, где сравниваются белки CD38 (ряд K01242) и BST-1 (ряд K18152) [1]. Однако, в ней они как раз указывают на сходство этих мембранных ADP-рибозил циклаз, хотя идентичность в лучшем случае составляла 33%. А вот в другой статье [2] сравниваются функционально важные нуклеотиды и вторичная структура CD38, BST-1 и Aplysia cyclase (фермент того же семейства из овоцитов Aplysia, показывающий 27-35% сходство последовательностей с CD38 и BST-1.

Рис.6 Выравнивание CD38, BST-1 и Aplysia cyclase с отмеченными функционально важными участками и вторичной структурой
На Рис. 6 видно, что функционально важные участки этих трех белков совпадают.
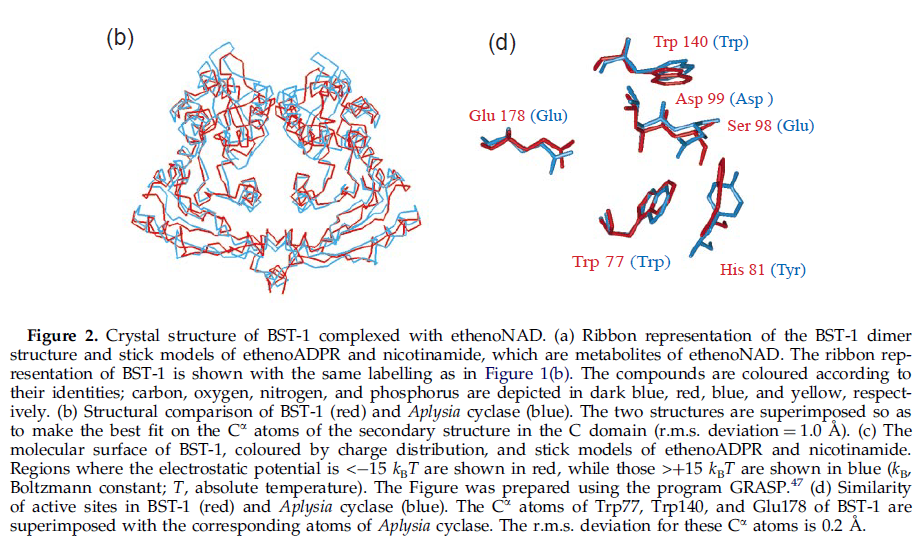
Рис.7 Сравнение вторичной структуры и аквтивных центров BST-1 и Aplysia cyclase
На Рис. 7 видно, что их вторичные стуктуры и активные центры очень похожи, что наводит на мысль о похожей ситуацией с CD38, т.е. схожести вторичной структуры без схожести последовательностей. Есть и много других статей, например [3] , где активно говорят о функциональной и стуктурной схожести, близкой связи этих белков (здесь о наличии особенных CpG island и других схожих особенностей). Это все позволило мне объяснить возможность таких различий между рядами, но не причины, которые стали наоборот туманне после прочтения статей и связи этих белков.
Список литературы
1. TSUNEYASU KAISHO, BST-1, a surface molecule of bone marrow stromal cell lines that facilitates pre-B-cell growth 2. Sumie Yamamoto-Katayama and other, Crystallographic Studies on Human BST-1/CD157 with ADP-ribosyl Cyclase and NAD Glycohydrolase Activities. 3. Ferrero E, The human CD38 gene: polymorphism, CpG island, and linkage to the CD157 (BST-1) gene.
©Карань Анна, 2015