|
Карань Анна |
|||
| Главная | О себе | Учеба | ФББ МГУ |
Поиск сигналов. Теория
Задание 1. Определение биологической роли определенного транскрипционного фактора в бактерии
Мы проводили ииследование на Mycobacterium tuberculosis H37Rv. LexA_Myco.fasta - файл с участками ДНК, с которыми связывается данный транскрипционный фактор. Сначала нужно с помощью программы MEME найти мотив связывания. Однако, данной программе нельзя подавать одинаковые последовательности, поэтому файл был отфильтрован с помощью скрипта meme.py, и был получен fasta файл c 57 gjcktljdfntkmyjcnzvb без повторений - single_LexA.fasta. Эта программа безумно медленная.
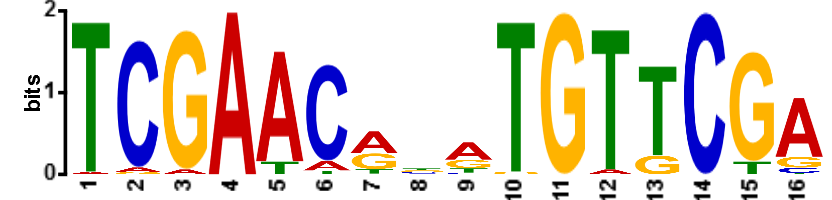
Рис.1 Logo сайта связывания транскрипционного фактора, полученное с помощью программы MEME.
| Таблица 1. Характеристика найденного MEME сайта | |
| E-value | PWM |
| 1,8e-329 |
[0.017544, 0.000000, 0.000000, 0.982456],
[0.035088, 0.947368, 0.017544, 0.000000],
[0.035088, 0.000000, 0.964912, 0.000000],
[1.000000, 0.000000, 0.000000, 0.000000],
[0.894737, 0.000000, 0.000000, 0.105263],
[0.087719, 0.877193, 0.000000, 0.035088],
[0.508772, 0.017544, 0.368421, 0.105263],
[0.122807, 0.280702, 0.280702, 0.315789],
[0.543860, 0.052632, 0.263158, 0.140351],
[0.000000, 0.000000, 0.017544, 0.982456],
[0.000000, 0.000000, 1.000000, 0.000000],
[0.035088, 0.000000, 0.000000, 0.964912],
[0.000000, 0.000000, 0.175439, 0.824561],
[0.000000, 1.000000, 0.000000, 0.000000],
[0.000000, 0.000000, 0.894737, 0.105263],
[0.771930, 0.070175, 0.140351, 0.017544] .
|
Далее полученный мотив нужно было отправить на вход программе tomtom, ищущей похожие мотивы, предсказанные для этого транскрипционного фактора в базе данных RegTransBase.
| Таблица 2. Характеристика находок/лучшей находки tomtom | ||||
| Число встреч мотива находки с похожей PWM в последовательности | Длина мотива | Число выдаваемых лучших мотивов | Название TФ находки с похожей PWM | E-value |
| 39 | 16 | 165 | Transcriptional regulator PsrA | 3.80e-03 |
| PWM |
[0.486624, 0.000588, 0.000588, 0.512200],
[0.000691, 0.000588, 0.000588, 0.998133],
[0.000691, 0.026164, 0.128465, 0.844680],
[0.128568, 0.537673, 0.026164, 0.307596],
[0.998133, 0.000588, 0.000588, 0.000691],
[0.895831, 0.000588, 0.000588, 0.102992],
[0.998133, 0.000588, 0.000588, 0.000691],
[0.000691, 0.998031, 0.000588, 0.000691],
[0.640077, 0.000588, 0.358644, 0.000691],
[0.384322, 0.358644, 0.128465, 0.128568],
[0.230869, 0.358644, 0.230767, 0.179718],
[0.000691, 0.384220, 0.026164, 0.588926],
[0.000691, 0.000588, 0.998031, 0.000691],
[0.000691, 0.026164, 0.000588, 0.972558],
[0.026266, 0.000588, 0.000588, 0.972558],
[0.000691, 0.000588, 0.000588, 0.998133],
[0.282020, 0.026164, 0.563249, 0.128568],
[0.819105, 0.000588, 0.000588, 0.179718],
[0.154143, 0.230767, 0.051739, 0.563351],
[0.282020, 0.102890, 0.026164, 0.588926]
| |||
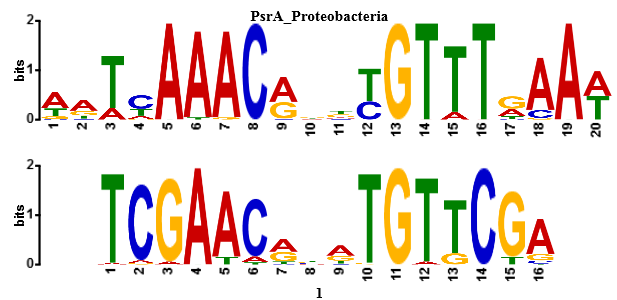
Рис.2 Logo находки tomtom и сайта, полученного с помощью программы MEME.
В Таблице 2 находка с похожей PWM была выбрана как первая находка, т.е. с минимальным E-value. Также Таблица 2 и Рис. 2 помогают сравнить находки tomtom и сайта, найденного МЕМЕ. Во-первых, находка короче, начинается со второго нуклеотида сайта и заканчивается за 2 до конца, и есть 2 несовпадающих консервативных нуклеотида. Далее найденный MEME мотив данного транскрипционного фактора нужно подать на вход программе FIMO с поиском по БД upstraem region, эта программа найдет данный мотив в регионах перед генами. Мотив нужно искать только в upstream region, потому что большинство транскрипционных факторов связываются с последовательностью до гена по направлению транскрипции (т.е. в upstream region). Файл с координатами сайтов связывания ТФ - fimo_upst.txt. Далее нужно найти координаты всех генов данной бактерии, по заданию это можно сделать с помощью команды featcopy из пакета emboss. Но в моем случае почему-то featcopy не распознавал ncRNA как feature, поэтому пришлось написать скрипт на питоне - gene_coord.py. В итоге был получен файл с координатами генов - Myco_genes.txt. Далее необходимо найти гены, экспрессию которых может регулировать ТФ. Считается, что ТФ регулирует экспрессию ближайшего гена в той же ориентации, что и сайт. А в файле fimo_upst.txt во второй колонке уже указаны гены, экспрессию которых может регулировать ТФ, нужно отобрать из их достоверные находки. Я считала достоверными находки с q-value меньше 0,01, таких оказалось 30 (Таблица 3)
| Таблица 3. Таблица найденных мотивов, полученная с помощью FIMO | |||||||
| # motif_id | motif_alt_id|sequence_name | start | stop | strand | score | p-value | q-value |
| 1 | YP_006516535.1|RVBD_3074 | 86 | 101 | + | 28.675 | 1.73e-10 | 0.000165 |
| 1 | YP_006514758.1|RVBD_1378c | 131 | 146 | + | 28.675 | 1.73e-10 | 0.000165 |
| 1 | YP_006517273.1|RVBD_3776 | 149 | 164 | + | 28.375 | 2.4e-10 | 0.000165 |
| 1 | YP_006516535.1|RVBD_3074 | 86 | 101 | - | 27.7875 | 3.45e-10 | 0.000165 |
| 1 | YP_006514758.1|RVBD_1378c | 131 | 146 | - | 27.7875 | 3.45e-10 | 0.000165 |
| 1 | YP_006516880.1|RVBD_3395A | 15 | 30 | + | 27.2 | 7.03e-10 | 0.00024 |
| 1 | YP_006516879.1|RVBD_3395c | 53 | 68 | - | 27.2 | 7.03e-10 | 0.00024 |
| 1 | YP_006516853.1|RVBD_3371 | 50 | 65 | + | 26.3625 | 1.82e-09 | 0.000346 |
| 1 | YP_006516853.1|RVBD_3371 | 50 | 65 | - | 26.3625 | 1.82e-09 | 0.000346 |
| 1 | YP_006516852.1|RVBD_3370c | 127 | 142 | + | 26.3625 | 1.82e-09 | 0.000346 |
| 1 | YP_006516852.1|RVBD_3370c | 127 | 142 | - | 26.3625 | 1.82e-09 | 0.000346 |
| 1 | YP_006513661.1|RVBD_0335c | 17 | 32 | - | 25.9 | 2.03e-09 | 0.000346 |
| 1 | YP_006513848.1|RVBD_0515 | 71 | 86 | + | 25.9 | 2.03e-09 | 0.000346 |
| 1 | YP_006513662.1|RVBD_0336 | 110 | 125 | + | 25.9 | 2.03e-09 | 0.000346 |
| 1 | YP_006515094.1|RVBD_1702c | 550 | 565 | - | 24.875 | 4.86e-09 | 0.000771 |
| 1 | YP_006515515.1|RVBD_2100 | 289 | 304 | - | 24.725 | 5.17e-09 | 0.000771 |
| 1 | YP_006514361.1|RVBD_1000c | 29 | 44 | + | 23.95 | 9.66e-09 | 0.00127 |
| 1 | YP_006516163.1|RVBD_2719c | 33 | 48 | + | 23.85 | 1.01e-08 | 0.00127 |
| 1 | YP_006516164.2|RVBD_2720 | 203 | 218 | - | 23.85 | 1.01e-08 | 0.00127 |
| 1 | YP_006516737.1|RVBD_3261 | 171 | 186 | - | 23.6375 | 1.14e-08 | 0.00128 |
| 1 | YP_006516736.1|RVBD_3260c | 216 | 231 | + | 23.6375 | 1.14e-08 | 0.00128 |
| 1 | YP_006516181.1|RVBD_2737c | 72 | 87 | + | 23.5 | 1.23e-08 | 0.00128 |
| 1 | YP_006516182.1|RVBD_2737A | 108 | 123 | - | 23.5 | 1.23e-08 | 0.00128 |
| 1 | YP_006516164.2|RVBD_2720 | 70 | 85 | - | 21.9375 | 3.41e-08 | 0.00312 |
| 1 | YP_006516163.1|RVBD_2719c | 166 | 181 | + | 21.9375 | 3.41e-08 | 0.00312 |
| 1 | YP_006516036.1|RVBD_2595 | 22 | 37 | - | 21.775 | 3.61e-08 | 0.00312 |
| 1 | YP_006516035.1|RVBD_2594c | 72 | 87 | + | 21.775 | 3.61e-08 | 0.00312 |
| 1 | YP_006516737.1|RVBD_3261 | 171 | 186 | + | 21.65 | 3.81e-08 | 0.00312 |
| 1 | YP_006516736.1|RVBD_3260c | 216 | 231 | - | 21.65 | 3.81e-08 | 0.00312 |
| 1 | YP_006516020.1|RVBD_2579 | 9 | 24 | - | 21.5125 | 4.06e-08 | 0.00312 |
| 1 | YP_006516019.1|RVBD_2578c | 84 | 99 | + | 21.5125 | 4.06e-08 | 0.00312 |
Однако FIMO дает только координаты мотива относительно гена, в регуляции которого он возможно участвует, поэтому необходимо найти абсолютные координаты генов и на их основе вычислить абсолютные координаты мотивов (Таблица 4).
| Таблица 4. Координаты мотивов и генов, которые ТФ возможно регулирует (а также белки, кодируемые этими генами) | |||
| Название гена | Координаты гена | Координаты найденного мотива | Функции (описание) гена |
| RVBD_3074 | 3436776 3438050 | 3436675 3436690 | hypothetical protein |
| RVBD_1378c | 1551234 1552661 | 1551088 1551103 | hypothetical protein |
| RVBD_3776 | 4221267 4222826 | 4221267 4221118 | hypothetical protein |
| RVBD_3074 | 3436776 3438050 | 3438136 3438151 | hypothetical protein |
| RVBD_1378c | 1551234 1552661 | 1552792 1552807 | hypothetical protein |
| RVBD_3395A | 3811899 3812525 | 3811914 3811929 | membrane protein |
| RVBD_3395c | 3811202 3811816 | 3811869 3811884 | hypothetical protein |
| RVBD_3371 | 3785112 3786452 | 3785047 3785062 | diacylglycerol acyltransferase |
| RVBD_3371 | 3785112 3786452 | 3786502 3786517 | diacylglycerol acyltransferase |
| RVBD_3370c | 3781681 3784920 | 3781539 3781554 | DNA polymerase III alpha subunit DnaE2 |
| RVBD_3370c | 3781681 3784920 | 3785047 3785062 | DNA polymerase III alpha subunit DnaE2 |
| RVBD_0335c | 399535 400050 | 400067 400082 | PE family protein PE6 |
| RVBD_0515 | 606554 608065 | 606468 606483 | 13E12 repeat family protein |
| RVBD_0336 | 400192 401703 | 400067 400082 | 13E12 repeat family protein |
| RVBD_1702c | 1927218 1928582 | 1929132 1929147 | hypothetical protein |
| RVBD_2100 | 2358397 2360049 | 2360338 2360353 | hypothetical protein |
| RVBD_1000c | 1116535 1117152 | 1116491 1116506 | hypothetical protein |
| RVBD_2719c | 3031050 3031547 | 3031002 3031017 | cell wall hydrolase |
| RVBD_2720 | 3031798 3032508 | 3032711 3032726 | repressor LexA |
| RVBD_3261 | 3640540 3641535 | 3641706 3641721 | F420 biosynthesis protein FbiA |
| RVBD_3260c | 3639869 3640138 | 1639638 1639653 | transcriptional regulator WhiB-like WhiB2 |
| RVBD_2737c | 3049062 3051434 | 3048975 3048990 | protein RecA |
| RVBD_2737A | 3051629 3051802 | 3051910 3051925 | cysteine rich protein |
| RVBD_2720 | 3031798 3032508 | 3032578 3032593 | repressor LexA |
| RVBD_2719c | 3031050 3031547 | 3030869 3030884 | cell wall hydrolase |
| RVBD_2595 | 2925502 2925747 | 2925769 2925784 | antitoxin VapB40 |
| RVBD_2594c | 2924827 2925393 | 2924740 2924755 | crossover junction endodeoxyribonuclease RuvC |
| RVBD_3261 | 3640540 3641535 | 3640354 3640369 | F420 biosynthesis protein FbiA |
| RVBD_3260c | 3639869 3640138 | 3640354 3640369 | transcriptional regulator WhiB-like WhiB2 |
| RVBD_2579 | 2903649 2904551 | 2904560 2904575 | haloalkane dehalogenase 3 |
| RVBD_2578c | 2902519 2903541 | 2902420 2902435 | hypothetical protein |
Также в Таблице 4 указаны кодируемыми этими генами белки. Функции большинства еще не определены, а вот про белки с известными функциями стоит рассказать подробнее. 1) Diacylglycerol acyltransferase относится к группе O-ацилтрансфераз, катализирует синтез триглицеридов из диацилглицерода и Ac-CoA, поэтому этот фермент необходим для образования жировой ткани. 2) DNA polymerase III alpha subunit DnaE2 - субъединица всем известной ДНК-полимеразы III, являющейся главным ферментативным комплексом, задействованным в репликации ДНК у прокариот. Оня обладает высокой процессивностью в отличие от других ДНК-полимераз прокариот. 3) Repressor LexA - транскрипционный репрессор генов SOS-ответа, кодирующих прежде всего полимеразы с высокой частотой ошибок, репарационные ферменты ДНК и ингибиторы клеточного деления. LexA фактически представляет собой двухкомпонентную регуляторную систему с RecA, которая распознает повреждение ДНК у остановившейся репликационной вилки и преобретает активную конформацию, способную связываться с LexA, что вызывает расщепление LexA - автопротеолиз. 4) Transcriptional regulator WhiB-like WhiB2 обеспечивает защиту от иммунитета хозяина. Так что нельзя говорить об общности всех белков, в которых был найден мотив, тут и ферменты синтеза предшественника жирных кислот и клеточной стенки, а есть и ДНК-полимераза и транскрипционные фактора, можно лишь выделить некоторые группы, например, LexA и RecA, обеспечивающие SOS-ответ.
Задание 2. Проверить, может ли метилирование повлиять на связывание исследуемого ТФ со своим сайтом?
Метилирование ДНК — это модификация молекулы ДНК без изменения самой нуклеотидной последовательности ДНК, что можно рассматривать как часть эпигенетической составляющей генома. Рассмотрим только несколько находок с самыми высокими p-value - met1, met2, met3, met4. Программа fuzznuc пакета EMBOSS предназначена для поиска паттернов в заданной последовательности. Эта программа использовалась для поиска сайтов метилирования, пересекающихся с найденными FIMO в Задании 1 мотивами (были взяты сами мотивы и участки ± 50 нуклеотидов по бокам от них, см. файлы строкой выше). В участках искались сайты из файла MT_sites.txt, содержащего сайты метилирования:
| fuzznuc -sequence metX.fasta -pattern @MT_sites.txt -outfile fuzznucX.out |
В результате были получены файлы - fuzznuc1.out, fuzznuc2.out, fuzznuc3.out, fuzznuc4.out. Замечтально то, что находок длиной меньше, чем 4 нуклеотида (а это минимальная длина паттерна метилирования) не было, т.е. программа искала конкретно именно эти паттерны, а не совпадения маленьких частей.
| Таблица 5. Число находок паттернов | ||||
| Идентификатор | YP_006516535.1 | YP_006514758.1 | YP_006517273.1 | YP_006516879.1 |
| Число находок | 185 | 140 | 169 | 180 |
Понятно, что не все эти находки являются реальными сайтами метилтрансфераз, так как задаваемые сайты либо просто короткие, либо длинные, но с большим числом неизвестных нуклеотидов. Так что если там есть какие-то из найденных сайтов, то однозначно не все, но все равно число находок впечатляет, поэтому какие-то из них наверняка являются истинными.
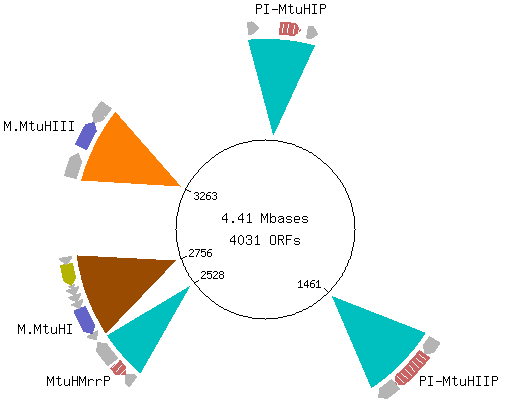
Рис.3. Выдача REBASE
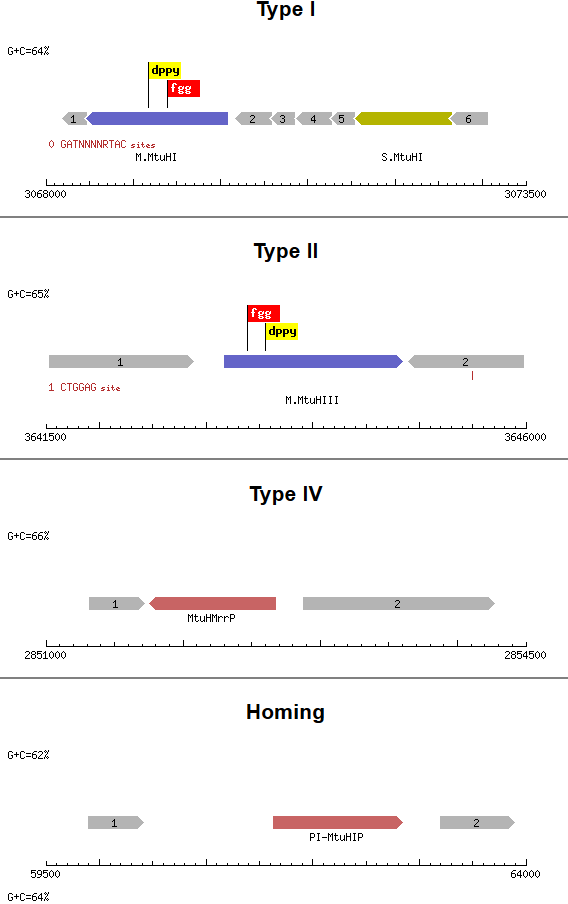
Рис.4. Сайты метитрансфераз генома Mycobacterium tuberculosis H37Rv
Как видно на Рис. 4 у данной бактерии один из сайтов метилтрансфераз - CTGGAG. И этот паттерн был найден программой fuzznac для нашего мотива, причем 4 раза в fuzznuc1.out: pattern221, pattern222, pattern225, pattern227. Второй сайт, присутствующий у Mycobacterium tuberculosis H37Rv по данным Rebase - это GATNNNNRTAC. Этот паттерн не был обнаружен fuzznac. Думаю, исходя из полученных данных можно говорить о влиянии метилирования на связывание ТФ со своим сайтом.
©Карань Анна, 2015