|
Карань Анна |
|||
| Главная | О себе | Учеба | ФББ МГУ |
Задание 9. Совмещение структур
Для данного практикума использовалась аминопептидаза N, уже анализируемая в прошлых практикумах этого семестра, с PDB ID 4QUO. С помощью PDBeFold ыли найдены 4 структурных гомолога рассматриваемого белка, а именно цепи А. Структуры выбирались так, чтобы RMSD был между 0.8 и 2.5, а длина выравнивания - более 50% от длины исследуемого белка. Summary выдачи показано на Рисунке 1, совмещение вторичных структур на Рисунке 2, и статистику по структурам на Рисунке 3.
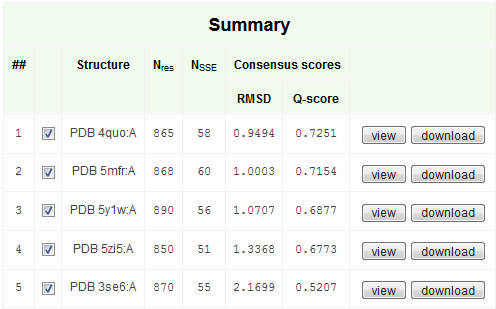
Рисунок 1. Summary выдачи PDBeFold

Рисунок 2. Выравнивание вторичных структур PDBeFold
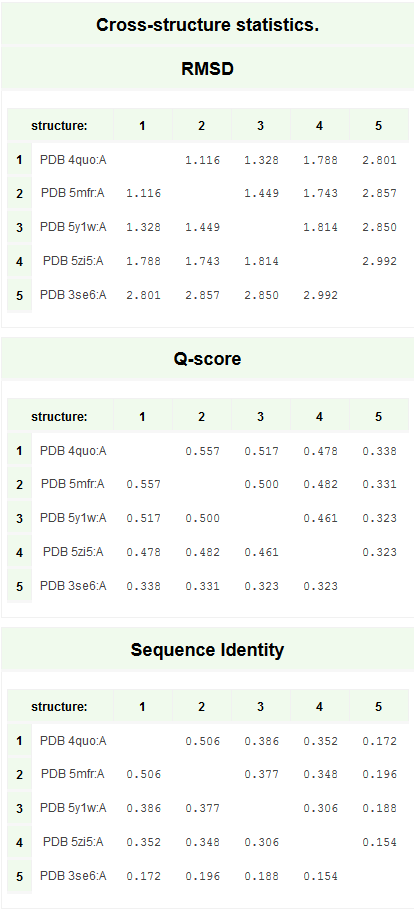
Рисунок 3. Статистика выдачи PDBeFold
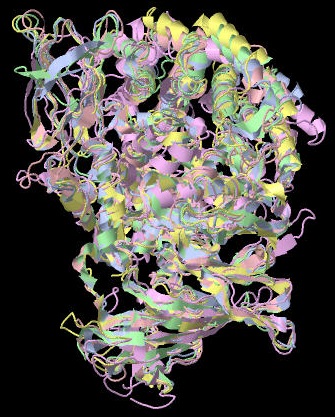
Рисунок 4. Совмещение структуры 4quo и его гомологов (5mfr, 5y1w, 5zi5, 3se6)
Как видно из Рисунка 4 общий вид структуры совпадает у всех 5-и, но многие конкретные элементы структуры совмещаются не очень хорошо.

Рисунок 5. Выравнивание на основе структур

Рисунок 6. Выравнивание программой Musle на основе последовательностей
На Рисунках 5 изображены выравнивания. На первый взгляд они достаточно сильно отличаются. Однако при более внимательном просмотре видно много одинаковых участков, и все они соответствуют крупным альфа-спиралям или бета-тяжам. Отсюда можно сделать вывод, что более правильным является выравнивание, построенное Musle, так как элементы вторичной структуры выравнились на основе последовательности и на основе структуры одинаково, а вот петли по структуре очевидно восстанавливать не стоит (а именно они и не совпадают в выравниваниях).
©Карань Анна, 2019