BLAST
Информация о параметрах BLAST
- Enter Query Sequence
В это поле вводится последовательность, по которой будет вестись поиск, или загружается из файла. Вместо последовательности можно написать индентификатор AC, но, как показывает практика, BLAST узнаёт не все AC-шники. - Choose Search Set
Это параметр, указывающий на базу данных, в которой будет проводится поиск, с возможностью ограничить организм и исключить некоторые классы последовательностей. По умолчанию стоит Non-redundant protein sequences, в задании же поиск в основном вёлся по базе данных SwissProt. - Program Selection
десь также можно указать, каким алгоритмом будет проводиться поиск.
- General Parameters - общие параметры поиска
Max target sequences - максимум находок, которые будут выводится в результатах поиска. По умолчанию 100.
Expect threshold - устанавливает верхний порог E-Value. E-value - это математическое ожидание получить выравнивание с большим или тем же весом в случайной базе данных одинакового размера и стеми же параметрами. Чем меньше E-Value, тем более значима находка. Соотвественно если верхняя граница E-Value достаточно мала, количество найденных последовательностей будет невелико.
Word size Алгоритмы BLAST работают быстро благодаря тому, что они сначала выкидывают "лишние" последовательности по такой схема: изначальная последовательность "разрезается" на "слова" определённой длины (по умолчанию - 6 а.к.), которые ищутся в последовательностях. Те последовательности, в которых нет хотя бы двух "слов", выкидываются, по оставшимся уже строится выравнивание. Так как слова меньшей длины найти проще, если уменьшить этот параметр, находок будет больше.
- Scoring Parameters
Matrix - матрица замен, по которой вычисляется вес выравнивания. Веса замены в каждой клетке матрица зависят от частоты замены одной а.к. на другую. По умолчанию BLOSUM62 - матрица, основанная на базе, в которой у любых двух белков Identity не больше 62%.
Gap costs - параметр, котоорый устонавливает штраф за гэпы и индели. За существование инделя берётся большой штраф, а за каждый гэп, увеличивающий длину инделя - относительно маленький, то есть штраф афинные.
Compositional adjustments - борьба с участками малой сложности.
Поиск гомологии
В данном практикуме предлогалось найти с помощью сервиса BLAST гомологичные данному белку последовательности. В моём случае белок - часть АТФ-азы типа V. В базе данный Swissprot при параметре wordsize = 6 было найдено 1638 предположительно гомологичных последовательностей. Из получившегося списка из конца, начала и середины списка были выбраны 10, их последовательности скачаны и выравнены в программе Jalview. После этого были исключены четыре самые отличающиеся последовательности, оставшиеся шесть снова выравнены и покрашены по % Identity. На получившейся картинке можно хорошо увидеть несколько блоков с высоким процентом консервтивности, длина некоторых из них превышает 6 колонок, что обычно является доказательством гомологии белков.
Ссылка на таблицу с находкамиСсылка на выравнивание Jalview

Построение карты выравнивания
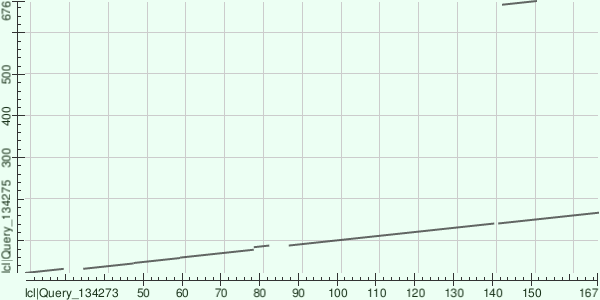
Для построяения карты выравнивания было выбрано два белка с ID Q5KK71_CRYNJ и G4TAW2_SERID. Первый - белок синтеза витамина B6, второй - не описанный белок. Оба встречаются у грибов подотдела Agaricomycotina. Так как BLAST не всегда распознаёт индентификаторы, последовательности белков были взяты с сайта Uniprot и вставлены в поля для сравнения в BLAST. Результатом является карта выравнивания, которую можно увидеть ниже. Гомологичным оказался участок приблизительно с 20-ой по 167-ую а.к., причём во второй последовательности в начале наблюдаются небольшие делеции. Также участку 143-151 первого белка нашёлся гомологичный участок 668-676 второго белка.
Поиск в BLAST
Для более глубокого понимания BLAST в последнем задании придлагалось провести поиск по случайной послдовательности. Я выбрала две строки Милтона из Paradise Lost без последнего слова (иначе поиск не давал результатов) и пробелов с заменой всех букв o на a (они, понятное дело, мешали поиску, так как "o" не соответствует ни одной а.к.): "allisnatlasttheuncanquerable".
Установленные параметры: база данный SwissProt, 20 тысяч находок и wordsize = 6. Удивительным образом нашлось 20 тысяч результатов(то есть скорее всего результатов ещё больше), правда с весом меньше 40. E-value варировало от 11 до десятков тысяч. Количество находок, наверное, можно объяснить маленькой длиной псевопоследовательности и, возможно, больши количеством буков a и l.
При замене базы данных с SwissProt на non-redundant protein sequences количество находок уменьшилось до 17,8 тысяч.
После замены параметра Expected Threshhold c 10 на 1e-20 количество результатов не сократилось, те осталось больше 20 тысяч. Тогда в добавление к низкому E-value штраф за существаование гэпа был увеличен до 13, а сам поиск проводился по алгоритму DELTA-BLAST. Только тогда удалось добиться значительного уменьшения результатов поиска: 1422.