Поиск в BLAST
Задание 1
Проводился поиск по megablast последовательности из 6-го практикума. Надо отметить, что в 6-м практикуме, где требовалось восстановить последовательность по хроматограмме, части хроматограммы для одной цепочки не было, поэтому последовательность пришлось довольно серьёзно обрезать и консенсусная последовательность вышла короче! Это могло повлиять на достоверность результатов поиска в BLAST. Параметры поиска - по умолчанию.
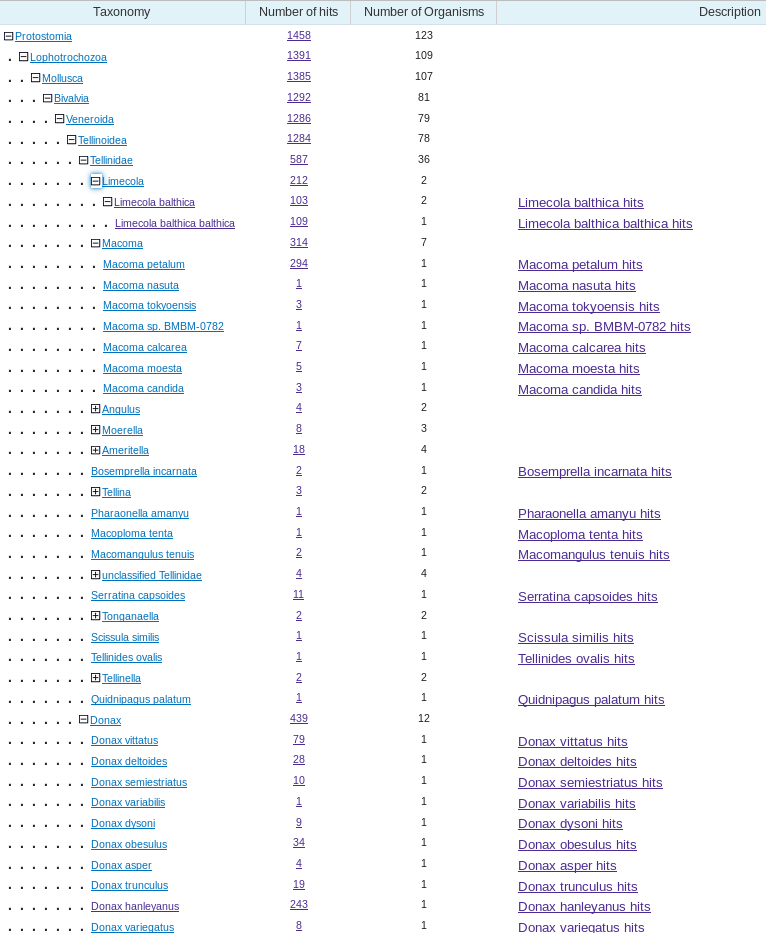
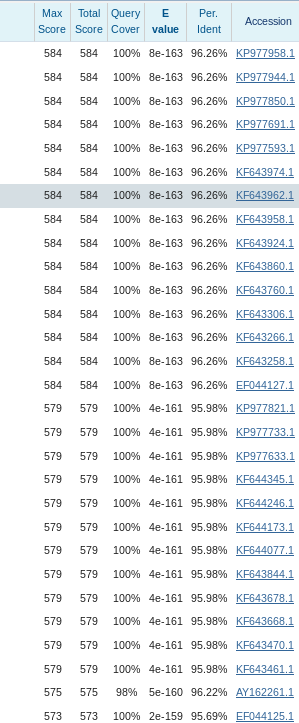
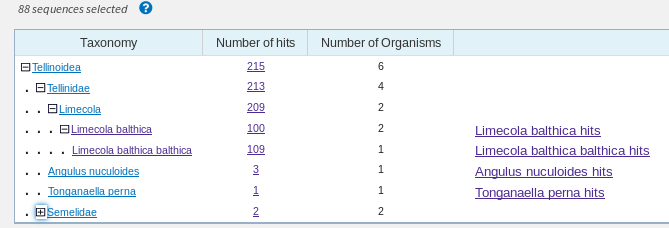
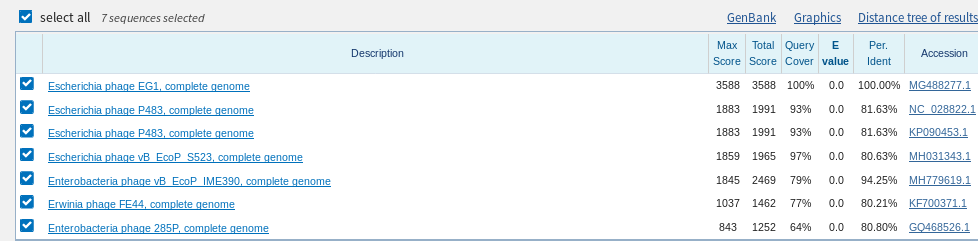
Для найденных 100 последовательностей Identity оказался > 90%, причём первые 15 находок - идентичны с E-Value 96.26%(Рис.1). Все они кодируют субъединицу 1 цитохромоксидазы (COI) из митохондриального генома. Во вкладке "Taxonomy"
представлены организмы, которым принадлежат последрвательности. Оказалось, что все - из генома моллюска вида Limecola balthica, а некоторые - подвиду Limecola balthica balthica (существует ещё и L. balthica rubra)(Рис. 2,3).
Дополнительные материалы:
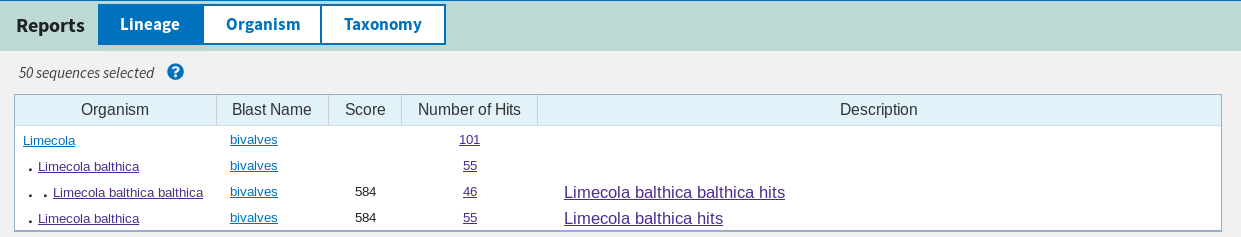
Рис.1 Taxonomy
Рис.2 Выравнивание первых 15 находок в Jalview

Рис.3 Limecola balthica.
Источник
Задание 2. Поиск по трём аналогам BlAST
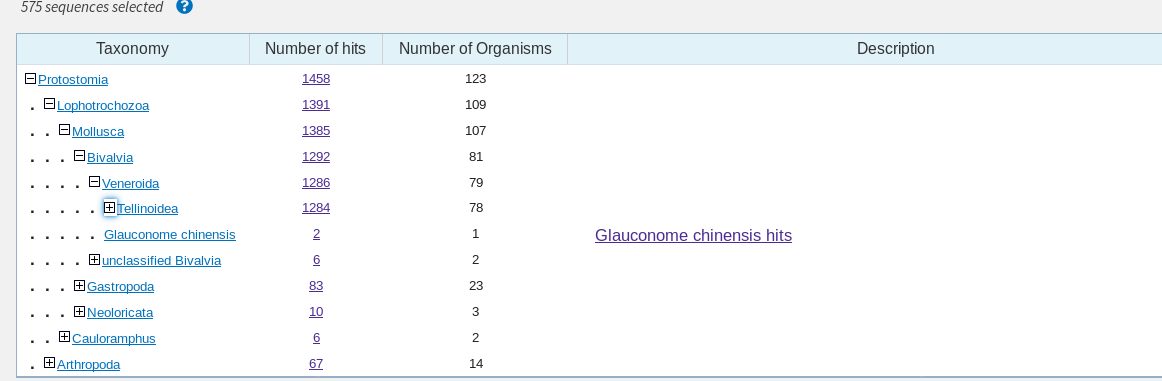
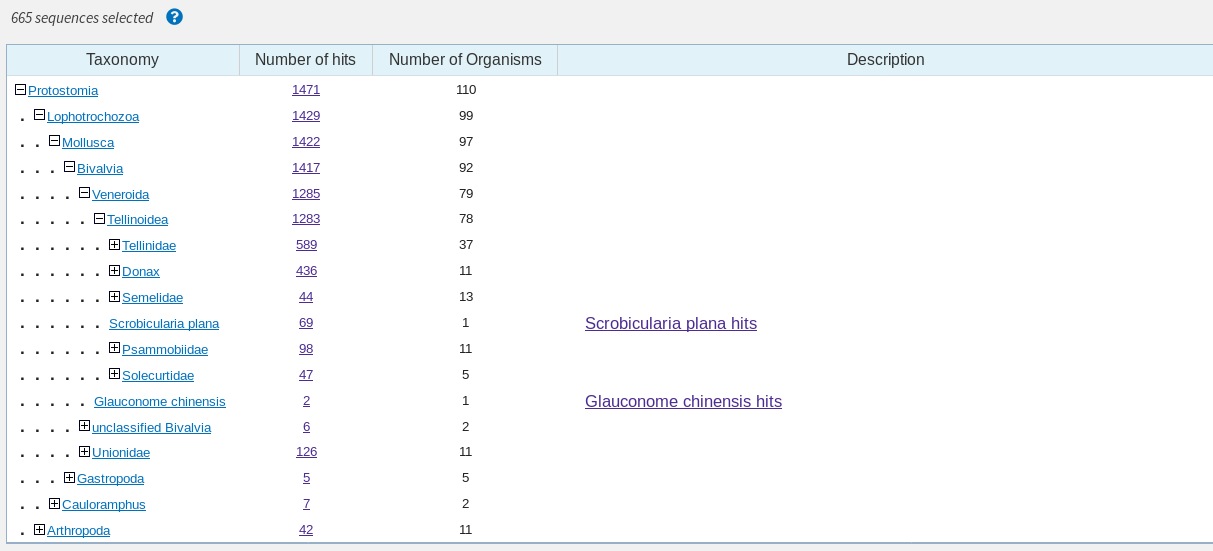
Поиск проводился по megablast, blastn с параметрами по умолчанию и blastn с чувствительными параметрами: wordsize=7, match/mismatch =1/-1. Для сравнения, в двугих двух случаях wordsize=28,11, а match/mismatch=1/-2, 2/-3 соответственно. Ожидалось, что количество находок будет меньше всего в megablast, больше всего - в blastn с чувствительными праметрами. Однако для hitlist size=1000 в megablast найдено 150, в blastn - 1000, а в blastn с чувствительными параметрами - 988, что вводит в некоторое замешательство. На поиск, вероятно, влияло то, что последовательность была неполной. При фильтре E-Value<0.0001, coverage от 90 до 100% "правильный" порядок восстановился - 88,575 и 665 находок соответственно. Identity в случае обоих blastn - от 66%, у megablast - 76%. В то время как в megablast основная масса находок - это вид Limevola bathlica, в blastn можно выделить три близких вида с большим количеством находок - Macoma petalum, Limecola balthicum(предположительной вид) и Donax hanleyanus. Кроме того, blastn с чувствительными настройками нашел значительное количество Униод, которых не нашел обычный blastn, хотя и %Identity у них был самый низкий - около 66%.
| mblast | blastn | lenient blastn | |
| Количество находок | 150 | 1000 | 988 |
| Количество отсортированных находок | 88 | 575 | 665 |


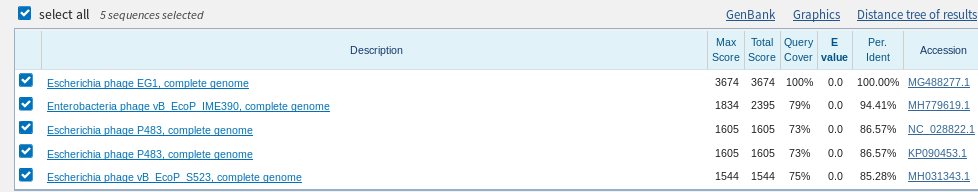
Для дальнейшего поиска была выбрана кодирующая последовательность белка(ATW57755.1) вируса из предыдущего практикума(Escherichia phage EG1). Поиск проводился по тем же алгоритмам. В этом случае была сильно заметна ращзница в работе BLAST с чувствительными параметрами и с обычными(меньше минуты против 7,5). Как и в прошлом примере, параметром, обеспечивающим хорошие находки, оказывается query coverage. При установек QC 60-100 BLAST во всех трёх случаях выводил несколько находок с высоким %Identity и E-Value=0. %Identity падает при снижении чувствительности, а разнообразие организмов - увеличивается, как и ожидалось.Интересно, что mblast не нашёл несколько вариантов с большим процентом Identity и Coverage, которые нашол blastn. Лучшие находки в blastn - различные фаги (первые 5 - фаги Escherichia и Enterobacteria). Есть небольшие различия, например, обычный BLAST нашёл фаг Ervinia, а BLAST с чувствительными параметрами - фаг Salmonella.
| mblast | blastn | lenient blastn | |
| Количество находок | 72 | 226 | 377 |
| Количество находок с coverage 60-100, помимо исходной | 4 | 7 | 15 |
| Разброс % Identity | 84-100 | 67-100 | 60-100 |
| Таксоны | Teseptimavirus(большинство), Synthetic phage- вирусы cellular organisms |
Teseptimavirus(большинство), Przondovirus, неклассифицированные Autographivirinae, Synthetic phage -вирусы cellular organisms |
Teseptimavirus(большинство), Przondovirus, неклассифицированные Autographivirinae, Synthetic phage, Litunavirus - вирусы cellular organisms, artificial sequences |
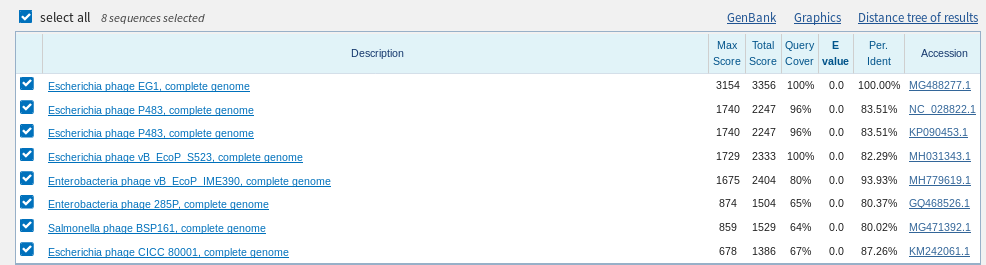
lenient blastn
blastn
mblast
Задание 3
Предлгоалось выбрать 3 любых белка, которые ,вероятно,распространены среди эукариот и посмотреть, сожержатся ли их гомологи в геноме Amoeboaphelidium protococcarum, предварительно скаченного с kodomo и фигурирующего здесь как X5.fasta. Для работы были выбраны: лигаза I, гистон IV и ДНК полимераза альфа. Соответствующие ID были найдены в SwissProt.Поиск проводился в tblastn. Таблица генома была выбрана исходя из того, что было написано в аннотациях к этому организму на сайте NCBI, то есть 6.Результаты - в таблице справа.
Команды работы:
seqret sw:dnli1_rat dnli1.fasta
seqret sw:h4_rat h4.fasta
seqret sw:dpola_rat dpola.fasta
tblastn -query dnli1.fasta -db "X5.fasta" -db_gencode 6 > dnli1.hits
tblastn -query dpola.fasta -db "X5.fasta" -db_gencode 6 > dpola.hits
tblastn -query h4.fasta -db "X5.fasta" -db_gencode 6 > h4.hits
| ID белка | dnli1_rat | h4_rat | dpola_rat |
| Длина последовательности | 918 | 103 | 1451 |
| Количество достоверных находок | 4 | 5 | 5 |
| Разброс bitscore | 114-612 | 40-160 | 86.7-753 |
| Разброс % Identity | 26-53 | 94 | 24-39 |
| Координаты лучшего выравнивания | 285-900 | 22-103 | 338-1446 |
| Покрытие | 0,670 | 0,786 | 0,764 |
| Файл | dnli1.hits | h4.hits | dploa.hits |
Чем последовательность длиннее, тем менее она консервативна, что логично. На консервативность гена гистона могло повлиять то, что гистон - это структурный элемент, а поэтому изменения в нём крайне нежелательны. Как видно из таблицы, для всех белков, вероятно, были найдены гомологи.
Результат работы blastx с фильтром % Identity: 60-100
Задание 4
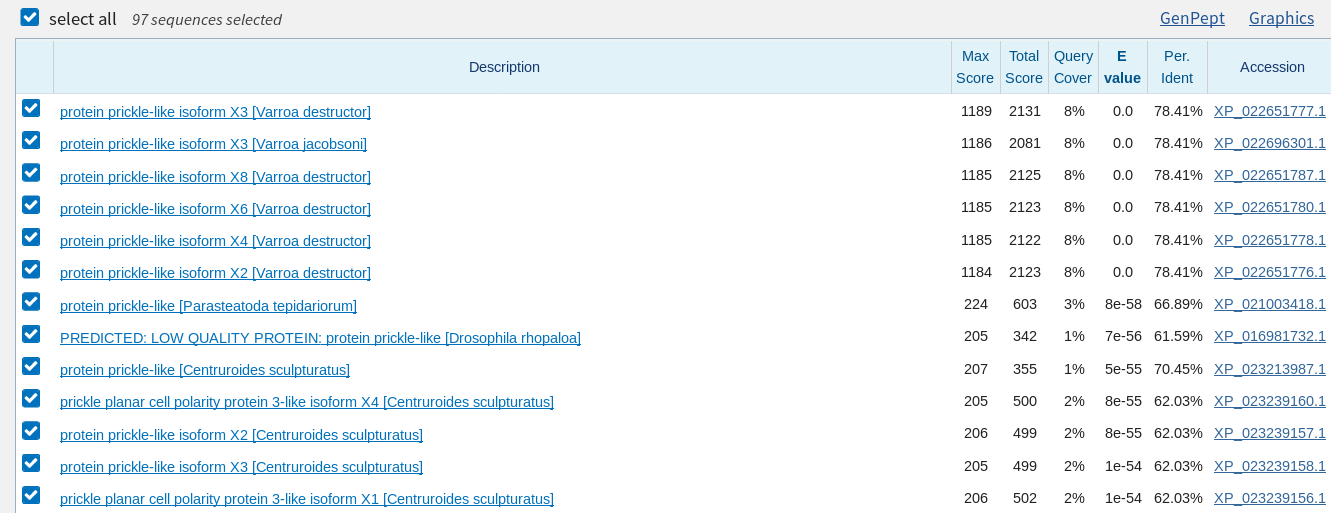
Требоавлось взять неаннотированный геном, выбрать один скэффолд или контиг, найти наиболее достоверный ген белка и понять, соответственно, какой белок закодирован. Удачным образом, в геноме, взятом мною в практикуме 7,белки не аннотированы, поэтому я взяла один из контигов из генома Varroha jacobsoni около 50 килобаз. Поиск проводился по blastx. Лучшие находки указывают на то, что закодирован изоформ белка-шипа (prickle protein) - рецептора ядерной транслокации.Ссылка на гомолог для Drosophila melanogaster. Самая лучшая находка - из генома Varroa jacobsoni и очень близкого ему виду Varroa destructor, что неудивительно.
Range 1: 179 to 933
Score:1186 bits(3067), Expect:0.0,
Method:Compositional matrix adjust.,
Identities:650/829(78%), Positives:665/829(80%), Gaps:91/829(10%)
Query 19994 LKPRCSAC-DEIIFADECTEAE----GLTWHMSHFCCYECDQQLGGQRYIMRDN------ 19848
L+ C+ C D II D A + WH + F C C + L Y M+D
Sbjct 179 LQIVCACCNDYIIGGDMAVFASRSGPNIGWHPACFQCSICKELLVDLIYYMKDGALYCGR 238
Query 19847 ------RPFCLACFDAIFAG*yrtyv*tiyiYSLYTMRLCPtalaasaa*fapttsSYTR 19686
+P C AC + IFA + S + C L
Sbjct 239 HHAESLKPRCSACDEIIFADECTEAEGLTWHMSHFCCYECDQQLGGQRY----------- 287
Query 19685 *YVSLSIAILFKLITLYMYVCIITEFCDTCGGPVGVDQGQMSLDGQHWHATEQCFRCSCC 19506
I + L + I EFCDTCGGPVGVDQGQMSLDGQHWHATEQCFRCSCC
Sbjct 288 ------IMRDNRPFCLACFDAIFAEFCDTCGGPVGVDQGQMSLDGQHWHATEQCFRCSCC 341
Query 19505 KMSLLGRPFLPKKGLIYCSHECSRSKFSTCSGQHLHEYRYYNLLLCTAPFAASNSHPTRN 19326
KMSLLGRPFLPKKG L+ C+ + SNS
Sbjct 342 KMSLLGRPFLPKKG----------------------------LIYCSHECSRSNS----- 368
Query 19325 GPRFLPLTPHEYKAYDTLNIAGNSNSRAHRSNSVRNFASQNSSTENGPTGSSALSPKDAV 19146
NSRAHRSNSVRNFASQNSSTENGPTGSSALSPKDAV
Sbjct 369 ------------------------NSRAHRSNSVRNFASQNSSTENGPTGSSALSPKDAV 404
Query 19145 SGMHTPTGLFVPGISNVPETVRKVTPGSFSPLANSSSDNVHSQRMLERNYSPSIMSNHSE 18966
SGMHTPTGLFVPGISNVPETVRKVTPGSFSPLANSSSDNVHSQRMLERNYSPSIMSNHSE
Sbjct 405 SGMHTPTGLFVPGISNVPETVRKVTPGSFSPLANSSSDNVHSQRMLERNYSPSIMSNHSE 464
Query 18965 PLRNFGASFSPNTSSEKSFNASQQIALIDPRGqqhqqqqilqqhqaqqqqqhqmlvqqtv 18786
PLRNFGASFSPNTSSEKSFNASQQIALIDPRGQQHQQQQILQQHQAQQQQQHQMLVQQTV
Sbjct 465 PLRNFGASFSPNTSSEKSFNASQQIALIDPRGQQHQQQQILQQHQAQQQQQHQMLVQQTV 524
Query 18785 aQPQRPGPPPYNVAAYNSNGLPPPISRLQSQLRGCQGNIEYTPQHKTTHTAPNVLSPMAR 18606
AQPQRPGPPPYNVAAYNSNGLPPPISRLQSQLRGCQGNIEYTPQHKTTHTAPNVLSPMAR
Sbjct 525 AQPQRPGPPPYNVAAYNSNGLPPPISRLQSQLRGCQGNIEYTPQHKTTHTAPNVLSPMAR 584
Query 18605 QSPNGINGRHEMALPNAGKNSPSNCTQAQRILGDVSSGSVSPAARNSPAQGRRDIQKPQQ 18426
QSPNGINGRHEMALPNAGKNSPSNCTQAQRILGDVSSGSVSPAARNSPAQGRRDIQKPQQ
Sbjct 585 QSPNGINGRHEMALPNAGKNSPSNCTQAQRILGDVSSGSVSPAARNSPAQGRRDIQKPQQ 644
Query 18425 PTVAGLQHSGSSQGGPMRSLSPTLPRREGSPGLGRRSLTDLSSNSLPRTTSRSGPSSLID 18246
PTVAGLQHSGSSQGGPMRSLSPTLPRREGSPGLGRRSLTDLSSNSLPRTTSRSGPSSLID
Sbjct 645 PTVAGLQHSGSSQGGPMRSLSPTLPRREGSPGLGRRSLTDLSSNSLPRTTSRSGPSSLID 704
Query 18245 TPTKYKQVYSHQGVEGSPQRKHMADFSLAEVNFGAKQKTKLTREGSLNEHYANLQRSGSV 18066
TPTKYKQVYSHQGVEGSPQRKHMADFSLAEVNFGAKQKTKLTREGSLNEHYANLQRSGSV
Sbjct 705 TPTKYKQVYSHQGVEGSPQRKHMADFSLAEVNFGAKQKTKLTREGSLNEHYANLQRSGSV 764
Query 18065 NALPEVEYSNTLLRRRPSEHFHGSTLPRNFSSGHIQMTQAAHCDNLESNLYSNYPASQDL 17886
NALPEVEYSNTLLRRRPSEHFHGSTLPRNFSSGHIQMTQAAHCDNLESNLYSNYPASQDL
Sbjct 765 NALPEVEYSNTLLRRRPSEHFHGSTLPRNFSSGHIQMTQAAHCDNLESNLYSNYPASQDL 824
Query 17885 YTNGPGQMHNGHVQHLNQSQEIYSNPQDFQHVADQTYDNVPSVRSIVYKNGRQEEAMNER 17706
YTNGPGQMHNGHVQHLNQSQEIYSNPQDFQHVADQTYDNVPSVRSIVYKNGRQEEAMNER
Sbjct 825 YTNGPGQMHNGHVQHLNQSQEIYSNPQDFQHVADQTYDNVPSVRSIVYKNGRQEEAMNER 884
Query 17705 DMAVTSAQAMTPSTRRREPLDMSDLCLKDLLAGSDQVFVEVVQEIQNGP 17559
DMAVTSAQAMTPSTRRREPLDMSDLCLKDLLAGSDQVFVEVVQEIQNGP
Sbjct 885 DMAVTSAQAMTPSTRRREPLDMSDLCLKDLLAGSDQVFVEVVQEIQNGP 933
Range 2: 921 to 1233
Score:298 bits(763), Expect:4e-79,
Method:Compositional matrix adjust.,
Identities:300/313(96%), Positives:306/313(97%), Gaps:4/313(1%)
Query 17355 ITIQILRAFQ----VHRTSLQAASAGPGSVRQNFSLPLMNPHRPSVQPPEALELESPITP 17188
+ +++++ Q VHRTSLQAASAGPGSVRQNFSLPLMNPHRPSVQPPEALELESPITP
Sbjct 921 VFVEVVQEIQNGPSVHRTSLQAASAGPGSVRQNFSLPLMNPHRPSVQPPEALELESPITP 980
Query 17187 QQLPSPPILKSIGPPNGILQRQVKTRAEVHTEDRVHPsderrsmrresksKVRFDPSLGH 17008
QQLPSPPILKSIGPPNGILQRQVKTRAEVHTEDRVHPSDERRSMRRESKSKVRFDPSLGH
Sbjct 981 QQLPSPPILKSIGPPNGILQRQVKTRAEVHTEDRVHPSDERRSMRRESKSKVRFDPSLGH 1040
Query 17007 ANSDEEQKSAsrnttrrhrrrhrrrssssesEGECENraekkstmssssssslssssark 16828
ANSDEEQKSASRNTTRRHRRRHRRRSSSSESEGECENRAEKKSTMSSSSSSSLSSSSARK
Sbjct 1041 ANSDEEQKSASRNTTRRHRRRHRRRSSSSESEGECENRAEKKSTMSSSSSSSLSSSSARK 1100
Query 16827 hgsggggsgggehasrhrsrsEDRCDGESSRERKTRKSLrssshgsrsrsgssshksrss 16648
HGSGGGGSGGGEHASRHRSRSEDRCDGESSRERKTRKSLRSSSHGSRSRSGSSSHKSRSS
Sbjct 1101 HGSGGGGSGGGEHASRHRSRSEDRCDGESSRERKTRKSLRSSSHGSRSRSGSSSHKSRSS 1160
Query 16647 sksrrdrddsscstcestestsstedEAEIYRLPERREYGGVRINYVQSSTLAAARQKAV 16468
SKSRRDRDDSSCSTCESTESTSSTEDEAEIYRLPERREYGGVRINYVQSSTLAAARQKAV
Sbjct 1161 SKSRRDRDDSSCSTCESTESTSSTEDEAEIYRLPERREYGGVRINYVQSSTLAAARQKAV 1220
Query 16467 ASQRAQNKNCLVQ 16429
ASQRAQNKNCLVQ
Sbjct 1221 ASQRAQNKNCLVQ 1233
Range 3: 151 to 305
Score:273 bits(697), Expect:5e-71,
Method:Compositional matrix adjust.,
Identities:126/156(81%), Positives:132/156(84%), Gaps:6/156(3%)
Query 20261 KRMFLFLVKHVRGGILKATLFQAIVCDVEFQ-----CNDYIIGGDMAVFASRSGPNIGWH 20097
+ + F + R + + T+ Q V Q CNDYIIGGDMAVFASRSGPNIGWH
Sbjct 151 RELRQFSAQRKRDSLGRGTVRQLPVSP-HLQIVCACCNDYIIGGDMAVFASRSGPNIGWH 209
Query 20096 PACFQCSICKELLVDLIYYMKDGALYCGRHHAESLKPRCSACDEIIFADECTEAEGLTWH 19917
PACFQCSICKELLVDLIYYMKDGALYCGRHHAESLKPRCSACDEIIFADECTEAEGLTWH
Sbjct 210 PACFQCSICKELLVDLIYYMKDGALYCGRHHAESLKPRCSACDEIIFADECTEAEGLTWH 269
Query 19916 MSHFCCYECDQQLGGQRYIMRDNRPFCLACFDAIFA 19809
MSHFCCYECDQQLGGQRYIMRDNRPFCLACFDAIFA
Sbjct 270 MSHFCCYECDQQLGGQRYIMRDNRPFCLACFDAIFA 305
Range 4: 101 to 187
Score:182 bits(463), Expect:7e-43,
Method:Compositional matrix adjust.,
Identities:86/87(99%), Positives:86/87(98%), Gaps:0/87(0%)
Query 20634 VHLYFSSLPEDKIPYVNSVGEKYRMKQLIQQLPAHDSDVRHCTSLSEEECRELRQFSAQR 20455
VHLYFSSLPEDKIPYVNSVGEKYRMKQLIQQLPAHDSDVRHCTSLSEEECRELRQFSAQR
Sbjct 101 VHLYFSSLPEDKIPYVNSVGEKYRMKQLIQQLPAHDSDVRHCTSLSEEECRELRQFSAQR 160
Query 20454 KRDSLGRGTVRQLPVSPHLQIVCACVN 20374
KRDSLGRGTVRQLPVSPHLQIVCAC N
Sbjct 161 KRDSLGRGTVRQLPVSPHLQIVCACCN 187
Range 5: 1 to 105
Score:140 bits(354), Expect:6e-30,
Method:Compositional matrix adjust.,
Identities:101/105(96%), Positives:104/105(99%), Gaps:0/105(0%)
Query 40601 MAEVERLMLPAPPQHMMtsptssgstaavgasgasgtatasaggavsnsSQQATTGGQNG 40422
MAEVERLMLPAPPQHMMTSPTSSGSTAAVGASGASGTATASAGGAVSNSSQQATTGGQNG
Sbjct 1 MAEVERLMLPAPPQHMMTSPTSSGSTAAVGASGASGTATASAGGAVSNSSQQATTGGQNG 60
Query 40421 HGPQQAPSPEMGSGHRQSDDDSGCALEEYTWVPQGLKPEQVRIFY 40287
HGPQQAPSPEMGSGHRQSDDDSGCALEEYTWVPQGLKPEQV +++
Sbjct 61 HGPQQAPSPEMGSGHRQSDDDSGCALEEYTWVPQGLKPEQVHLYF 105