Поиск белков, гомологичных CLPX_ECOLI и построение дерева
C kodomo из директории '/P/y18/term4/Proteomes' были скачены полные протеомы бактерий из первого практикума для создания базы данных, по которой проводился поиск гомологов CLPX_ECOLI в standalone blast. Команда запуска:
blastp -query clpx_ecoli.fasta -db db.fasta -evalue 0.001 -outfmt 7 -out homol.out
Файл homol.out доступен для скачивания. Было получено 43 находки, но только 36 белков. По их идентификаторам были скачены белковые последовательности с сайта Uniprot и выравнены программой muscle на сервере kodomo. Результат был загружен в программу MEGA для построения дерева методом минимальной эволюции. Его можно увидеть на Рис.1, ортологичные группы, содержащие более 3х гомологов, покрашены в разные цвета. На Рис.2 эти же группы "схлопнуты". Ортологами являются пары CLPX_GEOKA - CLPX_MOOTA, HSLU_LISMO - HSLU_BACAN. Парологами - пары CLPX_BACAN - HSLU_BACAN, Y979_STAEQ - CLPX_STAEQ, CLPL_STAA8 - HSLU_STAA8. Ссылка на дерево в newick-формате

Рис.1 Результат работы MEGA, дерево гомологов CLPX_ECOLI, построенное по методу минимальной эволюции
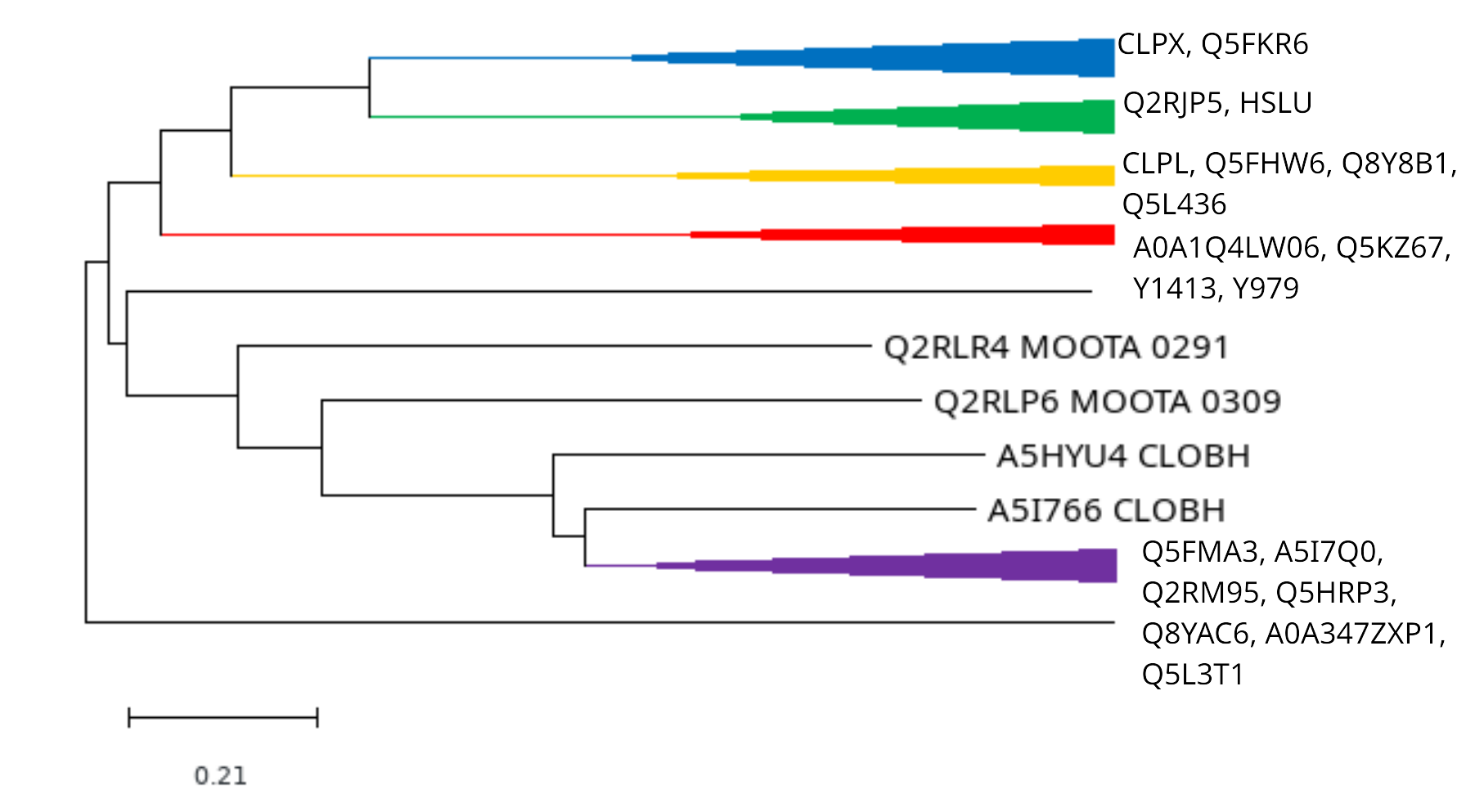
Рис.2 Дерево со схлопнутыми ортологичными группами