Главная (Main)
Занятие 1
Отобранные бактерии
| Название | Мнемоника |
| Bacillus anthracis | BACAN |
| Bacillus subtilis | BACSU |
| Enterococcus faecalis | ENTFA |
| Lactobacillus acidophilus | LACAC |
| Lactobacillus delbrueckii | LACDA |
| Lactococcus lactis | LACLM |
| Streptococcus pyogenes | STRP1 |
| Streptococcus pneumoniae | STRPN |
Скобочная формула дерева
(((((STRP1,STRPN),LACLM),ENTFA),(LACDA,LACAC)),(BACAN,BACSU));
Изображение дерева
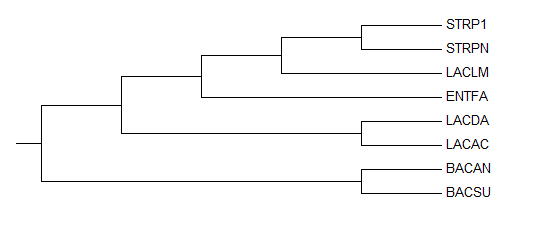
Ветви дерева
Дерево содержит 5 нетривиальных ветвей:
1) {BACAN, BACSU} против {LACDA, LACAC, ENTFA, LACLM, STRP1, STRPN}
2) {BACAN, BACSU, LACDA, LACAC} против {ENTFA, LACLM, STRP1, STRPN}
3) {BACAN, BACSU, LACDA, LACAC, ENTFA} против {LACLM, STRP1, STRPN}
4) {BACAN, BACSU, LACDA, LACAC, ENTFA, LACLM} против {STRP1, STRPN}
5) {LACDA, LACAC} против {BACAN,BACSU,ENTFA, LACLM, STRP1, STRPN}
|
Занятие 2
1. Таксоны отобранных бактерий
| Название | Таксономия |
| Bacillus anthracis | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group |
| Bacillus subtilis | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group |
| Enterococcus faecalis | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| Lactobacillus acidophilus | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Lactobacillus delbrueckii | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Lactococcus lactis | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Lactococcus |
| Streptococcus pyogenes | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
| Streptococcus pneumoniae | Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
{BACAN, BACSU} против {LACDA, LACAC, ENTFA, LACLM, STRP1, STRPN} выделяет порядок Bacillales от порядка Lactobacillales
{BACAN, BACSU, LACDA, LACAC, ENTFA} против {LACLM, STRP1, STRPN} выделяет семейство Streptococcaceae
{LACDA, LACAC} против {BACAN,BACSU,ENTFA, LACLM, STRP1, STRPN} выделяет семейство Lactobacillaceae
2.
Выбрала функцию - Фактор инициации трансляции 2 ( мнемоника - IF2)
3.
Создала выравнивание отобранных белков программой muscle
5.
Программа выдала одно дерево:
+-----IF2_ENTFA
+--------5
! ! +--IF2_BACSU
! +--4
+--3 +--IF2_BACAN
! !
! ! +--IF2_STRPN
! ! +--7
+--2 +--------6 +--IF2_STRP1
! ! !
! ! +-----IF2_LACLM
1 !
! +-----------------IF2_LACDA
!
+--------------------IF2_LACAC
Его скобочная формула: ((((IF2_ENTFA,(IF2_BACSU,IF2_BACAN)),((IF2_STRPN,IF2_STRP1),IF2_LACLM)),IF2_LACDA),IF2_LACAC)
В полученном дереве отсутствует ветвь:
{BACAN, BACSU, LACDA, LACAC} против {ENTFA, LACLM, STRP1, STRPN}
Присутствует ветвь:
{ENTFA, BACSU, BACAN} против {LACDA, LACAC, LACLM, STRP1, STRPN}
6.
IF2_LACAC 0.000000 0.377576 0.878548 0.685297 0.696291 0.668801 0.835311 0.802408
IF2_LACDA 0.377576 0.000000 0.816650 0.678457 0.702378 0.663696 0.775212 0.784278
IF2_LACLM 0.878548 0.816650 0.000000 0.642859 0.689511 0.608363 0.643630 0.603738
IF2_BACAN 0.685297 0.678457 0.642859 0.000000 0.319598 0.515598 0.590458 0.569989
IF2_BACSU 0.696291 0.702378 0.689511 0.319598 0.000000 0.577003 0.633702 0.611892
IF2_ENTFA 0.668801 0.663696 0.608363 0.515598 0.577003 0.000000 0.542306 0.527410
IF2_STRP1 0.835311 0.775212 0.643630 0.590458 0.633702 0.542306 0.000000 0.401616
IF2_STRPN 0.802408 0.784278 0.603738 0.569989 0.611892 0.527410 0.401616 0.000000
Расстояния отклоняются от ультраметричности:
d(LACAC, LACLM) > d(LACLM, STRPN), но (LACAC, STRPN) не равно (LACAC, LACLM)
d(LACAC,STRPN)=0.802408
d(LACAC,LACLM)=0.878548
Отклонение от ультраметричности: (0.878548-0.802408)/0.878548=0.086666 %
Зато выполняется свойство аддитивности:
d(BACSU, BACAN)+d(STRPN, STRP1)=0,319598+0,401616=0,721214
d(BACSU, STRPN)+d(BACAN, STRP1)=0,611892+0,590458=1,20235
d(BACSU, STRP1)+d(BACAN, STRPN)=0,633702+0,569989=1,203691
7. Реконструкция дерева программой fneighbor.
По алгоритму Neighbor-Joining:
+----------IF2_LACDA
!
! +-------IF2_BACAN
! +------2
! ! +---------IF2_BACSU
1-------------4
! ! +------------IF2_ENTFA
! +--5
! ! +-------------------IF2_LACLM
! +-6
! ! +------------IF2_STRP1
! +---3
! +-----------IF2_STRPN
!
+-----------IF2_LACAC
По алгоритму UPGMA:
+----------IF2_LACAC
+----------2
! +----------IF2_LACDA
--7
! +------------------IF2_LACLM
! !
+--6 +--------IF2_BACAN
! +-------1
! ! +--------IF2_BACSU
+-5
! +---------------IF2_ENTFA
+-4
! +-----------IF2_STRP1
+---3
+-----------IF2_STRPN
Сравнивая 3 дерева, полученные программой fprotpars, fneighbor (Neighbor-Joining и UPGMA), наиболее
правильным оказалось дерево, полученное алгоритмом Neighbor-Joining (полностью совпадает с првильным),
не смотря на то, что UPGMA даёт укоренённое дерево, а fprotpars - несколько вариантов. Самым неточным
оказался алгоритм UPGMA. В дереве UPGMA имеются 2 ветви, отсутствующие в правильном дереве: {LACAC, LACDA, LACLM}
против {BACAN,BACSU,ENTFA, STRP1, STRPN} и {ENTFA, STRP1, STRPN} против {BACAN, BACSU, LACAC, LACDA, LACLM}
и отсутствуют 2 ветви, присутствующие в правильном дереве: {LACLM, STRP1, STRPN} против {BACAN, BACSU, LACAC, LACDA, ENTFA}
и {LACLM, STRP1, STRPN, ENTFA} против {BACAN, BACSU, LACAC, LACDA}
© Лунева Анна
