Практикум 7. Работа с банками нуклеотидных последовательностей
Задание 1. Сборка генома Danio rerio
Danio rerio (данио рерио), или zebra fish, - это важный модельный объект для биологических исследований, в частности в области биологии развития и функций генов. Поэтому геном данио рерио представляет большой интерес. Всего есть 4 сборки генома этого организма. В этом отчете я опишу сборку GCA_000767325.1 WGS31
Число скаффолдов - 32,031
Число контигов - 119,179
N50 - 24,925
L50 - 16,539
Число аннотированных белков - 57100
Статьи, посвященной именно этой сборке, нет.
Ссылка на контиг 0000000 (Contig_0000000)

Задание 2. Описание ключей, используемых в табличах особенностей
Ключ 1. CDS. Идентифицирует белок кодирующую последовательность (protein-coding sequence), то есть последовательность нуклеотидов, которой соответствует последовательность аминокислот в белке, плюс стоп-кодон.
Опциональные характеритики:
/allele="text"
/artificial_location="[artificial_location_value]"
/citation=[number]
/codon_start=<1 or 2 or 3>
/db_xref=":"
/EC_number="text"
/exception="[exception_value]"
/experiment="[CATEGORY:]text"
/function="text"
/gene="text"
/gene_synonym="text"
/inference="[CATEGORY:]TYPE[ (same species)][:EVIDENCE_BASIS]"
/locus_tag="text" (single token)
/map="text"
/note="text"
/number=unquoted text (single token)
/old_locus_tag="text" (single token)
/operon="text"
/product="text"
/protein_id=""
/pseudo
/pseudogene="TYPE"
/ribosomal_slippage
/standard_name="text"
/translation="text"
/transl_except=(pos:,aa:)v
/transl_table =
/trans_splicing
Пример из help-a:
![]()
Ключ 2. tRNA. Идентефицирует последовательность, кодирующую транспортную РНК. Из характеристик этого ключа понятно, какую аминокислоту переносит данная тРНК.
Опциональные характеристики:
/allele="text"
/anticodon=(pos:,aa:,seq:)
/citation=[number]
/db_xref=":"
/experiment="[CATEGORY:]text"
/function="text"
/gene="text"
/gene_synonym="text"
/inference="[CATEGORY:]TYPE[ (same species)][:EVIDENCE_BASIS]"
/locus_tag="text" (single token)
/map="text"
/note="text"
/old_locus_tag="text" (single token)
/operon="text"
/product="text"
/pseudo
/pseudogene="TYPE"
/standard_name="text"
/trans_splicing
Пример из help-a:
![]()
Ключ 3. assembly_gap. Идентефицирует гэпы между двумя компонентами геномной или транскриптомной сборки.
Обязательные характеристики:
/estimated_length=unknown or
/gap_type="TYPE"
/linkage_evidence="TYPE" (Note: Mandatory only if the
/gap_type is "within scaffold" or "repeat within
scaffold".If there are multiple types of linkage_evidence
they will appear as multiple /linkage_evidence="TYPE"
qualifiers. For all other types of assembly_gap
features, use of the /linkage_evidence qualifier is
invalid.)
Mandatory qualifiers under assembly_gap feature for transcriptome
shotgun assemblies (TSA):
/estimated_length=
/gap_type="within scaffold" and /linkage_evidence="TYPE" where TYPE
can not be "unspecified";
Пример (идентефикатор генбанка RAZU01000074.1):
![]()
Ключ 4. misc_feature. Интересный с биологической точки зрения регион, который не может быть описан ни одним другим ключом. Новая или редкая особенность.
Опциональные характеристики:
/allele="text"
/citation=[number]
/db_xref=":"
/experiment="[CATEGORY:]text"
/function="text"
/gene="text"
/gene_synonym="text"
/inference="[CATEGORY:]TYPE[ (same species)][:EVIDENCE_BASIS]"
/locus_tag="text" (single token)
/map="text"
/note="text"
/number=unquoted text (single token)
/old_locus_tag="text" (single token)
/phenotype="text"
/product="text"
/pseudo
/pseudogene="TYPE"
/standard_name="text"
Пример (идентефикатор генбанка AC068946.4):
![]()
Ключ 5. repeat_region. Идентефицирует участок генома, содержащий повтояющиеся фрагменты.
Опциональные характеристики:
/allele="text"
/citation=[number]
/db_xref=":"
/experiment="[CATEGORY:]text"
/function="text"
/gene="text"
/gene_synonym="text"
/inference="[CATEGORY:]TYPE[ (same species)][:EVIDENCE_BASIS]"
/locus_tag="text" (single token)
/map="text"
/note="text"
/old_locus_tag="text" (single token)
/rpt_family="text"
/rpt_type=
/rpt_unit_range=
/rpt_unit_seq="text"
/satellite="[:][ ]"
/standard_name="text"
Пример (идентефикатор генбанка AC068946.4):
![]()
Ключ 6. misc_RNA. Любой транскрипт или РНК-продукт, которые не может быть обозначен другим РНК ключом(prim_transcript, precursor_RNA, mRNA,
5'UTR, 3'UTR, exon, CDS, sig_peptide, transit_peptide,
mat_peptide, intron, polyA_site, ncRNA, rRNA and tRNA)
Опциональные характеристики:
/allele="text"
/citation=[number]
/db_xref=":"
/experiment="[CATEGORY:]text"
/function="text"
/gene="text"
/gene_synonym="text"
/inference="[CATEGORY:]TYPE[ (same species)][:EVIDENCE_BASIS]"
/locus_tag="text" (single token)
/map="text"
/note="text"
/old_locus_tag="text" (single token)
/operon="text"
/product="text"
/pseudo
/pseudogene="TYPE"
/standard_name="text"
/trans_splicing
Пример (идентефикатор генбанка CP002687.1):
![]()
Ключ 7. exon. Идентефицирует участок генома, кодирующий сплайсируемый участток мРНК, рРНК или тРНК. Может содержать 5'-нетранслируемые области, белок кодирующие последовательности и 3'-нетранслируемые области.
Опциональные характеристики:
/allele="text"
/citation=[number]
/db_xref=":"
/EC_number="text"
/experiment="[CATEGORY:]text"
/function="text"
/gene="text"
/gene_synonym="text"
/inference="[CATEGORY:]TYPE[ (same species)][:EVIDENCE_BASIS]"
/locus_tag="text" (single token)
/map="text"
/note="text"
/number=unquoted text (single token)
/old_locus_tag="text" (single token)
/product="text"
/pseudo
/pseudogene="TYPE"
/standard_name="text"
/trans_splicing
Пример (идентефикатор генбанка UNSH01000041.1)
![]()
Задание 3. Состояние проекта 100k pathogen genome project.
Проект был запущен в июле 2012 года профессором университета University of California, Davis (США). Цель проекта - отсеквенировать 100.000 проектов патогенных микроорганизмов. Больше информации о проекте, включая ссылки на все публикации, можно найти по ссылке. Полное описание проекта в 2017 году было также опубликовано в формате статьи. На момент публикации (13 июля 2017) были отсеквенировать геномы 6 микроорганизмов. Дата окончания проекта не обозначена. Самая недавняя публикация по проекту была сделана в августе 2017 года
Задание 4. Таблица митохондриальных генов
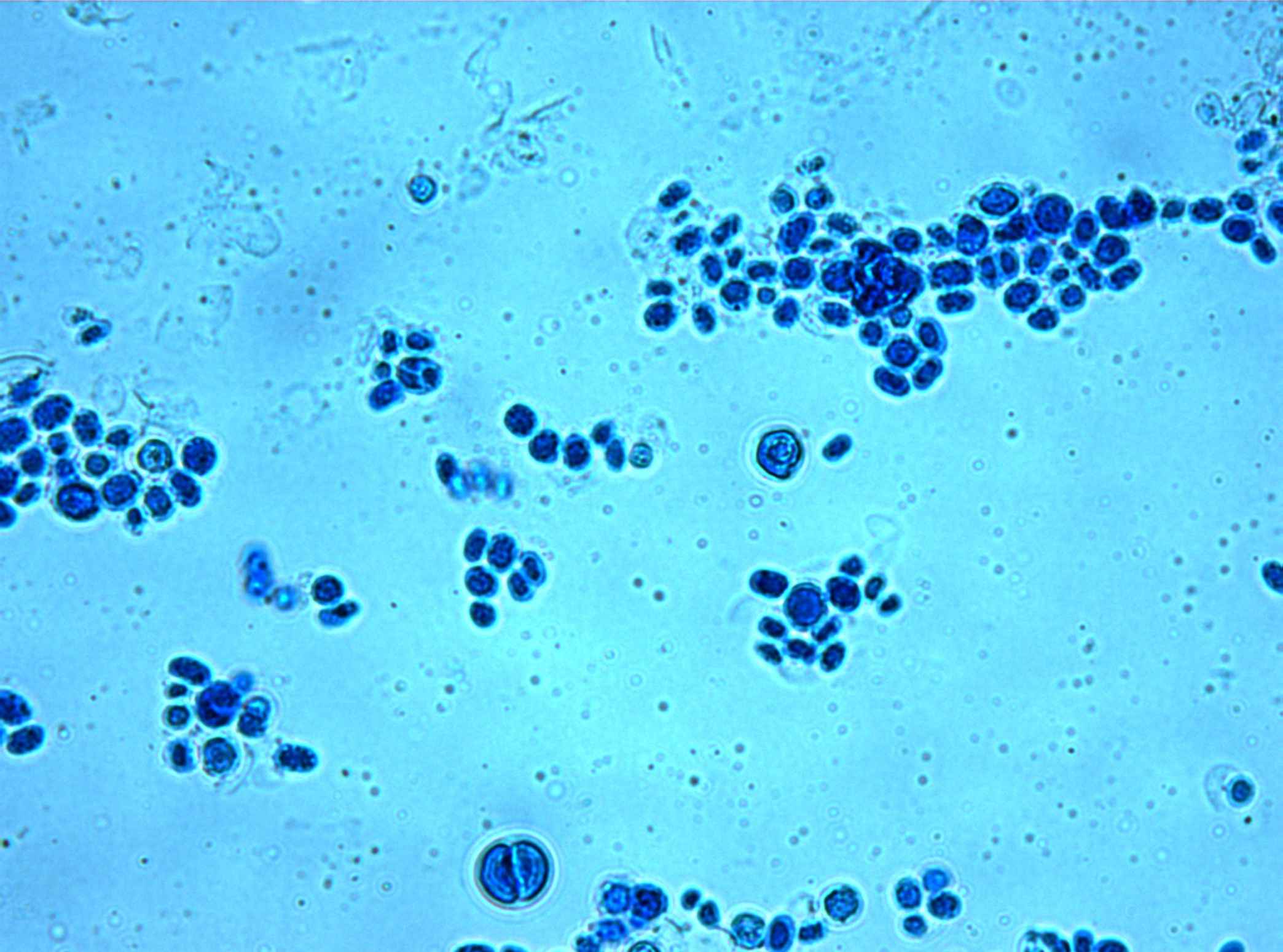
Текст запроса: tax_tree(3041) AND mol_type="genomic DNA" AND topology="CIRCULAR" AND organelle="mitochondrion". Нашлось 3 записи в update и 94 в release. Я выбрала геном митохондрии прототеки (Prototheca zopfii strain SAG 2063), AC: MF197533.1. Ниже приведена фотография этого организма.
По ссылке Вы найдете таблицу с данными о митохондриальных генах прототеки зопфии.
