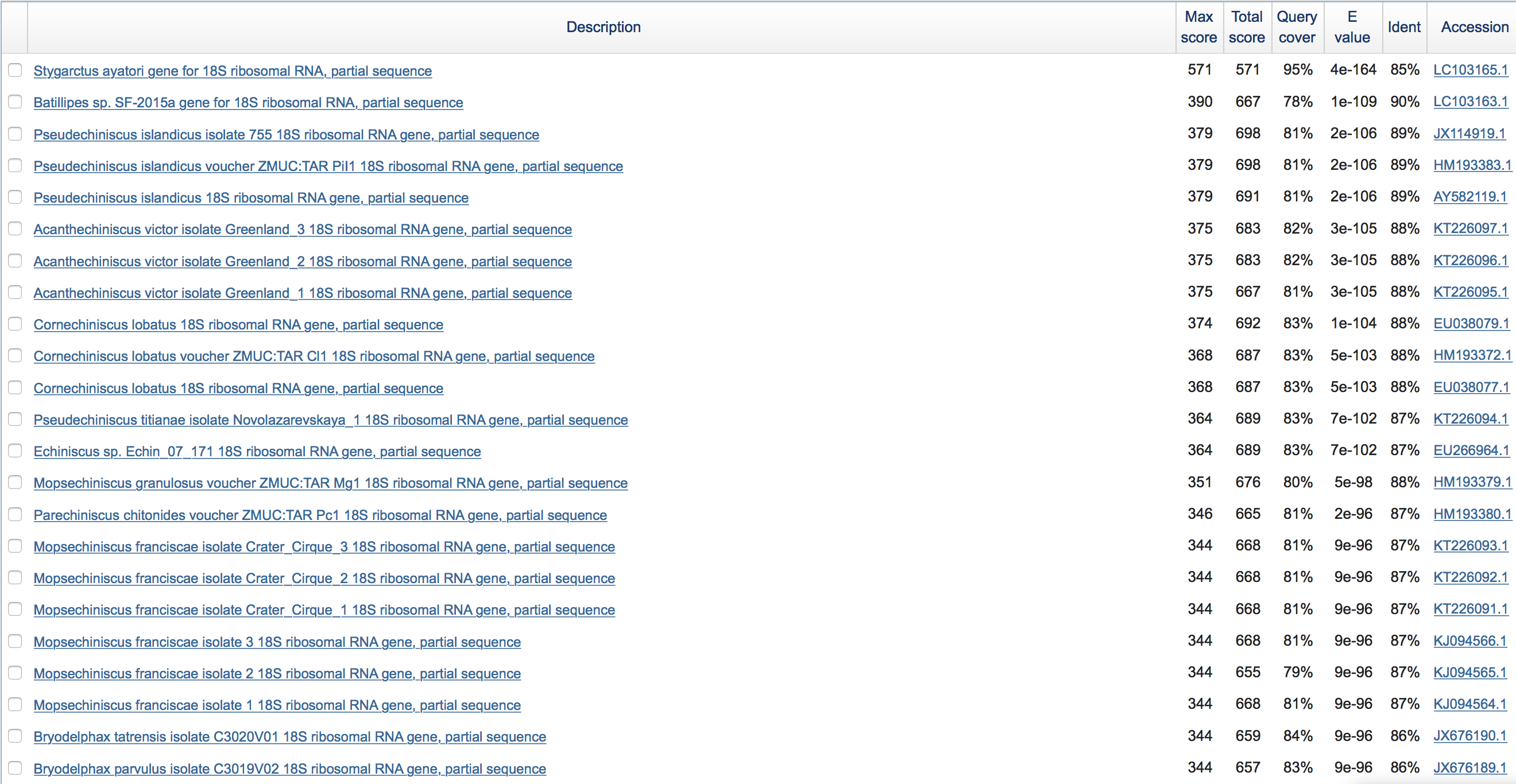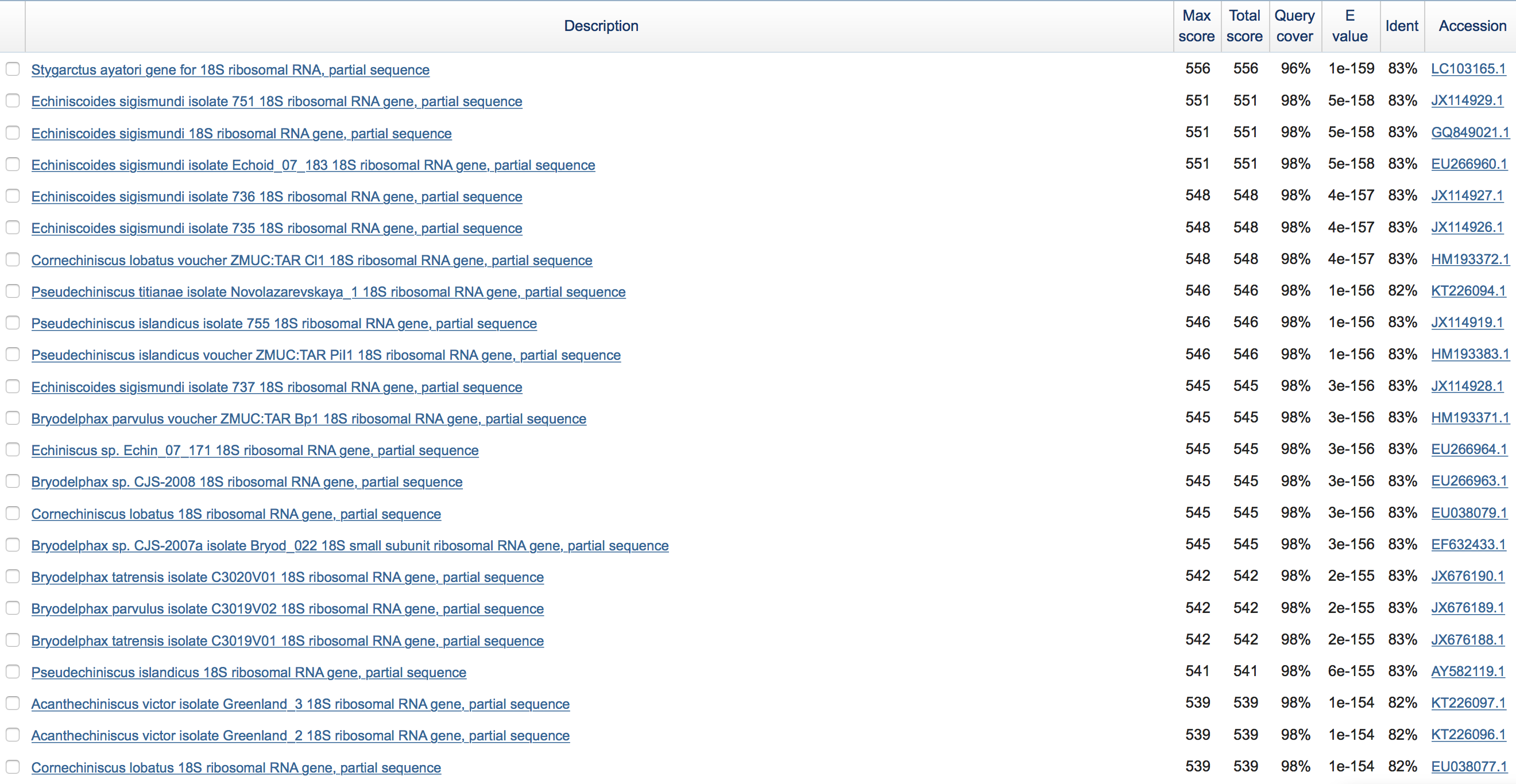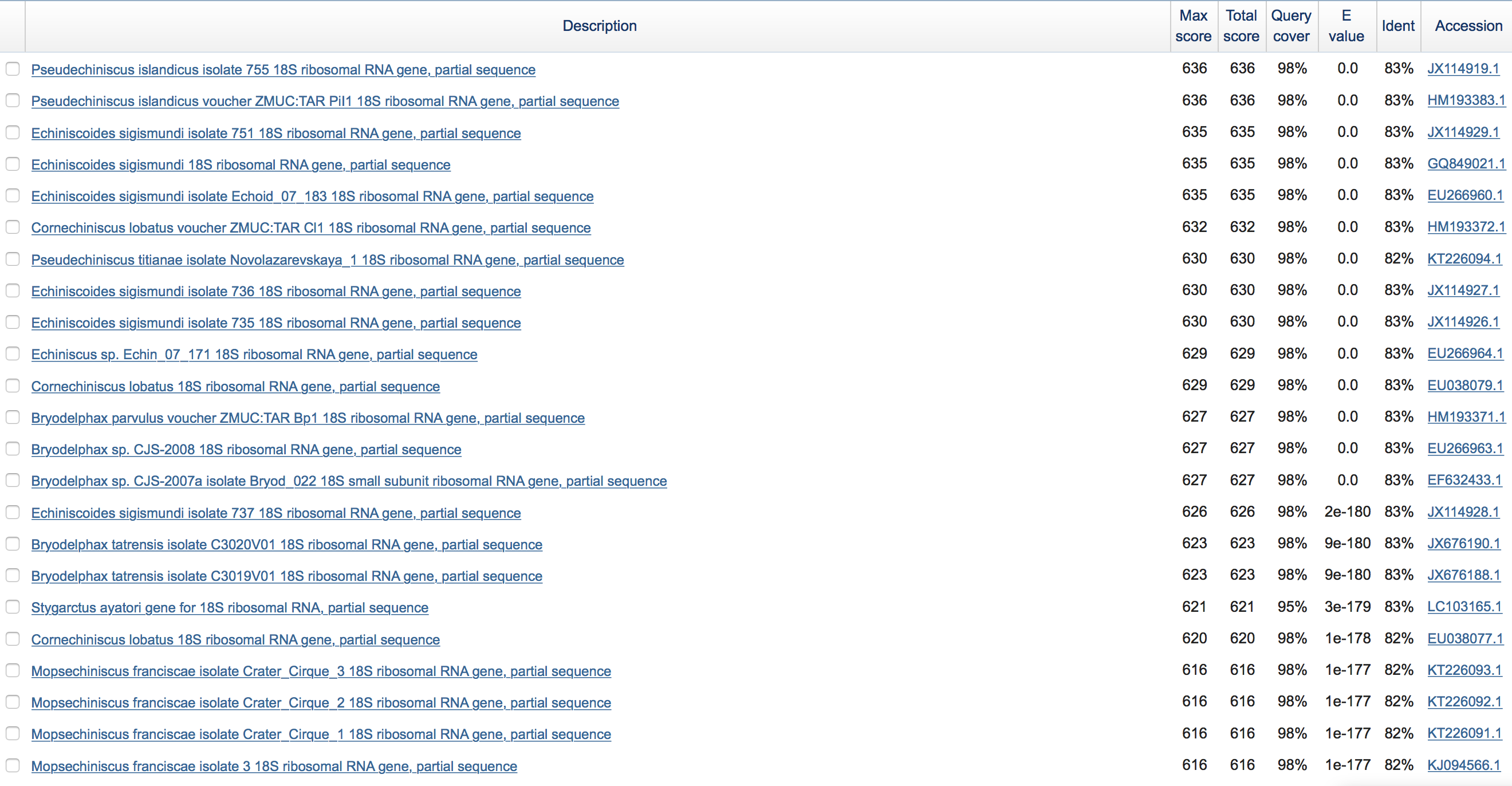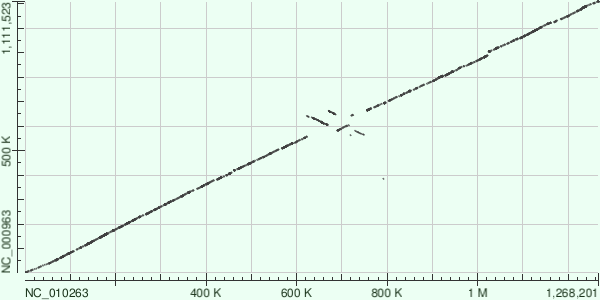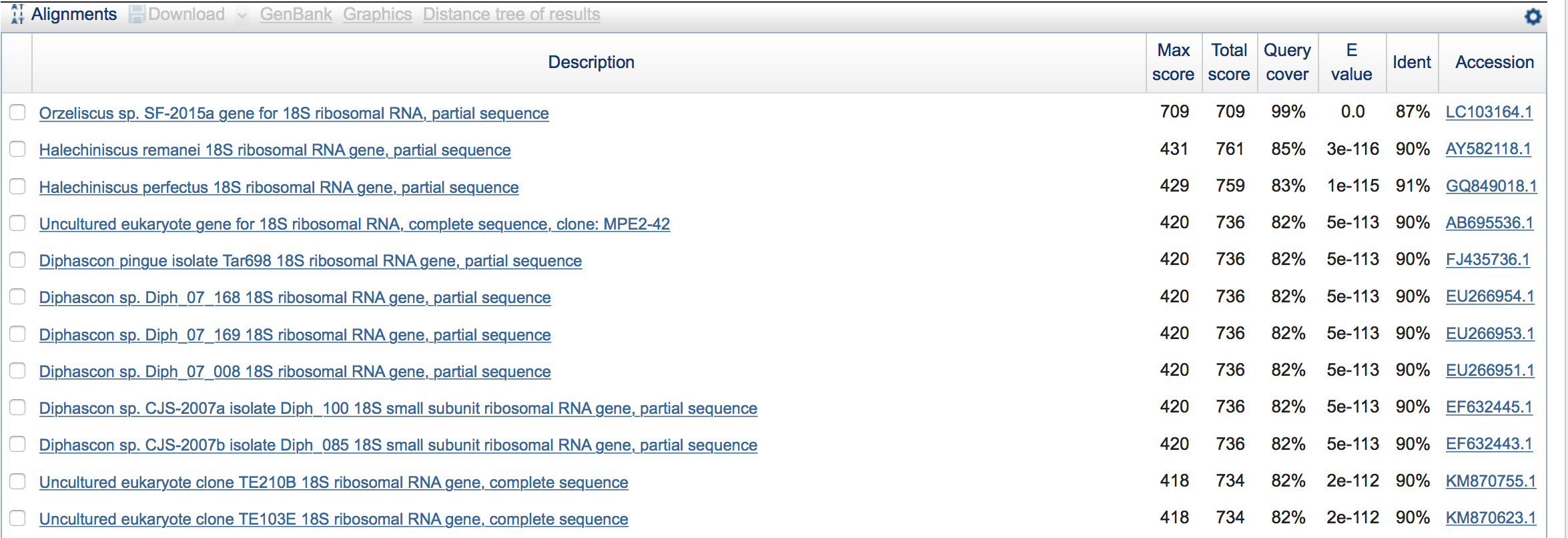
| параметр | значение для megablast | значение для blastn с максимально чувствительными параметрами | значение для blastn с параметрами по умолчанию |
| expect threshold | 10 | 10 | 10 |
| word size | 28 | 7 | 11 |
| match/mismatch scores | 1,-2 | 1,-1 | 2, -3 |
| gap costs | linear | existence:2, extension: 1 | existence:5, extension: 2 |
| organism | Heterotardigrada (taxid:89368) Exclude Halechiniscidae (taxid:286673) |
Heterotardigrada (taxid:89368) Exclude Halechiniscidae (taxid:286673) |
Heterotardigrada (taxid:89368) Exclude Halechiniscidae (taxid:286673) |
| mucleotide collection | (nr/nt) | (nr/nt) | (nr/nt) |