Практикум 14. Сборка генома de novo.
1. Подготовка чтений программой trimmomatic
Удаляем остатки адаптеров:
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240361.fastq SRR_no_adapters.fastq ILLUMINACLIP:adapters.fasta:2:7:7
Результат:
Input Reads: 7272621 Surviving: 7238064 (99,52%) Dropped: 34557 (0,48%)
Удаляем плохие буквы с концов чтений:
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR_no_adapters.fastq SRR_trimm.fastq LEADING:20 TRAILING:20 MINLEN:30
Результат:
Input Reads: 7238064 Surviving: 6876983 (95,01%) Dropped: 361081 (4,99%)
Исходный файл SRR_no_adapters.fastq весил 764,7 МБ, получившийся весил 722,6 МБ (меньше на 42,1 МБ)
2. Подготовка k-меров
Следующая команда создает директорию kmeri, в которую кладет log и файлы c 29-мерами из исходного файла с нашими короткими ридами (поэтому -short), который был в формате fastq (поэтому -fastq)
velveth kmeri 29 -fastq SRR_trimm.fastq -short
3. Cборка на основе k-меров
velvetg kmeri
Результат:
Final graph has 1173 nodes and n50 of 49972, max 155850, total 690667, using 0/6876983 reads
N50: 49972
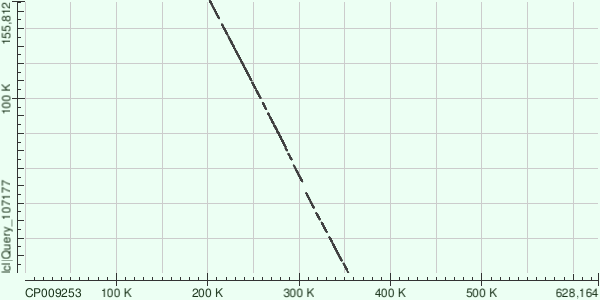
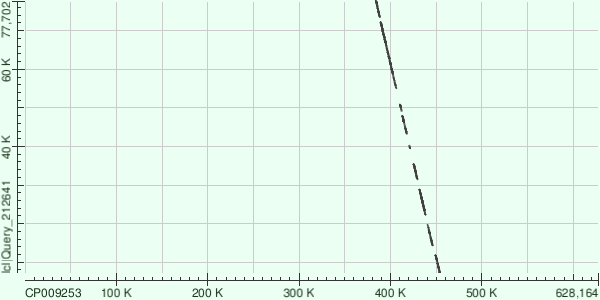
самые длинные контиги [длина/покрытие]: ID 3 [155850/33.07], ID 11 [85024/34.7], ID 1 [72780/35.5]. Загадкой остается, в чем прокрамма velvetg измеряет покрытие (столбцы short1_cov и short1_Ocov). Проблема в том, что больше 20 ридов имеет "покрытие" больше 30, что говорит о том, что это явно не проценты. Причем самый длинный рид не имеет самого большого покрытия. В документации найти информацию не удалось.
4. Анализ
| ID |
length |
score |
e-value |
identity |
gaps |
координаты выравнивания по хромосоме |
query cover |
dot matrix |
| 3 |
155850 |
6154 (max score) |
0.0 |
79% |
355 (3%) - для лучшего из выравниваний |
81937-91416 - для лучшего из выравниваний |
18% |
 |
| 11 |
85024 |
3605 (max score) |
0.0 |
74% |
373 (3%) - для лучшего из выравниваний |
62724-72162 - для лучшего из выравниваний |
7% |
 |
| 1 |
72780 |
4047 (max score) |
0.0 |
77% |
206 (2%) - для лучшего из выравниваний |
57822-65135 - для лучшего из выравниваний |
7% |
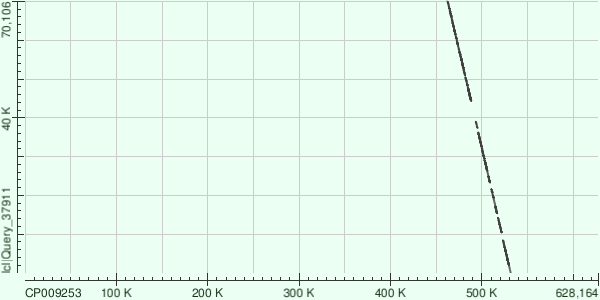 |
Каждый из трех контигов дал несколько выравниваний с разными участками референсной последовательности, что иллюстрируют представленные графики: соответствие контигов и хромосомы перемежаются разрывами.
Замечательно, что контиги ложатся на неперекрывающиеся участки хромосомы (мы этого и ждали).





