Практикум 3. Построение дерева, содержащего паралоги
Последовательность CLPX_ECOLI, для которой искали гомологов:
>sp|P0A6H1|CLPX_ECOLI ATP-dependent Clp protease ATP-binding subunit ClpX OS=Escherichia coli (strain K12) OX=83333 GN=clpX PE=1 SV=2
MTDKRKDGSGKLLYCSFCGKSQHEVRKLIAGPSVYICDECVDLCNDIIREEIKEVAPHRE
RSALPTPHEIRNHLDDYVIGQEQAKKVLAVAVYNHYKRLRNGDTSNGVELGKSNILLIGP
TGSGKTLLAETLARLLDVPFTMADATTLTEAGYVGEDVENIIQKLLQKCDYDVQKAQRGI
VYIDEIDKISRKSDNPSITRDVSGEGVQQALLKLIEGTVAAVPPQGGRKHPQQEFLQVDT
SKILFICGGAFAGLDKVISHRVETGSGIGFGATVKAKSDKASEGELLAQVEPEDLIKFGL
IPEFIGRLPVVATLNELSEEALIQILKEPKNALTKQYQALFNLEGVDLEFRDEALDAIAK
KAMARKTGARGLRSIVEAALLDTMYDLPSMEDVEKVVIDESVIDGQSKPLLIYGKPEAQQ
ASGE
Бактерии, в протеомах которых искали гомологов:
| Название |
Мнемоника |
| Burkholderia cenocepacia |
BURCA |
| Neisseria meningitidis |
NEIMA |
| Roseobacter denitrificans |
ROSDO |
| Serratia proteamaculans |
SERP5 |
| Shewanella denitrificans |
SHEDO |
| Thiobacillus denitrificans |
THIDA |
| Yersinia pestis |
YERPE |
Команды, которые были использованы для запуска локального бласта:
makeblastdb -in proteomes.fasta -dbtype prot
blastp -query CLPX_ECOLI.txt -db proteomes.fasta -outfmt "6 sseqid evalue sseq" -evalue 0.0001 > blast_result.fasta
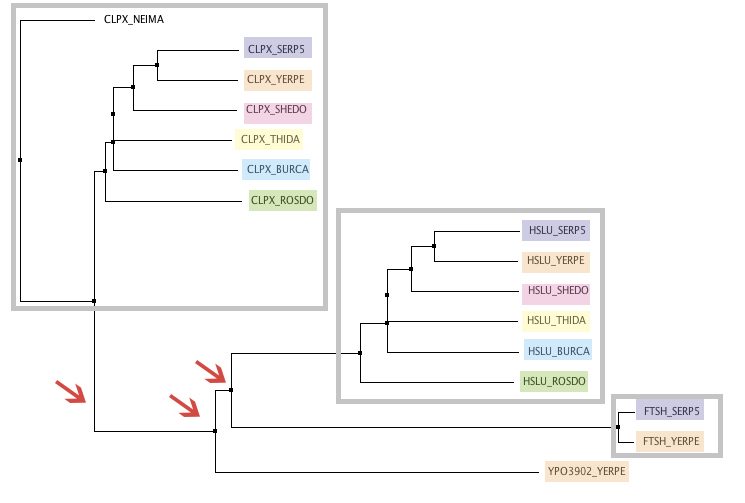
Из файла blast_result.fasta доставались id, по ним в файле со всеми протеомами искались последовательности (ведь дерево строить нужно по глобальному выравниванию, не по локальному, которое предоставляет бласт). После по выравниванию алгоритмом ClustalWS в программе JalView методом neighbour joining с использованием матрицы Blosum62 было построено дерево. На рисунке ниже разными цветами показаны паралоги, в серые рамки заключены ортологи, красными стрелочками отмечены события дупликации генов в некоторых из организмов.
Примечательно, что внутри групп ортологов CLPX и HSLU родственные отношения между белками разных видов совпадают. Поскольку эти белку очень схожи по функции (АТФ-связывающие домены протеаз), это ни о чем не говорит. Если бы они по функции отличались, можно было бы пытаться делать выводы об эволюционных отношениях видов.
Рисунок 1. Филогенетическое дерево белков-находок blast.