Трансмембранные белки
База данных OPM
Таблица 1. Описание мембранных белков| Белок | 5DSG | 1I78 |
| Название | Muscarinic acetylcholine receptor M4 | Outer membrane protease OmpT |
| Толщина гиброфобной части мембраны | 34.6 ± 2.1 A | 26.5 ± 1.6 A |
| Трансмембранные спирали/бета-тяжи (а.о.) | 1( 28- 55), 2( 70- 95), 3( 100- 129), 4( 148- 174), 5( 195- 220), 6(401- 425), 7( 432- 456) | 1(11-20), 2(50-61), 3(64-72), 4(111-121), 5(126-136), 6(178-187), 7(192-201), 8(230-240), 9(245-254), 10(287-296) |
| Среднее количество остатков в одной спирали/тяже | 22 | 9 |
| Локализация | Эукариотическая плазматическая мембрана | Внешняя мембрана грам-отрицательных бактерий |
| Изображение белка | 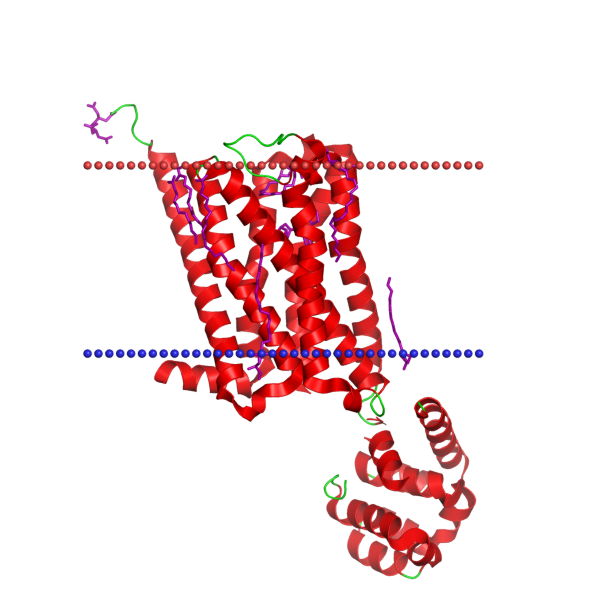 | 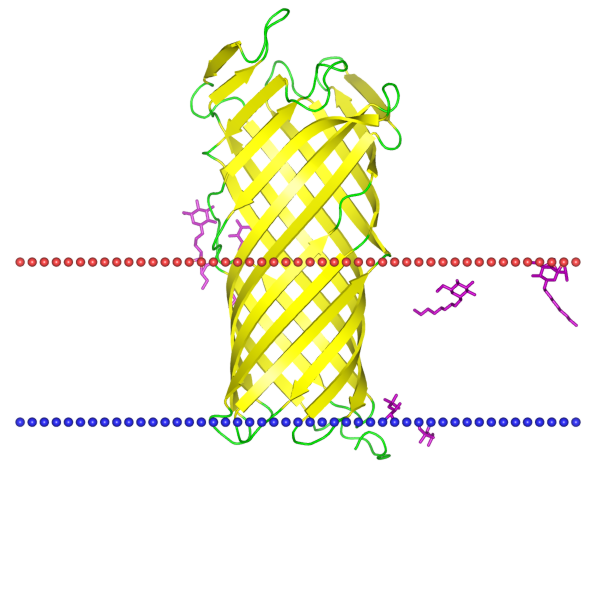 |
Анализ предсказания трансмембранных спиралей
| TMHMM | Phobius |
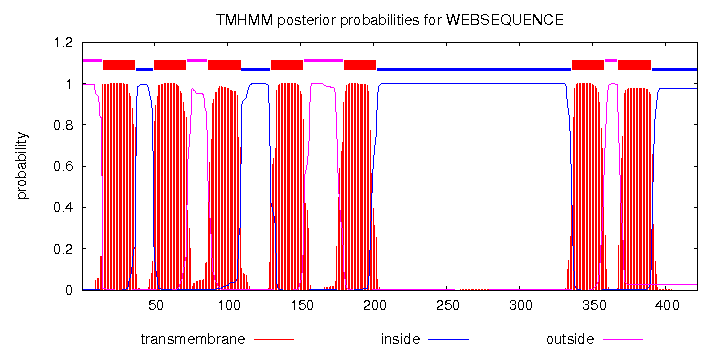 | 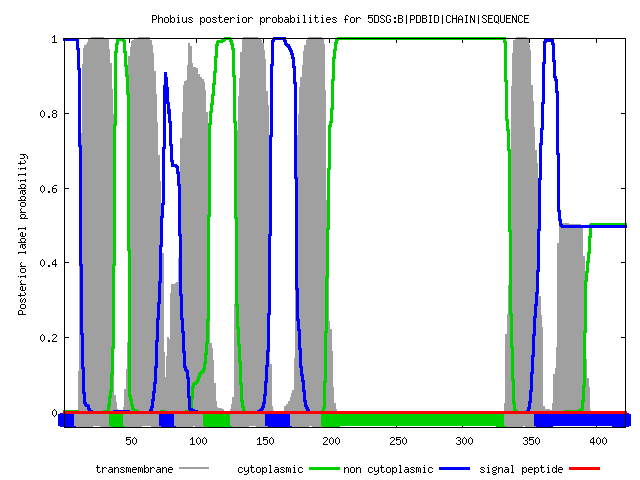 |
| По оси абсцисс отложены координаты аминокислотных остатков белковой последовательности, по оси ординат - вероятность того, что в данном месте есть трансмембранная спираль. Линии разных цветов соответсвуют: красные - трансмембранные участки, синии - выходящие в цитоплазму, розовые - выходящие наружу от цитоплазмы. | По оси абсцисс отложены координаты аминокислотных остатков белковой последовательности, по оси ординат - вероятность того, что в данном месте есть трансмембранная спираль. Линии разных цветов соответсвуют: серые - трансмембранные участки, зеленые - выходящие в цитоплазму, синие - не присутсвующие в цитоплазме, красные - сигнальная последовательность |
Текстовая выдача TMHMM
outside 1 14
TMhelix 15 37
inside 38 49
TMhelix 50 72
outside 73 86
TMhelix 87 109
inside 110 129
TMhelix 130 152
outside 153 179
TMhelix 180 202
inside 203 335
TMhelix 336 358
outside 359 367
TMhelix 368 390
inside 391 422
| Текстовая выдача Phobius TOPO_DOM 1 12 NON CYTOPLASMIC. TRANSMEM 13 38 TOPO_DOM 39 49 CYTOPLASMIC. TRANSMEM 50 76 TOPO_DOM 77 87 NON CYTOPLASMIC. TRANSMEM 88 109 TOPO_DOM 110 129 CYTOPLASMIC. TRANSMEM 130 155 TOPO_DOM 156 174 NON CYTOPLASMIC. TRANSMEM 175 197 TOPO_DOM 198 335 CYTOPLASMIC. TRANSMEM 336 357 TOPO_DOM 358 422 NON CYTOPLASMIC |
| Из приведенных выше графиков видно, что сервером TMHMM были найдены все 7 спиралей, присутствующие в OPM, однако координаты не совпадают с координатами из OPM, сдвиг наблюдается где-то на 20 аминокислот. Сервером Phobius были найдены лишь 6 спиралецй (с 1 по 6). Седьмой пик присутствует на графике, однако его вероятность около 0.5. Координаты спиралей не совпадают с координатами с OPM, однако близки к координатам, предсказанным TMHMM за исключением последней спирали. | |
База данных TCBD
Белок 5DSG в базе не представлен.TCID код для 1I78: 9.B.50.1.1;
9 - белки с неполностью охарактеризованными транспортными системами (Incompletely Characterized Transport Systems);
B - предположительно транспортные белки (Putative transport proteins);
50 - Эндопротеаза наружной мембраны бета-тяжа (The Outer Membrane Beta-barrel Endoprotease, Omptin (Omptin) Family);
информация о субстрате отсуствтует.
©Шкарина Анастасия Николаевна 2016


