Исследование структуры тРНК
I. Краткое описание структуры в файле 1il2.pdb
В файле приведены координаты атомов комплекса аспартил-тРНК-синтетазы (из Escherichia coli) с аспартил-тРНК (из Saccharomyces cerevisiae). Обе молекулы в количестве 1 каждая. Для исследования была выбрана цепь C, представляющая аспартил-тРНК со следующей последовательностью:
[901] 5' - U C C G U G A U A G U U PSU A A H2U G G H2U C A G A A U G G G C G C PSU U G U C 1MG C G U G C C A G A U 5MC G G G G 5MU PSU C A A U U C C C C G U C G C G G A G C C A - 3' [976].
В последовательности на 3'-конце есть триплет CCA, к которому присоединяется аминокислота, координаты его атомов приведены.
В файле PDB представлена неверная нумерация нуклеотидов: 46-й по счету нуклеотид имеет номер 946, а 47-й — 948, 48-й — 949 и т.д. Поэтому, несморя на то что всего нуклеотидов 75, номер последнего нуклеотида — 976. 46-й и 47-й нуклеотиды находятся в промежутке между антикодоновым и T-стеблем.
II. Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями (ссылка на выходной файл). В соответствии с полученными данными:
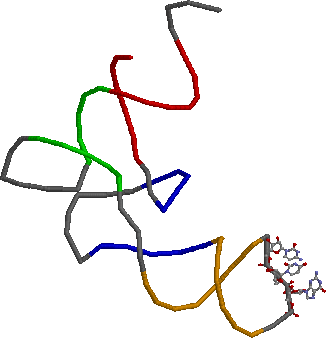
- акцепторный стебель состоит из участка 901-907 и комплементарного ему участка 966-972;
- Т-стебель состоит из участка 949-953 и комплементарного ему участка 961-965;
- D-стебель состоит из участка 910-914 и комплементарного ему участка 921-925;
- антикодоновый стебель состоит из участка 926-932 и комплементарного ему участка 938-944.
Рис. 1. Вторичная структура аспартил-тРНК из Saccharomyces cerevisiae
|
Скрипт для получения изображения: restrict nucleic |
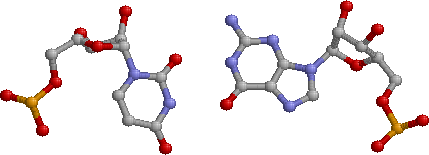
Структуру стеблевых дуплексов поддерживают 18 канонических и 12 неканонических пар оснований. Пример неканонической пары — пара U905-G968:
Вариабельная петля имеется (945-948 нуклеотиды). Остатка тимидина, строго говоря, нет, но есть остаток 5-метилуридина (с азотистым основанием тимином) (нуклеотид 954). Дигидроуридины в D-петле присутствуют (нуклеотиды 916, 919).
III. Исследование третичной структуры
1. Стэкинг-взаимодействия присутствуют между соседними краями акцепторного и T-стеблей:
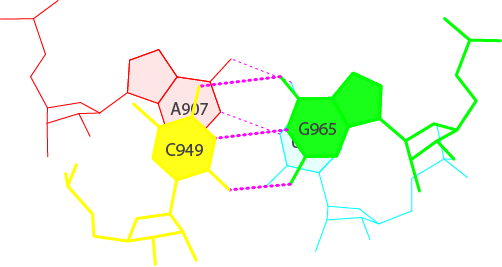
а также антикодонового и D-стеблей:
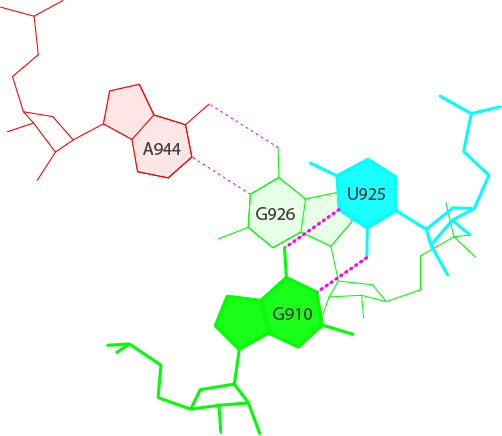
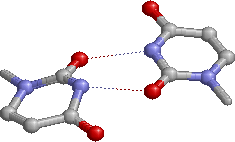
2. Имеются дополнительные водородные связи между основаниями D- и T-петель: нуклеотиды H2U916-U959, G917-PSU955 и G918-C956. 2 неканонические пары и одна каноническая. Пример неканонической пары — пара H2U916-U959:
IV. Предсказание вторичной структуры тРНК
| Участок структуры | Позиции в структуре по результатам find_pair | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-901-907-3' 5'-966-972-3' Всего 7 пар |
Предсказаны 0 пар | Предсказаны 7 пар из 7 реальных |
| D-стебель | 5'-910-914-3' 5'-921-925-3' Всего 5 пар |
Предсказаны 0 пар | Предсказаны 4 пары из 5 реальных |
| T-стебель | 5'-949-953-3' 5'-961-965-3' Всего 5 пар |
Предсказаны 5 пар из 5 реальных | Предсказаны 3 пары из 5 реальных |
| Антикодоновый стебель | 5'-926-932-3' 5'-938-944-3' Всего 7 пар |
Предсказаны 0 пар | Предсказаны 5 пар из 7 реальных |
| Общее число канонических пар нуклеотидов | В составе стеблей 24 пары | 5 пар | 19 пар |
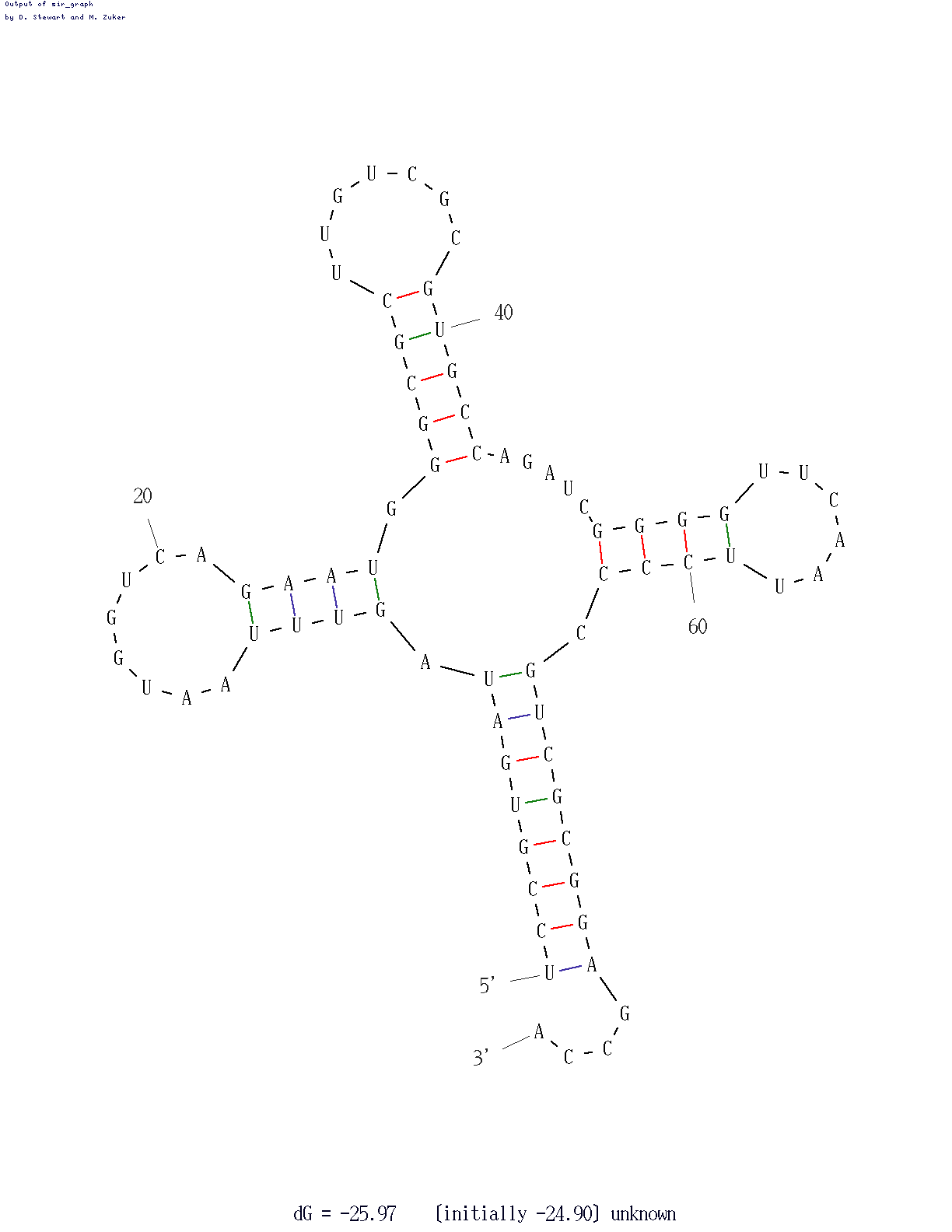
Рисунок, полученный с помощью mfold (3-я по счету структура):
Параметры использованных программ:
- find_pair: параметр "-t" (читать также записи в HETATM);
- einverted: все параметры по умолчанию, кроме "-threshold 0" (minimum score threshold) — при значении 50 (по умолчанию) программа ничего не находит;
- mfold: все параметры web-варианта по умолчанию, кроме P=15 (percent) — при значениях 5 (по умолчанию) и 10 не находит структур, похожих на реальную, а при значениях 15, 20, ... среди найденных структур есть одна близкая к реальной.
Вывод: mfold (т.е. используемый им алгоритм Зукера) "не сумел" предсказать реальную вторичную структуру исследуемой тРНК. Это объясняется, прежде всего, тем, что алгоритм не учитывают другие виды взаимодействий, отличные от водородных связей между основаниями, влияющие как на вторичную, так и на третичную структуру молекулы.