4. Реконструкция деревьев по нуклеотидным последовательностям. Анализ деревьев, содержащих паралоги.
1. Построение дерева по нуклеотидным последовательностям
Строю филогенетическое дерево своих бактерий, используя последовательности 16S рРНК.
| Организм | AC записи EMBL, описывающей полный геном | Координаты выбранной рРНК в записи | Направление |
| Lactobacillus delbrueckii | CR954253 | 45160--46720 | + |
| Lactococcus lactis | AM406671 | 511423--512971 | + |
| Listeria monocytogenes | AL591981 | 100741--99187 | − |
| Staphylococcus aureus | AP009324 | 531922--533476 | + |
| Staphylococcus epidermidis | AE015929 | 1599559--1598006 | − |
| Streptococcus pyogenes | AE004092 | 17067--18616 | + |
| Streptococcus pneumoniae | AE005672 | 15344--16903 | + |
Построив выравнивание программой muscle, реконструирую дерево с помощью fdnaml:
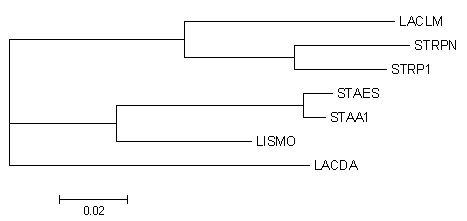
Полученное дерево не противоречит правильному, однако в нем отсутствует ветвь {STAA1,STAES,LISMO} против {STRP1,STRPN,LACLM,LACDA}, что делает это дерево небинарным. Поэтому в данном случае такая реконструкция уступает в качестве построению деревьев с использованием белковых последовательностей.
2. Построение и анализ дерева, содержащего паралоги
Провожу поиск достоверных гомологов белка CLPX_BACSU в своих бактериях. Для этого использую программу blastp; fasta-последовательности нужных находок загружаю через uniprot.org > Retrieve, используя их ID.
Реконструирую дерево с помощью программы fprotpars, предварительно выровняв их программой muscle. Полученное дерево:
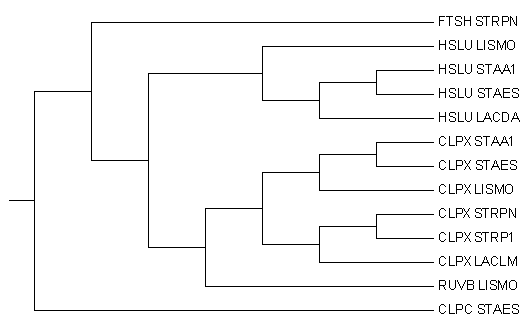
Судя по полученному дереву, делаю вывод, что ортологами являются пары CLPX_STAA1 --- CLPX_LISMO и HSLU_STAES --- HSLU_LISMO, а паралогами пары HSLU_STAES --- CLPC_STAES, FTSH_STRPN --- CLPX_STRPN, CLPX_LISMO --- RUVB_LISMO.