Миниобзор генома археи Methanosarcina vacuolata
Аннотация
Метаногенные археи – удивительные микроорганизмы, которые освоили чрезвычайно широкий спектр экологических ниш. Вследствии этого они имеют уникальный, ни на что не похожий геном и протеом, изучению которых и посвящен данный миниобзор.
Введение
Methanosarcina – это род анаэробных метаногенных архей, которые широко распространены в самых различных местах обитания: от мусорных свалок и сточных вод до глубоких подземных вод и кишечника млекопитающих [1]. Данный организм относится к семейству Methanosarcinaceae , порядку Methanosarcinales , классу Methanomicrobia , филлуму Euryarchaeota царства Euryarchaeota . В 2002 году, исследуя M. barkeri , ученые обнаружили необычную аминокислоту пирролизин, которая, как оказалось, была закодирована одним из стоп кодонов, а именно UAG [2]. Считается, что метаносарцины могли оказать решающее влияние на развитие жизни на нашей планете. Так, например, метан и углекислый газ, выделяемый ими в больших количествах, мог стать главной причиной великого пермско-триасового вымирания(250 млн. лет назад), в результате которого биоразнообразие упало примерно на 90% [3]. В последнее время эти археи интересуют исследователей как потенциальный источник биотоплива [4].
Данная работа посвящена анализу протеома и генома археи с целью выявления уникальных особенностей их организации, позволившим им занять столь необычную нишу.
Материалы и методы
В качестве исходных данных для анализа генома и протеома использовались следующие файлы, взятые из базы данных NCBI[5]: feature_table, cds, genomic.fna, *translated_cds_faa.
Анализ протеома выполнен при помощи методов электронных таблиц, а также сценария на Python. [S1, S2]
Анализ распределения генов по категориям выполнен с помощью методов электронных таблиц и bash-сценария. [S3, S4]
Анализ старт-кодонов выполнен с помощью bash-сценария, а стоп-кодонов - с помощью скрипта на Python[S5, S6]
Анализ межгенных промежутков был сделан методами электронных таблиц, а предсказание оперонов – Python-сценарием.[S7, S8, S9]. Будем считать, что два соседних гена находятся в одном опероне, если между ними расстояние меньше или равно 50 нуклеотидов.
Результаты
1. Анализ протеома
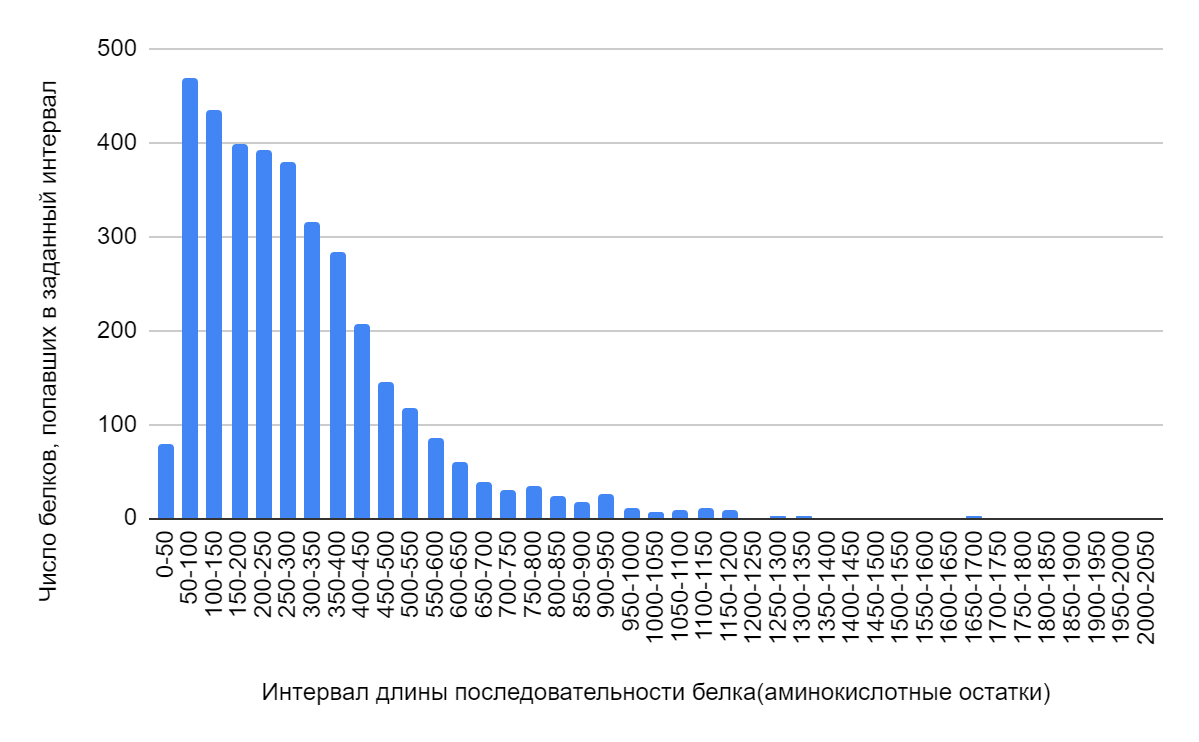
Зависимость распределения числа белков от их длины представлена в виде столбчатой диаграммы( Рис. 1 ). Общее количество белков, закодированных в геноме археи, – 3601, при этом их длины расположены в промежутке от 24 до 2002 аминокислотных остатков. Согласно полученным данным, медианное значение длины белка лежит в пределах от 50 до 100 аминокислот, а среднее арифметическое равно 298. На диаграмме виден резкий скачок при переходе от длины 0-50 к 50-100. По-видимому, для выполнения большинства функций длина белка должна быть более 50 аминокислотных остатков. Я думаю, что это связано с тем, что белку для выполнения своей функции нужно иметь определенную пространственную структуру, которая определяется физико-химическими свойствами аминокислот. В случае маленькой длины белка разнообразие физико-химических пептидных последовательностей будет маленьким, что не даст эволюции достаточно материала для отбора нужных функций
Рассмотрим какие функции выполняют в организме некоторые из самых коротких и длинных белков(см. Табл. 1 ). Заметим, что какой-либо закономерности в функциональном распределении белков по размерам нет

Рассмотрим некоторые закономерности содержания аминокислот в протеоме(см. Табл. 2 ). Среди гидрофобных аминокислот больше всего лейцина, изолейцина и глицина, среди отрицательно заряженных - глутамата, среди положительно заряженных - лизина, а среди полярных незаряженных - серина. По-видимому, первые во Вселенной белки состояли не из 20 разных видов аминокислот, а из гораздо меньшего количества. Таким образом, процентное содержание “древних” аминокислот в современных белка намного больше, чем приобретенных позднее в процессе эволюции. Так например, содержание лейцина 9,45%, а триптофана – всего 1%. Это означает, что древние белки содержали в качестве главной гидрофобной аминокислоты лейцин, а не триптофан. Кроме того, в аминокислотном составе протеома присутствую нестандартные аминокислоты(*). Это может быть как раз пирролизин или селеноцистеин – дополнительные 21 и 22 аминокислоты.

2. Анализ распределения генов по категориям
Согласно данным из Табл. 3 больше всего в основной хромосоме археи генов кодирующих белки, в то время как гены, кодирующие различные виды РНК, составляют лишь небольшую часть генома. Так, например, присутствуют некодирующие РНК. Одна из них – RNase P RNA component – весьма интересна, так как является рибозимом, катализирующим некоторые стадии синтеза тРНК. Этот реликтовый рибозим – остаток некогда богатого мира каталитических РНК, которые ушли на второй план из-за белков, которые могут катализировать реакции значительно более сложные, чем РНК. Хотя, например, рРНК тоже сохраняет свою каталитическую активность в рибосомах каждого из нас и по сей день. Рибосомальные РНК данной археи имеют константы седиментации 5-S, 16-S и 23-S. Транспортные РНК подробно анализируются ниже. Стоит отметить, что генов РНК на плазмиде нет вообще. В ней закодированы белки метаболизма гликопротеинов, которые образуют их сложную систему клеточных покровов. [6]

Проанализируем подробнее число тРНК, которые соответствуют определенной аминокислоте(см. Табл. 4 ). Заметим интересную особенность: в стандартном генетическом коде 6 кодонов соответствуют лейцину, в то время как существует 7 разных тРНК. Получается, либо нужно смириться с тем, что генетический код археи отличается от стандартного, либо одному кодону могу соответствовать две разных тРНК. Напротив, для серина в стандартном генетическом коде есть 6 кодонов, в то время как разных тРНК для него всего 4. Значит, опять либо генетический код нестандартный, либо какая-то тРНК серина может распознавать сразу два его кодона. Подобные противоречия можно найти и в тРНК для других аминокислот.

3. Анализ старт- и стоп-кодонов
Типичным старт кодоном у всех форм жизни является ATG, что и подтверждается данными из Табл. 5 . Помимо этого довольно часто в качестве старт кодона встречаются TTG и GTG 395 и 259 раз соответственно. Они согласно данным из статьи [7], тоже являются довольно типичными старт- кодонами для прокариот. А вот остальные старт-кодоны таковыми на самом деле не являются, потому что принадлежат к псевдогенам, которые возникают из обычных работающих генов путем мутаций и перестают работать. Поскольку у них нет нормального старт-кодона рискну предположить, что точно имела место быть делеция участка гена с 5’-конца. По данным Табл. 6 , число канонических стоп-кодонов TAA, TGA и TAG соответственно равно 2080, 1443 и 155. Остальные же стоп-кодоны таковыми не являются, потому что отвечают псевдогенам. Так как у этих псевдогенов отсутствует нормальный стоп-кодон, то можно предположить, что здесь, наоборот, имела место делеция со стороны 3’-конца.


4. Анализ межгенных промежутков и предсказание оперонов
Исходя из приведенных ниже данных(см. Рис. 2 ), видно, что у большинства генов межгенное расстояние лежит в интервале от 100 до 200 нуклеотидов. Это может свидетельствовать о том, что большинство генов археи организовано в опероны для совместной регуляции экспрессии. Заметим, что порядка 110 генов перекрываются. То есть некоторые белки могут иметь участки с общей аминокислотной последовательностью и следовательно схожей третичной структурой. Возможно, что это различные изоформы одного и того же белка с немного различающимися функциями. В Табл. 7 показаны продукты трех предсказанных оперонов(I, II, III). Оказывается, что все три оперона кодируют белки, участвующие в процессе метаногенеза[8]. По большому числу субъединиц этих белков можно понять, что они имеют очень сложную пространственную структуру и , самое главное, то что по отдельности каждая субъединица белка не может выполнять свою функцию. Таким образом, смысл объединения этих генов в оперон очевиден.


Сопроводительные материалы
S1. Таблица для подсчета длины белков(см. листы proteins и protein length)
S2. Программа для подсчета аминокислот(программа берет на вход файл *translated_cds_faa и возвращает таблицу с содержанием каждой аминокислоты).
S3. Таблица распределения генов по категориям(см. лист per-replicon).
S4. Bash-скрипт для подсчета тРНК каждого типа(скрипт берет на вход *feature_table а на выходе выдаёт число тРНК каждого типа).
S5. Bash-скрипт для подсчета старт-кодонов(скрипт получает на вход файл с CDS и выдаёт на выходе таблицу с числом старт-кодонов).
S6. Python-скрипт для подсчета стоп-кодонов(скрипт берет на вход файл с CDS и выдает на выходе таблицу с числом стоп-кодонов).
S7. Таблица подсчета межгенных интервалов(см. лист inter_cds_intervals-hist).
S8. Программа на Python для предсказания оперонов(программа берёт на вход *feature table и на выходе дает таблицу с названиями и координатами белков, которые теоретически могут быть объединены в оперон).
S9.Полный список предсказанных оперонов.
Литература
1. Galagan, J. E.; Nusbaum, C.; Roy, A.; Endrizzi, M. G.; MacDonald, P.; Fitzhugh, W.; Calvo, S.; Engels, R.; Smirnov, S.; Atnoor, D.; Brown, A.; Allen, N.; Naylor, J.; Stange-Thomann, N.; Dearellano, K.; Johnson, R.; Linton, L.; McEwan, P.; McKernan, K.; Talamas, J.; Tirrell, A.; Ye, W.; Zimmer, A.; Barber, R. D.; Cann, I.; Graham, D. E.; Grahame, D. A.; Guss, A. M.; Hedderich, R.; Ingram-Smith, C. (2002). The Genome of M. Acetivorans Reveals Extensive Metabolic and Physiological Diversity". Genome Research. 12 (4): 532–542. doi:10.1101/gr.223902. PMC 187521
2. "Science Notebook". The Washington Post. May 27, 2002. p. A09.
3. Rothman, D. H.; Fournier, G. P.; French, K. L.; Alm, E. J.; Boyle, E. A.; Cao, C.; Summons, R. E. (2014-03-31). Methanogenic burst in the end-Permian carbon cycle". Proceedings of the National Academy of Sciences. 111 (15): 5462–7. Bibcode:2014PNAS..111.5462R. doi:10.1073/pnas.1318106111. PMC 3992638.
4. "Researchers Engineer New Methane-production Pathway in Microoganism" (Press release). University of Arkansas. December 8, 2010.
5. https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/969/905/GCF_000969905.1_ASM96990v1/
6. https://biomolecula.ru/articles/arkhei-khamiat-i-pomogaiut
7. Blattner, F. R.; Plunkett g, G.; Bloch, C. A.; Perna, N. T.; Burland, V.; Riley, M.; Collado-Vides, J.; Glasner, J. D.; Rode, C. K.; Mayhew, G. F.; Gregor, J.; Davis, N. W.; Kirkpatrick, H. A.; Goeden, M. A.; Rose, D. J.; Mau, B.; Shao, Y. (1997). "The Complete Genome Sequence of Escherichia coli K-12". Science. 277 (5331): 1453–1462. doi:10.1126/science.277.5331.1453. PMID 9278503.
8. Baumer S, Ide T, Jacobi C, Johann A, Gottschalk G, Deppenmeier U (June 2000). "The F420H2 dehydrogenase from Methanosarcina mazei is a Redox-driven proton pump closely related to NADH dehydrogenases". The Journal of Biological Chemistry. 275 (24): 17968–73. doi:10.1074/jbc.M000650200. PMID 10751389.