Практическая работа 10
Сравнение dotplot карт локального сходства геномов, построенных с помощью megablast, blastn и tblastx
Для построения карт локального сходства я выбрал геномы вирусов иммунодефицита человека HIV1(OR400997) и HIV2(MK686247), найденные в базе данных ENA по запросу tax_tree(11632) AND description="complete genome". Далее было осуществлено построение карт локального сходства с помощью megablast с длиной слова 16 и blastn с длиной слова 7, остальные параметры программ были использованы по-умолчанию. На Рис. 1 и Рис. 2 приведены соответствующие карты локального сходства геномов. Кроме того, была построена карта локального сходства с помощью tblastx(см. Рис. 3) с параметрами по-умолчанию.
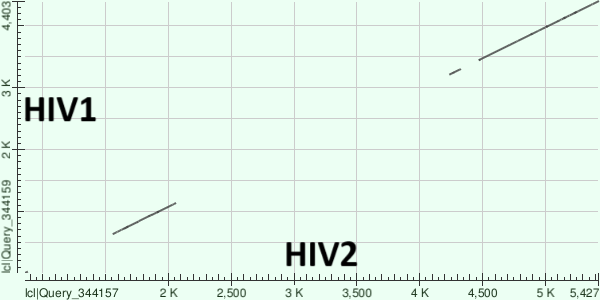
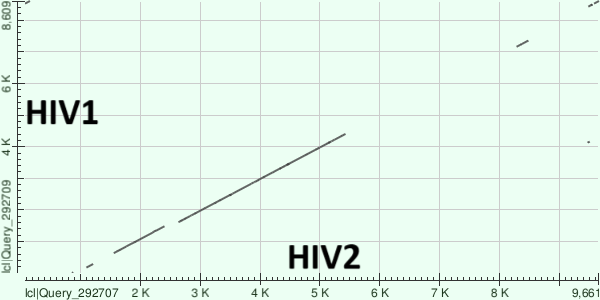
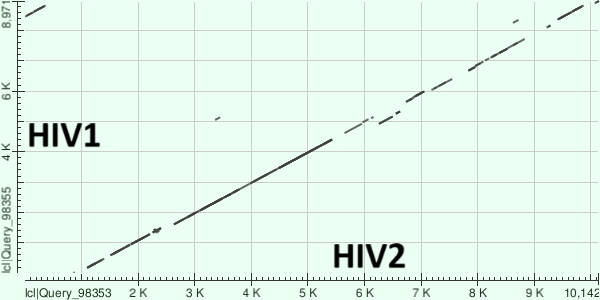
С помощью megablast выровнялась лишь небольшая часть генома вирусов(примерно половина генома каждого), причем участки гомологии последовательностей довольно маленькие. С помощью blastn в карту локального сходства геномы попали практически целиком, найденные участки гомологии значительно больше, чем те, что нашел megablast. Наиболее подробной получилась карта, построенная tblastx, потому что эта программа сравнивает последовательности на уровне трансляции, что имеет больший биологический смысл. Далее эволюционные изменения имеет смысл обсуждать только по карте tblastx. Виден участок наибольшего сходства с координатами 1000-5500(по HIV2) и участок, где накопилось большее число мутаций, с координатами 5500-10000(по HIV2). В качестве генома ВИЧ имеет линейную РНК, поэтому судя по карте локального сходства имела место транспозиция концевого участка генома(см. координаты 0-1000 по HIV2).