Сборка генома de novo
Скачивание чтений генома
Одноконцевые чтения генома Buchnera aphidicola str. Tuc7 с ID SRR4240361 были скачаны при помощи команды:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/001/SRR4240361/SRR4240361.fastq.gz
Подготовка чтений программой Trimmomatic
Файл со всеми возможными вариантами адаптеров, которые могли бы встретится в чтениях, был создан командой:
cat /mnt/scratch/NGS/adapters/*.fa > adapters.fa
Удаление адаптеров осуществлялось с помощью TrimmomaticSE следующей командой:
TrimmomaticSE -phred33 SRR4240361.fastq.gz SRR4240361_cleaned.fastq.gz ILLUMINACLIP:adapters.fa:2:7:7
В итоге, 0.47% чтений содержали адаптеры и были удалены:
Input Reads: 7272621 Surviving: 7238089 (99.53%) Dropped: 34532 (0.47%)
Далее были удалены чтения, на правом конце которых были нуклеотиды с качеством менее 20, а также чтения длины менее 32 нуклеотидов:
TrimmomaticSE -phred33 SRR4240361_cleaned.fastq.gz SRR4240361_final.fastq.gz MINLEN:32 TRAILING:20
В итоге, были удалены 1.28% чтений:
Input Reads: 7238089 Surviving: 7145146 (98.72%) Dropped: 92943 (1.28%)
Создание k-меров
Набор k-меров для k=31 создавался с помощью программы velveth:
velveth . 31 -short -fastq SRR4240361_final.fastq.gz
Сборка генома на основе k-меров
Cборка генома осуществлялась с помощью программы velvetg следующей командой:
velvetg .
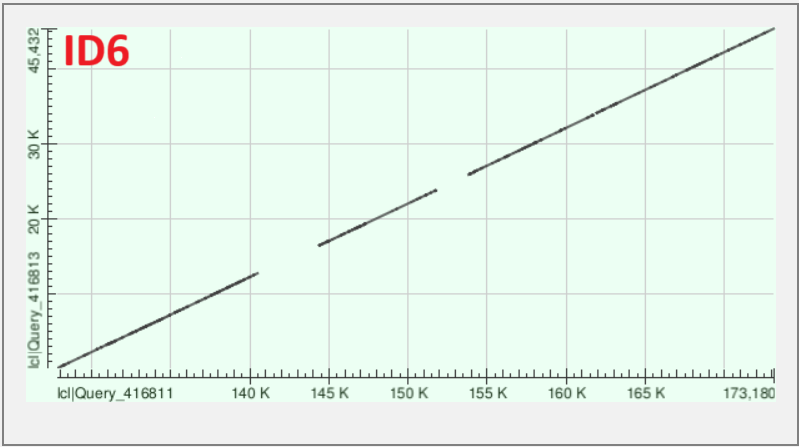
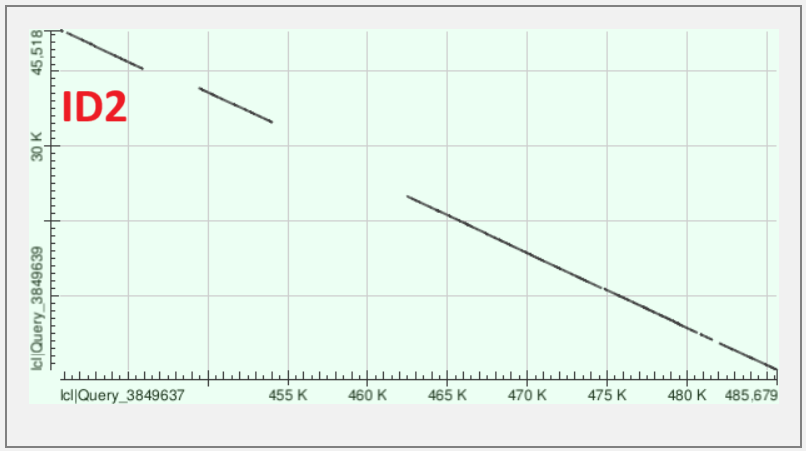
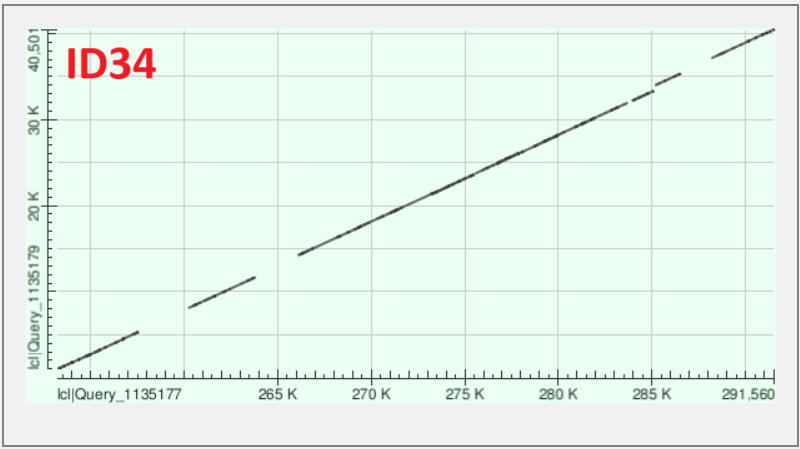
У получившейся сборки генома NP составляет 25683, наиболее длинные контиги с номерами (ID) 6, 2, 34 имеют длину 49238, 45555, 43866 и покрытия 26.667147, 26.456459, 23.521292 соответственно. Полная таблица контигов доступна по ссылке. Кроме того, нашелся контиг с аномально большим покрытием (ID=62) 214000 при среднем значении около 484. Также нашлось около десятка контигов с аномально низким покрытием равным 1.
Анализ контигов с помощью megablast
На Рис. 1-3 представлены карты локального сходства DotPlot для контигов ID6, ID2, ID34 с геномом Buchnera aphidicola (CP009253.1) из банка , полученные с помощью megablast.