Практическая работа 10
Поиск консервативного мотива в выравнивании
Для работы был выбран домен фосфолипазы А1 (Phospholipase A1) - PF02253. Фосфолипаза А1 - фермент, осуществляющий специфический гидролиз фосфолипидов. В сиде находится 57 последовательностей, в SwissProt 8 белков с этим доменом. Поиск мотива осуществлялся с помошью Jalview: были удалены последовательности на 90% идентичные (таких не оказалось), а затем производилась окраска по порогу консервативности. Проект Jalview доступен по ссылке. В результате, был найден мотив в позициях 178-183 с паттерном [SE]R[ST][WQL]N[RKY] в формате Jalview ([SE]-R-[ST]-[WQL]-N-[RKY] в формате Prosite).
Далее был произвден поиск данного мотива в выравнивании сид при помощи команды:
fuzzpro PF02253.fasta -pattern [SE]R[ST][WQL]N[RKY] result.txt
В выдаче мотив был найден по одному разу в каждой из 57 последовательностей. Таким образом, можно сказать, что паттерн мотива определен корректно.
Далее с помощью ScanProsite был проведен поиск белков в SwissProt по паттерну мотива. В выдаче была 141 находка. При этом были найдены все 8 фосфолипаз А1, содержащие изучаемый домен, которые есть в SwissProt. Остальные находки, по-видимому, случайные и к домену Phospholipase A1 никакого отношения не имеют.
Поиск мотива, специфичного для клады
При помощи метода NJ было построено филогенетическое дерево для белковых последовательностей, входящих в сид. Для поиска мотива, хапрактерного для клады была выбрана клада, состоящая из 8 организмов (ссылка на проект Jalview). Характерный для этой клады мотив располагается в позициях 1-8 и имеет паттерн [LI][SD].[RIL]WEL[DSA]. Этот мотив был обнаружен в 9 последовательностях сида из 57 (одна лишняя) при помощи команды (выдача):
fuzzpro PF02253.fasta -pattern [LI][SD]X[RIL]WEL[DSA] result2.txt
PSI-BLAST
Для поиска дальних гомологов с помощью psi-blast был выбран белок probable septum site-determining protein MinC из Prochlorococcus marinus с АС Q7VDL2. Данный белок учавствует в клеточном делении у цианобактерий. На Рис. 1 представлена информация об итерациях psi-blast. Стабилизация числа находок с E-value > 0.005 произошла на третьей итерации. Все найденные белки с E-value выше порога имеют названия probable septum site-determining protein MinC и, кроме того, разница между E-value худшей праильной находкой и лучшей неправильной находкой довольно большое. Все это говорит о том, что семейство найдено корректно.

Поиск мотивов de novo с помощью MEME
Были скачаны последовательности из SwissProt, содержащие исследуемый домен фосфолипазы А1 (PF02253). В них производился поиск 10 мотивов длины от 4 до 10 аминоксилота при помощи программы MEME:
meme PF02253_SP.fasta -o meme_results -minw 4 -maxw 10 -nmotifs 10
Выдача MEME доступна по ссылке. На Рис. 2 показана лого-диаграмма мотива, найденного вручную в первом задании. Почти все позиции на всех 10 найденных мотивах очень сильно консервативны, то есть почти в любой позиции встречается преимущественно только одна аминокислота.

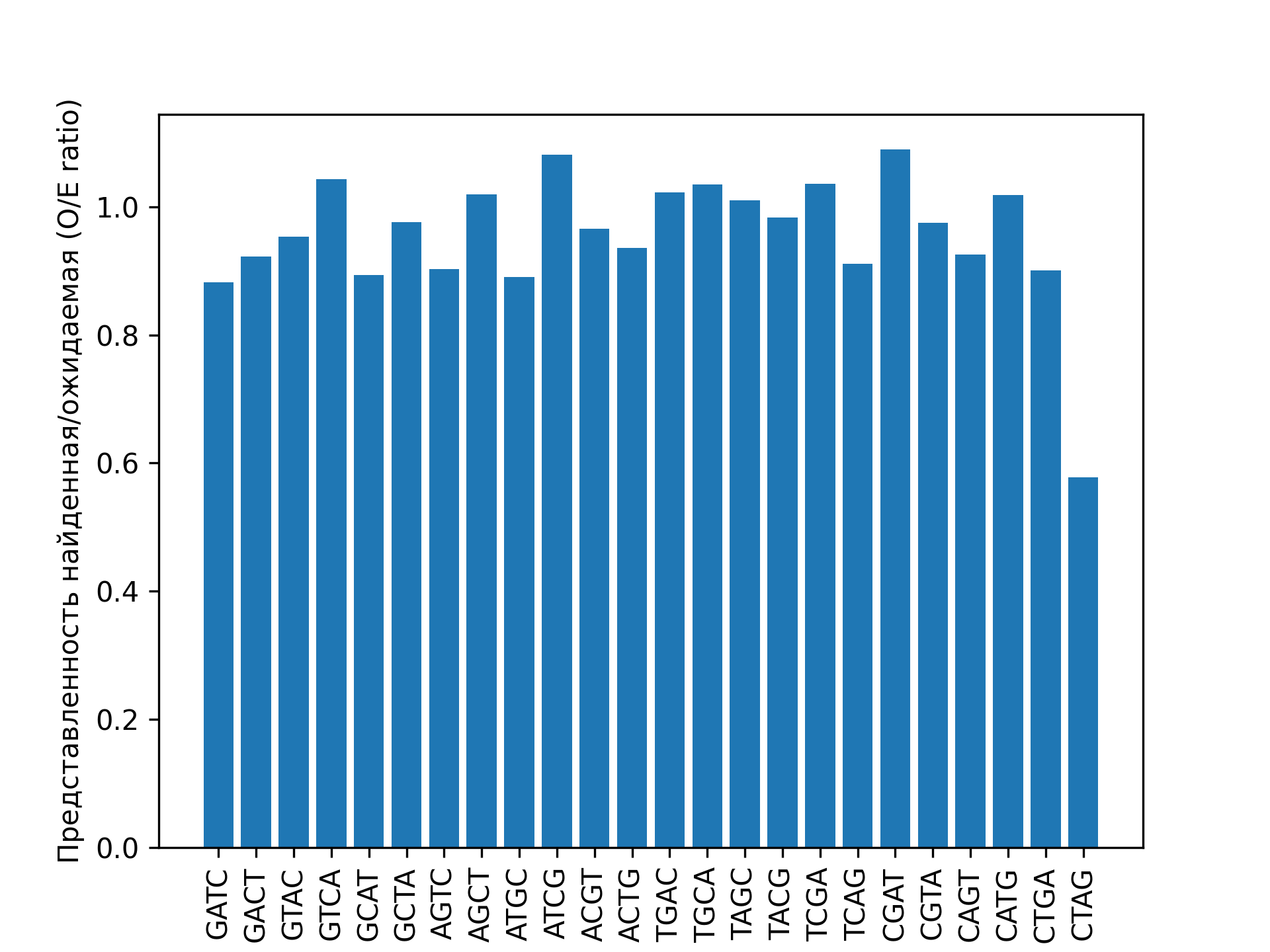
Оценка представленности сайта GATC
В геноме археи Methanosarcina vacuolata Z-761 (CP009520.1) была произведена оценка представлености всех 24 четырехнуклеотидных сайтов без повторений нуклеотидов при помощи команды:
cbcalc -s 24sites.txt -M -o res.tsv CP009520.1.fasta
Результаты оценки представлены на Рис. 3. Относительная представленность для сайта GATC равна 0.88. Если бы метилирование данного сайта играло бы важную роль в клеточных процессах этой археи, то стоило бы ожидать более высокую представленность (как минимум больше 1). Таким образом, по-видимому, GATC у археи Methanosarcina vacuolata не метилируется.