Практическая работа 2
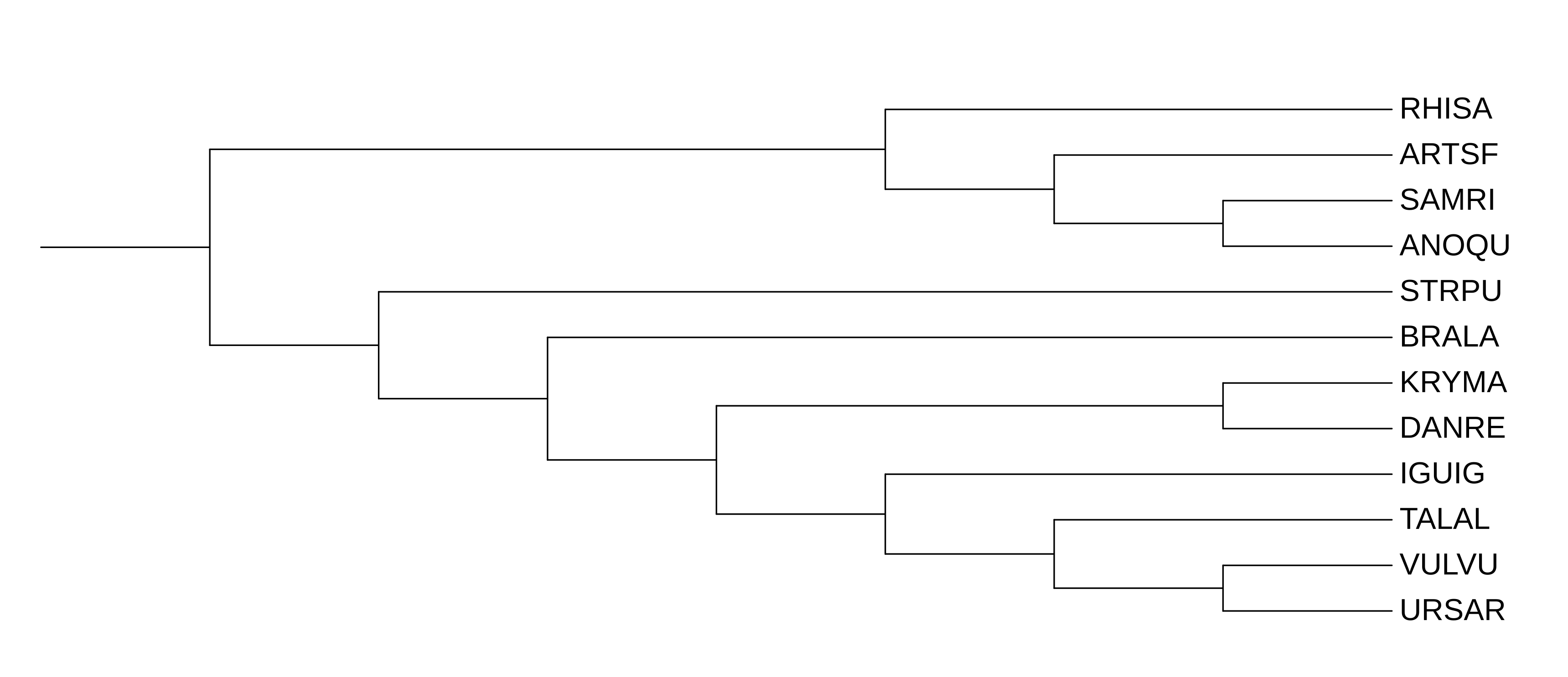
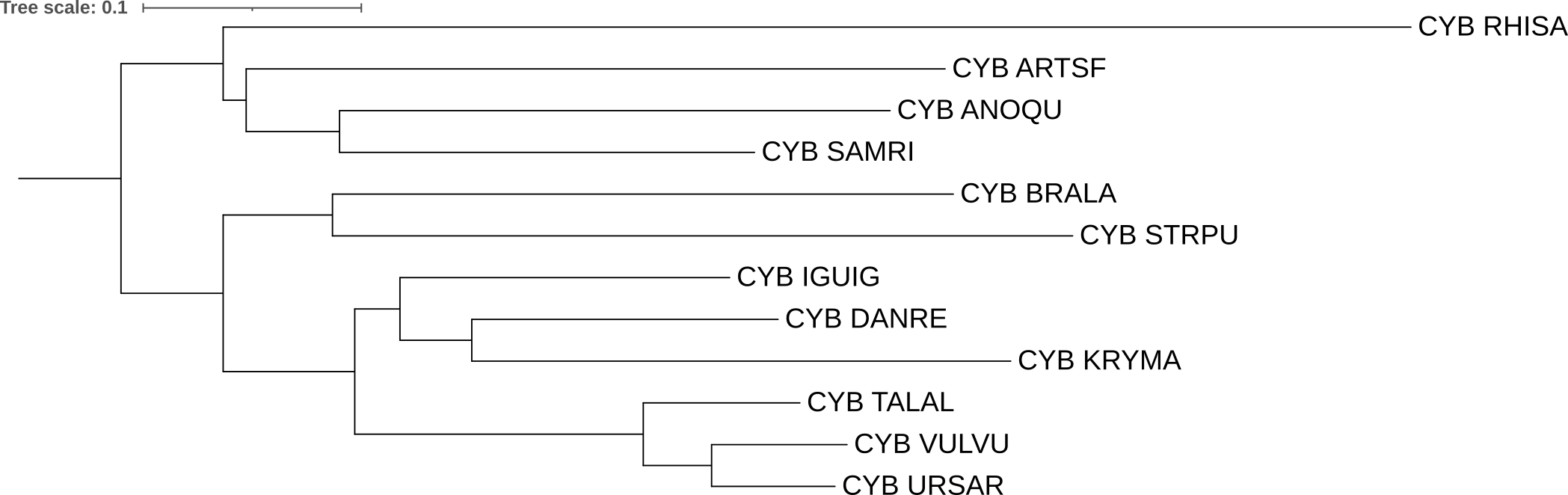
На Рис. 1 представлено филогенетическое дерево, построенное на основе данных NCBI Taxonomy, на Рис. 2 – дерево, построенное программой Fastme с моделью p-distance, на Рис. 3 – дерево, построенной программой Fastme с моделью MtREV и, наконец, на Рис. 4 – дерево, построенной программой IQtree с параметрами по умолчанию. Все деревья, полученные с помощью выще перечисленных программ, топологически эквивалентны и незначительно отличаются только эволюционными расстояниями, то есть длиной ветвей и листьев.
Теперь сравним полученное программами филогенетическое дерево с эталонным из NCBI Taxonomy. Единственная неточность реконструкции дерева заключается в местоположении ланцетника(BRALA) и морского ежа(STRPU). Программы объединили морского ежа и ланцетника в единую группу сестринскую к позвоночным. Но ,по данным NCBI Taxonomy, ланцетник должен быть базальной ветвью к позвоночным, а морского ежа следует расположить как базальную ветвь к группе (ланцетник + позвоночные). Таким образом, все три программы выдали топологически одинаковое дерево, которое отличается от эталонного лишь положением двух организмов: морского ежа и ланцетника.