Практическая работа 3
Реконструкция дерева по нуклеотидным последовательностям
На Рис. 1 - 2 приведено сравнение филогенетических деревьев, полученных на основе систематики NCBI Taxonomy и построенных по 12S-рРНК.
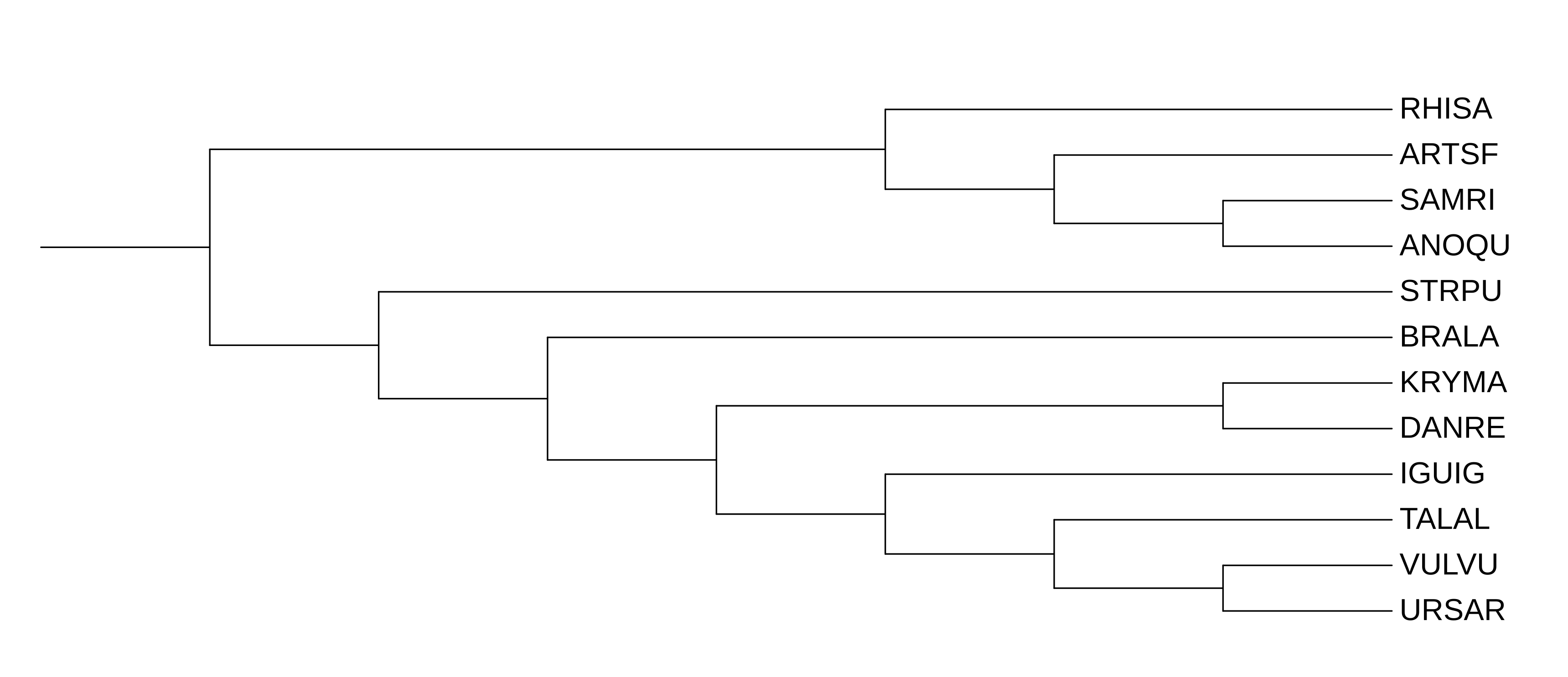
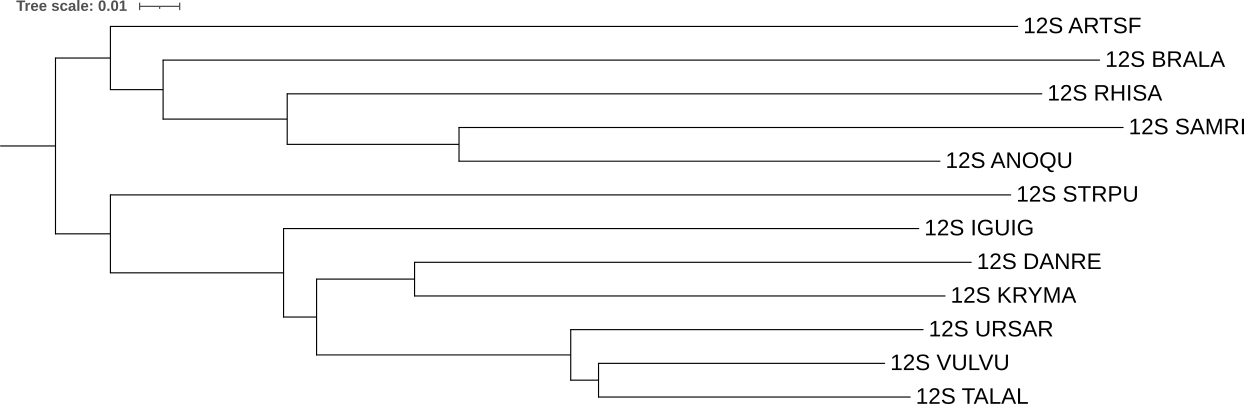
В первую очередь заметим, что ланцентник(BRALA) попал в группу к первичноротым животным. Кроме того, артемия(ARTSF) должна быть ближе к насекомым (SAMRI+ANOQU), чем клещ(RHISA). Также нарушен порядок эволюции позвоночных: сначала от дерева должны были отделится рыбы(DANRE+KRYMA), а только потом рептилии(IGUIG). Наконец, медведь(URSAR) и лисица(VULVU) должны были быть объединены в одну группу, сестренскую к кроту(TALAL), однако на дереве, построенном на основе 12S-рРНК, крот и лисица объединены вместе, а медведь - сестренская к ним группа.
Ниже представлено сравнение дерева, полученного на основе белковой последовательности цитохрома B(Рис. 3), и дерева на основе 12S-рРНК:


В данном случае, на двух деревьев порядок отделение рептилий и рыб совпадает. Кроме того, отличается положение ланцетника(BRALA) и морского ежа(STRPU). Все остальные отличия совпадают с таковыми из предыдущего сравнения. Таким образом, дерево, построенное на основе белковой последовательности цитохрома B, лучше отражает современные представления об эволюции животных, чем дерево, построенное на основе 12S-рРНК.
Укоренение во внешнюю группу
На Рис. 4 представлено филогенетическое дерево, построенное программой Fastme на основе белковой последовательности цитохрома B. В качестве внешней группы был выбран С. elegans(CYB_CAEEL).

Как видно из представленных данных, укоренение полученного дерева в аутгруппу дало корректно укоренненное дерево с точки зрения расположения крупных таксономических клад.
Бутсреп
На Рис. 5 представлено дерево, построенное на основе 12s-рРНК c указанием бутстреп-поддержки.

Наиболее низкую поддержку имеют ветви: (VULVU+TALAL) - 64%, (BRALA+RHISA+SAMRI+ANOQU) - 4% и (STRPU+позвоночные) - 0%. Как отмечалось выше, все три ветви реконструированны неправильно. Стоит также заметить, что клада (RHISA+SAMRI+ANOQU) имеет бутсреп-поддержку 100%, однако реконструированна неправильно. Большиство ветвей с высокой бутстреп-поддержкой всё-же реконстроированны праильно.