Практическая работа 4
Составление списка гомологичных белков, включающих паралоги
Для практической работы я выбрал организмы со следующими мнемониками:
- ACICJ
- AGRFC
- AROAE
- BARHE
- BORPE
- BRUSU
- BURMA
Соответствующией файлы с протеомами были скопированы в отдельную папку и затем объединены в единый файл:
cat * > db.fasta
Затем была создана белковая база данных с помощью программы makeblastdb:
makeblastdb -in db.fasta -dbtype prot
Далее была запущена программа blastp для поиска по созданной белковой базе данных гомологов белка CLPX_ECOLI (ссылка) с порогом E-value в 0.0001 :
blastp -task blastp -query aim.fasta -db db.fasta -out blast.out -evalue 0.0001
Список находок можно посмотреть здесь
Выделение последовательностей всех найденных гомологов в отдельный файл осуществлялось с помощью сценария на Python.
Построение филогенетических деревьев
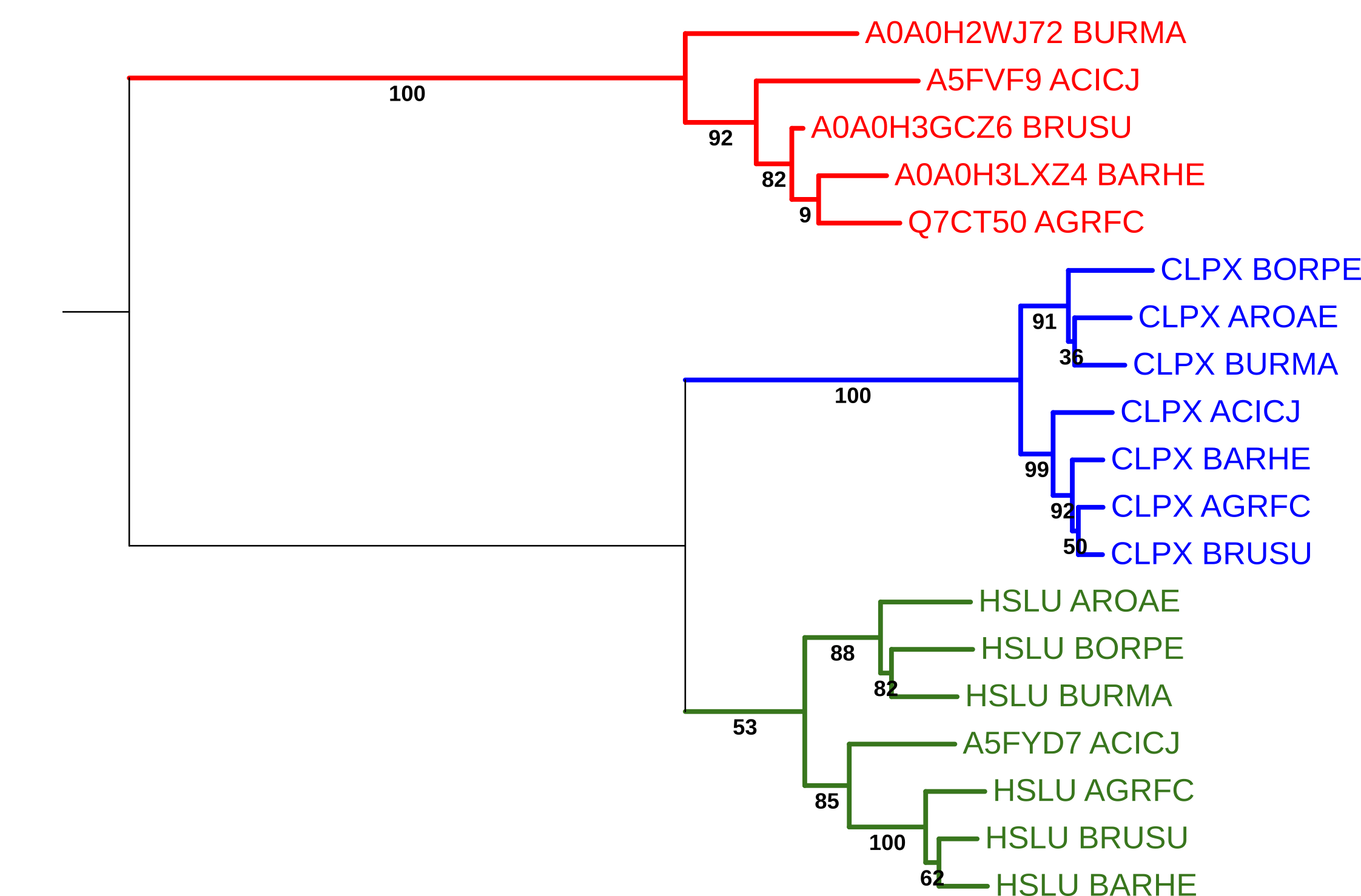
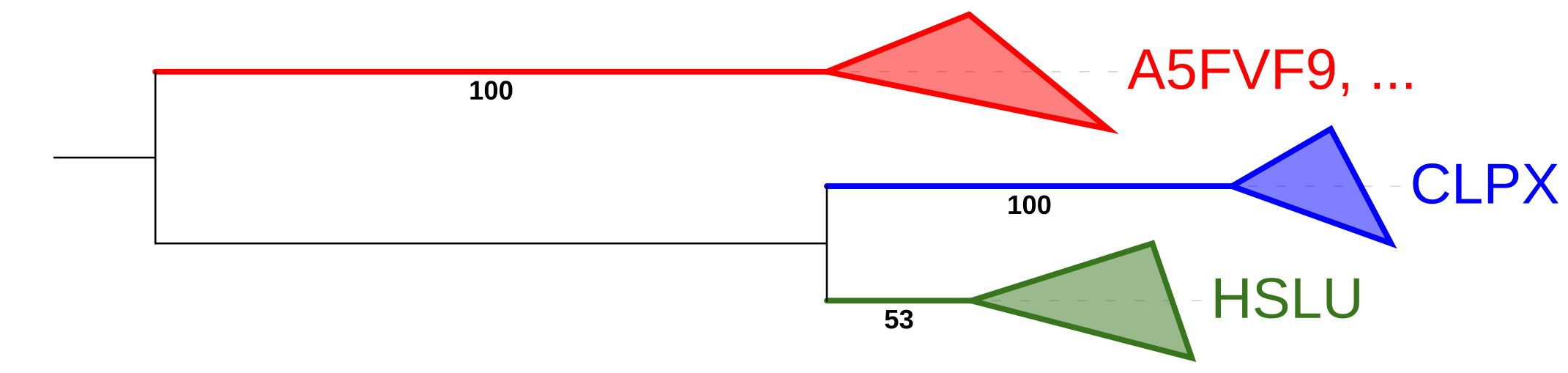
Множественное выравнивание находок осуществлялось с помощью программы Muscle, а построение дерева при помощи FastME с бутстреп-поддержкой со 100 репликами. Формула Newick полученного дерева доступна по ссылке. На Рис. 1 представлено филогенетическое дерево, где различные ортологические группы окрашены в разные цвета. Всего выделилось 3 группы: зелёная - ATP-dependent protease ATPase subunit HslU, синяя - ATP-dependent Clp protease ATP-binding subunit ClpX, красная - ATP-dependent zinc metalloprotease FtsH. Примеры пар ортологов: HSLU_AROAE и HSLU_BORPE, CLPX_AROAE и CLPX_BURMA, Q7CT50_AGRFC и A0A0H3LXZ4_BARHE. Примеры пар паралогов: CLPX_BORPE и HSLU_BORPE, A5FVF9_ACICJ и CLPX_ACICJ, HSLU_BRUSU и A0A0H3GCZ6_BRUSU.
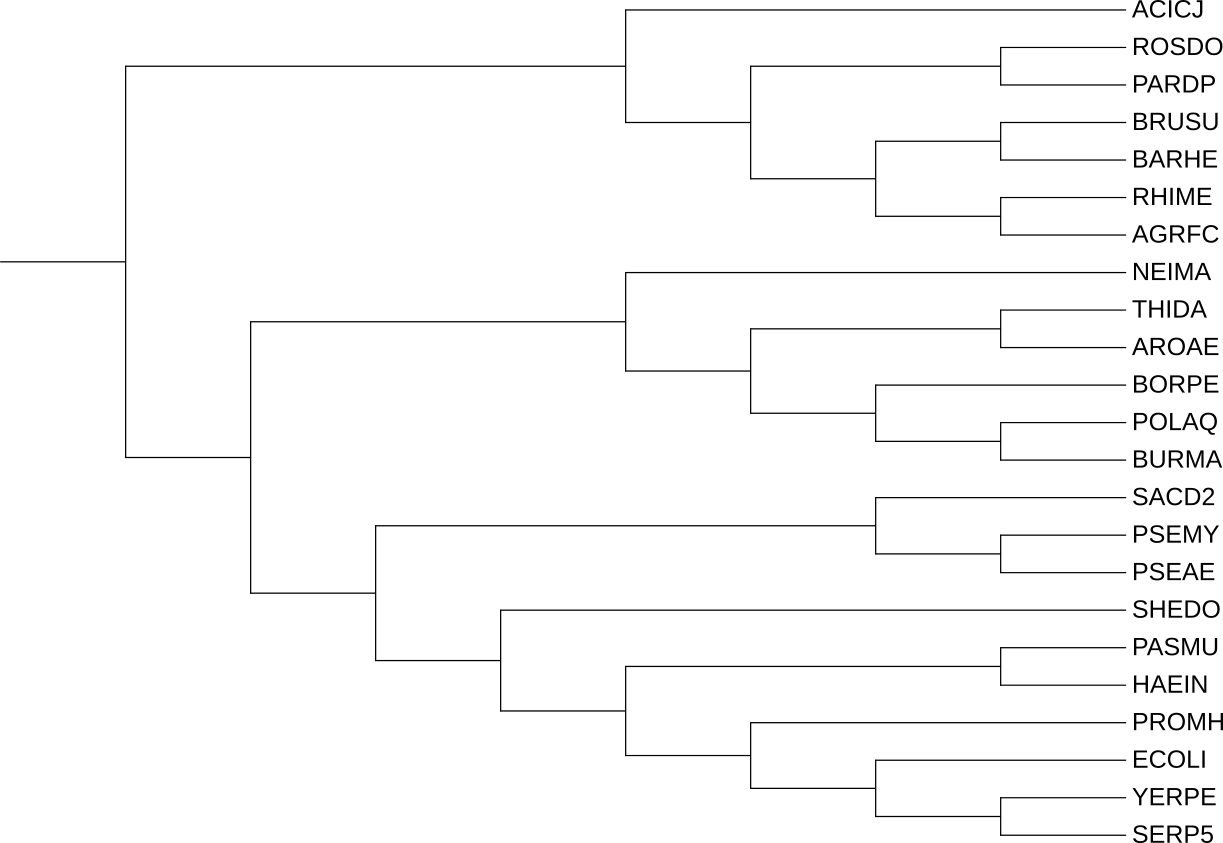
Зеленая группа ортологов полностью соответствует "правильной филогении"(Рис.3) и в нее попали ортологи из всех 7 выбранных организмов. В синюю группу также попали белки всех 7 организмов, однако клада (CLPX_BARHE,(CLPX_AGRFC,CLPX_BRUSU)) реконструирована неверно: вместо нее должна быть клада (CLPX_AGRFC,(CLPX_BARHE,CLPX_BRUSU)). Остальные ветви реконструированы верно. В красную группу ортологов попали белки из всего лишь 5 организмов. Клада (A0A0H3GCZ6_BRUSU,(A0A0H3LXZ4_BARHE,Q7CT50_AGRFC)) реконструирована неверно: вместо нее должна быть клада (Q7CT50_AGRFC,(A0A0H3LXZ4_BARHE,A0A0H3GCZ6_BRUSU)). Остальные ветви реконструированы верно.