Практическая работа 7
Задание 1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
Для выполнения задания я выбрал белок Bacterial Nucleoside Transporter Ts (1tly, TSX_ECOLI) - бактериальный транспортер нуклеоизидов Ts. Этот белок располагается во внешней мембране грам-отрицательной бактерии Escherichia coli и выполняет функцию транспорта нуклеозидов.
В Табл. 1 приведены координаты бета-листов по данным OMP и предсказанных при помощи DeepTMHMM, а на Рис. 1 показана выдача программы DeepTMHMM. Последовательность белка была взята из базы данных Uniprot. Текстовая выдача доступна по ссылке.
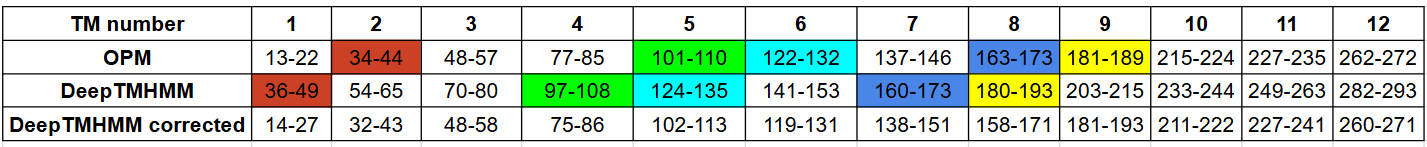
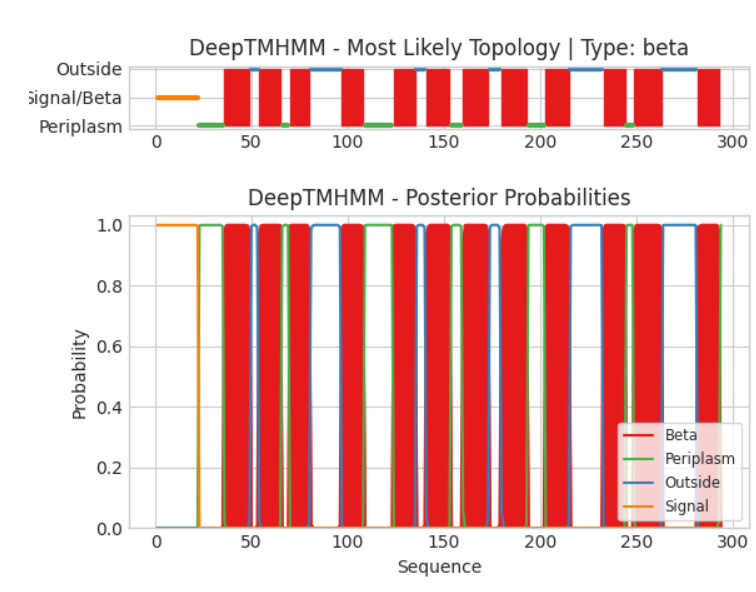
С помощью DeepTMHMM было предсказано 12 трансмембранных бета-листов, из которых только 5 почти полностью совпали с OPM (выделены цветами в Табл. 1). Дело в том, что не была учтена сигнальная последовательность длиной в 22 аминокислоты, расположенная на N-конце поданной DeepTMHMM последовательности из Uniprot. Если пересчитать предсказанный координаты с учетом сигнальной последовательности (см. третью строчку в Табл. 1), то все 12 трансмембранных участков были с хорошей степенью точности предсказаны.
Задание 2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Для анализа был предложен белок Adenosine receptor A2a (2ydv, AA2AR_HUMAN) - рецептор аденозина A2a. Данный белок представляет из себя рецептор к аденозину, сопряженный с G-белком, и располагается на цитоплазматической мембране.
В Табл. 1 приведены координаты альфа-спиралей по данным OMP и предсказанных при помощи DeepTMHMM, а на Рис. 1 показана выдача программы DeepTMHMM. Последовательность белка была взята из базы данных Uniprot. Текстовая выдача доступна по ссылке.
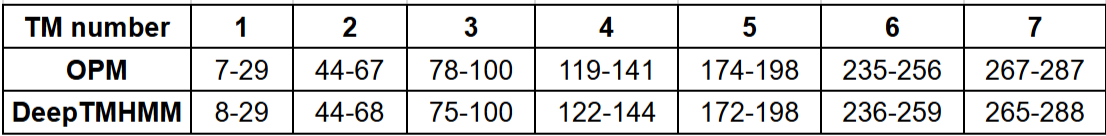
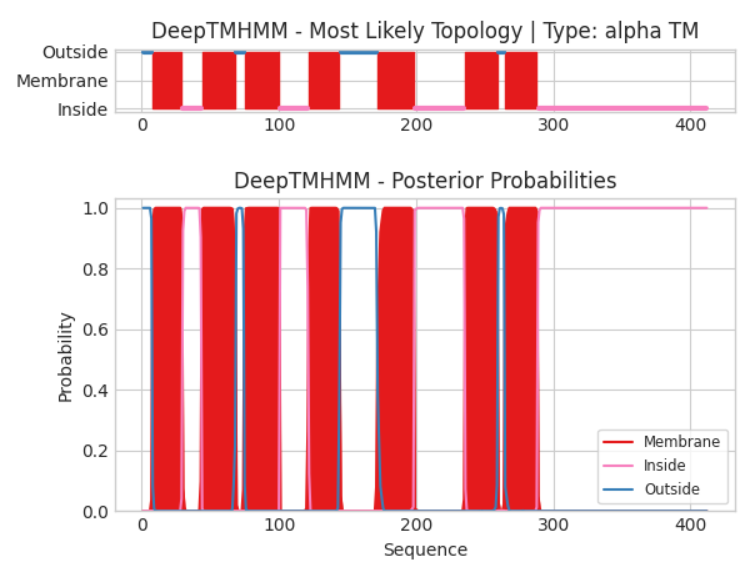
Программа DeepTMHMM предсказала правильно все трансмембранные альфа-спирали белка с точностью до 1-3 аминокислоты.