Поиск по сходству (нуклеотидный BLAST)
Задание 1. Таксономия и функция нуклеотидной последовательности из практикума 6.
Для консенсусной последовательности, полученной в практикуме 6, был произведен поиск в BLASTN ("Somewhat similar sequences") по банку nr.
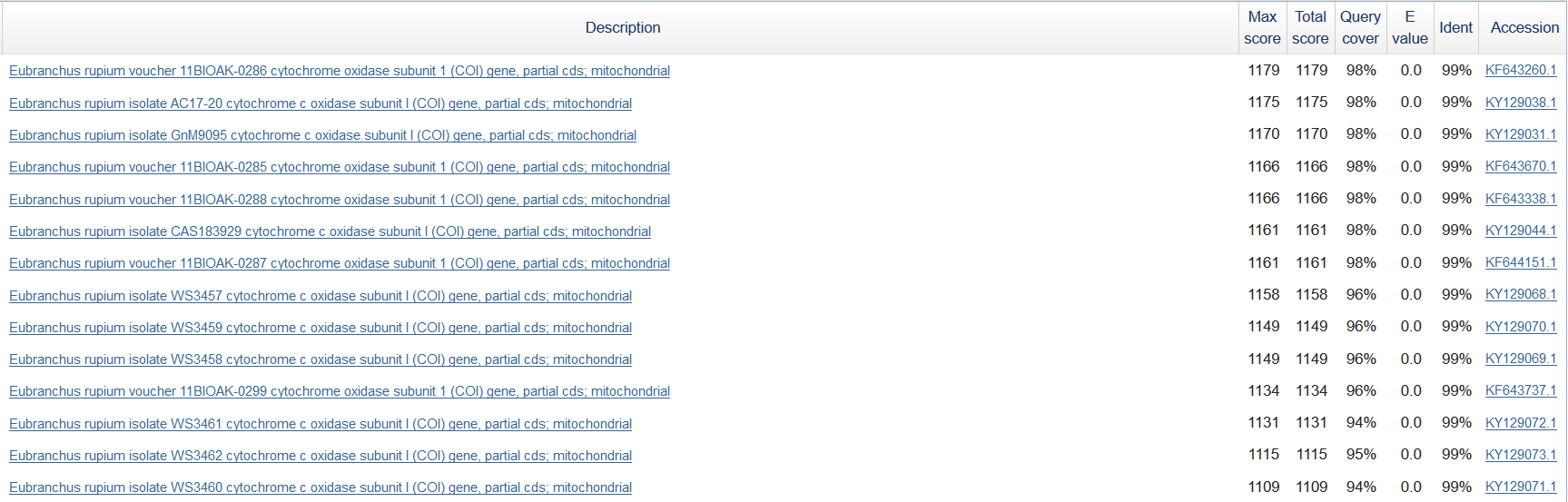
Исходя из полученных данных можно утверждать, что данная последовательность, кодирующая I субъединицу цитохромоксидазы (cytochrome oxidase subunit 1), является частью митохондриального генома брюхоногого моллюска Eubranchus rupium. Полученный выравнивания обладают очень высоким сходством и не содержат гэпов и замен, а также обладают высоким покрытием, что позволяет сделать такой вывод.
Систематическое положение до вида: Eukaryota; Metazoa; Lophotrochozoa; Mollusca; Gastropoda; Heterobranchia; Euthyneura; Nudipleura; Nudibranchia; Aeolididina; Fionoidea; Fionidae; Eubranchus; Eubranchus rupium.
Выравнивание исходной последовательности с
Eubranchus rupium voucher 11BIOAK-0286 cytochrome oxidase subunit 1 (COI)
gene, partial cds; mitochondrial
Sequence ID: KF643260.1
-----------------------------------------------------------------------------
Score Expect Identities Gaps Strand
1179 bits(1307) 0.0 656/658(99%) 0/658(0%) Plus/Plus
-----------------------------------------------------------------------------
Query 9 AACTCTCTATGTTTTATTAGGGATGTGATGTGGTTTAGTGGGAACTGGACTTAGATTGTT 68
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1 AACTCTCTATGTTTTATTAGGGATGTGATGTGGTTTAGTGGGAACTGGACTTAGATTGTT 60
Query 69 AATTCGATTTGAGCTAGGGACTGCCGGAGCTTTGCTTGGAGACGATCATTTGTATAATGT 128
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 61 AATTCGATTTGAGCTAGGGACTGCCGGAGCTTTGCTTGGAGACGATCATTTGTATAATGT 120
Query 129 GATTGTAACTGCTCATGCTTTTGTTATAAttttttttATAGTTATACCTCTTATAATTGG 188
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 121 GATTGTAACTGCTCATGCTTTTGTTATAATTTTTTTTATAGTTATACCTCTTATAATTGG 180
Query 189 GGGTTTTGGGAATTGAATAGTTCCTCTTTTAATTGGTGCTCCTGATATAAGGTTTCCTCG 248
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 181 GGGTTTTGGGAATTGAATAGTTCCTCTTTTAATTGGTGCTCCTGATATAAGGTTTCCTCG 240
Query 249 GATAAATAACATAAGATTCTGGTTGCTTCCTCCTTCTTTTATGCTTTTAATGTCTAGTAC 308
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 241 GATAAATAACATAAGATTCTGGTTGCTTCCTCCTTCTTTTATGCTTTTAATGTCTAGTAC 300
Query 309 ATTAATAGAAGGTGGTGCTGGGACGGGATGGACAGTATACCCTCCTCTCTCTGGTCCTAT 368
|||||||||||||||||||||||||||||||||||||||||||||||| |||||||||||
Sbjct 301 ATTAATAGAAGGTGGTGCTGGGACGGGATGGACAGTATACCCTCCTCTTTCTGGTCCTAT 360
Query 369 AGGCCATGGGGGTTGTTCTGTAGATCTGGCTATTTTTTCTTTACATTTAGCGGGTATGTC 428
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 361 AGGCCATGGGGGTTGTTCTGTAGATCTGGCTATTTTTTCTTTACATTTAGCGGGTATGTC 420
Query 429 TTCTCTTTTAGGTGCTATTAACTTTATTACAACTATTTTTAATATACGGTCTCCTGAGAT 488
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 421 TTCTCTTTTAGGTGCTATTAACTTTATTACAACTATTTTTAATATACGGTCTCCTGAGAT 480
Query 489 AACATGAGACCGATTAAGTTTATTTGTTTGATCGGTGTTAGTTACGGCTTTTTTACTCTT 548
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 481 AACATGAGACCGATTAAGTTTATTTGTTTGATCGGTGTTAGTTACGGCTTTTTTACTCTT 540
Query 549 GTTATCACTTCCTGTGCTAGCTGGGGCTATTACNATGTTACTTACGGATCGTAATTTTAA 608
||||||||||||||||||||||||||||||||| ||||||||||||||||||||||||||
Sbjct 541 GTTATCACTTCCTGTGCTAGCTGGGGCTATTACGATGTTACTTACGGATCGTAATTTTAA 600
Query 609 TACTAGGTTCTTTGATCCTGCAGGTGGAGGAGATCCTATTTTATATCAACATCTGTTT 666
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 601 TACTAGGTTCTTTGATCCTGCAGGTGGAGGAGATCCTATTTTATATCAACATCTGTTT 658
Задание 2. Сравнение списков находок.
Для выполнения данного задания из поиска был исключен весь класс брюхоногих моллюсков, поскольку получался высокий процент идентичности для представителей данного класса. Объем поиска ограничивался 500 последовательностями. Параметры поисков представлены в таблице.
| Параметры поиска | Megablast | BLASTn (default) | BLASTn (custom) |
| Word size | 28 | 11 | 7 |
| Match/Mismatch scores | 1, -2 | 2, -3 | 1, -4 |
| Gapcosts | 0, 2.5 | 5, 2 | 5, 2 |
Все найденные последовательности кодируют I субъединицу цитохромоксидазы. Сравнения результатов поиска представлены в таблице.
| Алгоритм | Top Score | Bottom Score | Top Query Cover | Bottom Query Cover | Top E-Value | Bottom E-Value | Top Ident | Bottom Ident |
| Megablast | 387 | 329 | 86% | 84% | 3e-103 | 6e-86 | 79% | 77% |
| BLASTn (default) | 577 | 471 | 96% | 97% | 3e-160 | 3e-128 | 80% | 76% |
| BLASTn (custom) | 331 | 362 | 94% | 65% | 2e-86 | 5e-36 | 85% | 89% |
В результате поиска были найдены последовательности с высоким сходством. Пр этом с помощью Megablast были были найдены выравнивания со средним процентом покрытия 85% и процентом сходства 78%. BLASTn c параметрами по умолчанию нашёл последовательности с высоким процентом покрытия (большинство более 95%) и процентом сходства между 75% и 80%. BLASTn c чувствительными параметра параметрами нашел последовательности как с выскоим процентом покрытия (85%), так и с низким (вплоть до 25%). Процент идентичности при этом оказался больше 85%. D каждом из 3 поисков список последовательностей в первых 20 результат. Исходя из полученных данных можно сделать вывод о том, что с помощью BLASTn с параметрами по умолчанию можно найти последовательности с выскоим процентом идентичности и высоким покрытием. С помощью более чувствительных параметров поиска можно найти небольшие консервативные участки в последовательности с высокой идентичностью.
Задание 3. Поиск гомолоичных белков.
Для выполнения данного задания была взята неаннотированная сборка генома Amoeboaphelidium protococcarum. С помощью команды
makeblastdb -in X5.fasta -dbtype nuclбыла получена база данных для локального поиска BLAST. С помощью команды seqret были получены последовательности следующих белков из SwissProt: HSP71_YEAST (шаперон HSP70), PRPC_EMENI (митохондриальная цитратсинтаза), TBB_NEUCR (тубулин). Для поиска по базе данных BLAST использовался алгоритм tblastn. Архив с результатами трёх поисков BLAST. Резюме по трём поискам представлено в таблице.
| Белок | Положение в геноме | Вес в битах | E-value | Positives (%) | Заключение |
| HSP71_YEAST | scaffold-199 | 920 | 0.0 | 90% | В выравнивании содержится только 3 гэпа. Есть длинные консервативные участки. Можно утверждать, что белки гомологичны. |
| PRPC_EMENI | scaffold-693 | 393 | 6e-121 | 72% | Выравнивание обладает относительно небольшим весом, однако заметны длинные консервативные участки (до 50 а.к.) в середине выравнивания. Белки могут быть гомологичными. |
| TBB_NEUCR | unplaced-665 | 742 | 0.0 | 88% | Выравнивание почти целиком состоит из одного консервативного участка длиной более 300 остатков а.к. Белки гомологичны. |
Задание 4. Поисков генов в контиге.
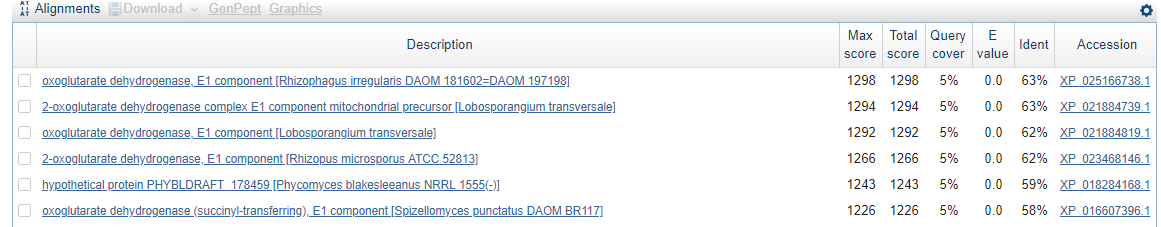
Для выполнения данного задания из генома Amoeboaphelidium protococcarum был взят контиг scaffold-51. По нему был произведен поиск с помощью BLASTX по базе данных NCBI Protein Reference Sequences. В результате были получены выравнивания с нулевым E-value, по которым можно утверждать, что в контиге закодирован белок 2-oxoglutarate dehydrogenase complex E1 component mitochondrial precursor.
Задание 5. Карта геномного сходства.
Для выполнения данного задания были взяты геномы бактерий Burkholderia cepacia (NZ_CP011301.1) и Burkholderia cenocepacia (NZ_CP019668.1). С помощью BLASTN с алгоритмом MEGABLAST была получена карта их локального сходства. Если принять линию, обозначенную синим, за основную, то участки, обозначенные красным можно считать инверсией.
