Филогенетические деревья
Отчет по практикумам 1 и 2
Список протеобактерий
| Бактерия | Мнемоника | Таксономия (с Bacteria; Proteobacteria) |
| Pseudomonas aeruginosa | PSEAE | Gammaproteobacteria; Pseudomonadales; Pseudomonadaceae; Pseudomonas; Pseudomonas aeruginosa group |
| Aromatoleum aromaticum | AROAE | Betaproteobacteria; Rhodocyclales; Rhodocyclaceae; Aromatoleum |
| Paracoccus denitrificans | PARDP | Alphaproteobacteria; Rhodobacterales; Rhodobacteraceae; Paracoccus |
| Proteus mirabilis | PROMH | Gammaproteobacteria; Enterobacterales; Morganellaceae; Proteus |
| Pasteurella multocida | PASMU | Gammaproteobacteria; Pasteurellales; Pasteurellaceae; Pasteurella |
| Roseobacter denitrificans | ROSDO | Alphaproteobacteria; Rhodobacterales; Rhodobacteraceae; Roseobacter |
| Pseudomonas mendocina | PSEMY | Gammaproteobacteria; Pseudomonadales; Pseudomonadaceae; Pseudomonas; Pseudomonas aeruginosa group |
| Neisseria meningitidis | NEIMA | Betaproteobacteria; Neisseriales; Neisseriaceae; Neisseria |
Скобочная формула дерева
((((PROMH, PASMU), (PSEAE, PSEMY)), (NEIMA, AROAE)), (PARDP, ROSDO));
Изображение дерева
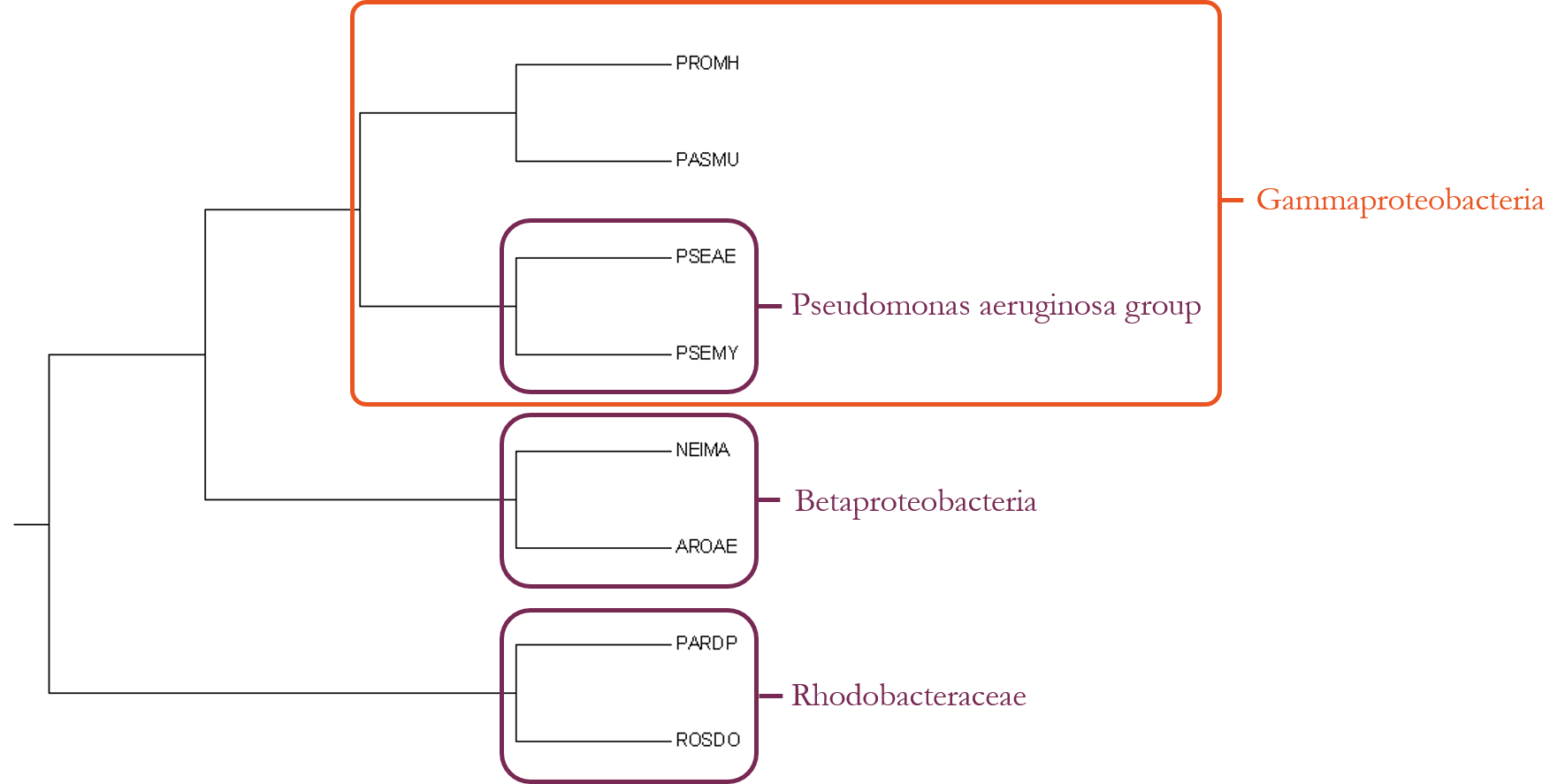
Ветви дерева
Дерево содержит 5 нетривиальных ветвей:
- {PROMH, PASMU} vs {PSEAE, PSEMY, NEIMA, AROAE, PARDP, ROSDO}
- {PROMH, PASMU, PSEAE, PSEMY} vs {NEIMA, AROAE, PARDP, ROSDO}
- {PROMH, PASMU, PSEAE, PSEMY, NEIMA, AROAE} vs {PARDP, ROSDO}
- {PSEAE, PSEMY} vs {PROMH, PASMU, NEIMA, AROAE, PARDP, ROSDO}
- {NEIMA, AROAE} vs {PROMH, PASMU, PSEAE, PSEMY, PARDP, ROSDO}
Ортологи и паралоги
Ход работы (практикум 3)
Из директории /P/y17/term4/Proteomes были взяты протеомы бактериий, указанных в начале отчета. Файлы были объединены в один командой:
cat * >> all.fa
Затем из полученного файла была создана база данных для локального поиска blast гомологов белка CLPX_ECOLI. Команды:
makeblastdb -in all.fa -dbtype prot blastp -query CLPX_ECOLI.fa -db all.fa -out res.out -evalue 0.001
Из файла res.out были взяты мнемоники белков, по которым в JalView с помощью Fetch Sequences были получены последовательности белков, которые были выровнены программой Muscle с параметрами по умолчанию. Полученное множественное выравнивание alignment.fa было открыто в программе Mega 7 для построения филогенетического дерева программой Maximum likelihood.
Результаты
Скобочная формула дерева
Изображение дерева
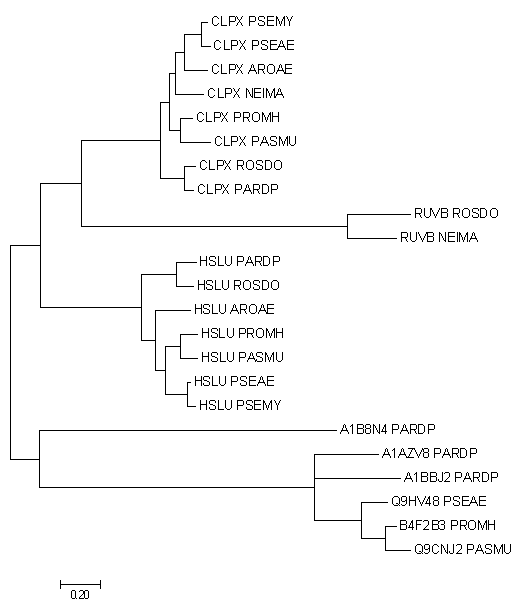
Список белков
| Мнемоника | Название (функция) |
| CLPX | ATP-dependent Clp protease ATP-binding subunit |
| HSLU | ATP-dependent protease ATPase subunit HslU |
| RUVB | Holliday junction ATP-dependent DNA helicase |
| A1B8N4 | ATP-dependent Clp protease, ATP-binding |
| A1AZV8 | ATP-dependent zinc metalloprotease FtsH |
| A1BBJ2 | ATP-dependent zinc metalloprotease FtsH |
| B4F2B3 | ATP-dependent zinc metalloprotease FtsH |
| Q9HV48 | ATP-dependent zinc metalloprotease FtsH |
| Q9CNJ2 | ATP-dependent zinc metalloprotease FtsH |
Обсуждение
Примерами ортологов являются белки с мнемониками CLPX, RUVB, HSLU, поскольку они гомологичны и представлены в разных организмах. Их пути эволюции разделилилсь в результате видообразования. Белки с мнемониками A1B8N4, A1AZV8 и A1BBJ2 из бактерии с мнемоникой PARDP - паралоги. Они произошли в результате дупликации гена.